+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4nxp | ||||||
|---|---|---|---|---|---|---|---|
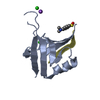
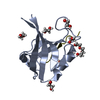
| タイトル | Crystal Structure of Free T-cell Lymphoma Invasion and Metastasis-1 PDZ Domain Quadruple Mutant (QM) | ||||||
 要素 要素 | T-lymphoma invasion and metastasis-inducing protein 1 | ||||||
 キーワード キーワード |  SIGNALING PROTEIN / Beta barrel fold protein / SIGNALING PROTEIN / Beta barrel fold protein /  PDZ domain (PDZドメイン) / PDZ domain (PDZドメイン) /  peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction / peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction /  signaling domain signaling domain | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation / regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity / regulation of small GTPase mediated signal transduction / regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity / regulation of small GTPase mediated signal transduction /  Wnt signaling pathway, planar cell polarity pathway / main axon / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / RAC2 GTPase cycle / ephrin receptor signaling pathway / RAC3 GTPase cycle / axonal growth cone / positive regulation of epithelial to mesenchymal transition / RAC1 GTPase cycle / EPHB-mediated forward signaling / cell-matrix adhesion / guanyl-nucleotide exchange factor activity / regulation of ERK1 and ERK2 cascade / response to cocaine / Wnt signaling pathway, planar cell polarity pathway / main axon / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / RAC2 GTPase cycle / ephrin receptor signaling pathway / RAC3 GTPase cycle / axonal growth cone / positive regulation of epithelial to mesenchymal transition / RAC1 GTPase cycle / EPHB-mediated forward signaling / cell-matrix adhesion / guanyl-nucleotide exchange factor activity / regulation of ERK1 and ERK2 cascade / response to cocaine /  receptor tyrosine kinase binding / receptor tyrosine kinase binding /  kinase binding / ruffle membrane / G alpha (12/13) signalling events / cell-cell junction / kinase binding / ruffle membrane / G alpha (12/13) signalling events / cell-cell junction /  遊走 / positive regulation of protein binding / 遊走 / positive regulation of protein binding /  microtubule binding / protein-containing complex assembly / microtubule binding / protein-containing complex assembly /  微小管 / 微小管 /  樹状突起スパイン / positive regulation of cell migration / neuronal cell body / glutamatergic synapse / 樹状突起スパイン / positive regulation of cell migration / neuronal cell body / glutamatergic synapse /  lipid binding / lipid binding /  シナプス / positive regulation of cell population proliferation / シナプス / positive regulation of cell population proliferation /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å | ||||||
 データ登録者 データ登録者 | Liu, X. / Speckhard, D.C. / Shepherd, T.R. / Hengel, S.R. / Fuentes, E.J. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2016 ジャーナル: Structure / 年: 2016タイトル: Distinct Roles for Conformational Dynamics in Protein-Ligand Interactions. 著者: Liu, X. / Speckhard, D.C. / Shepherd, T.R. / Sun, Y.J. / Hengel, S.R. / Yu, L. / Fowler, C.A. / Gakhar, L. / Fuentes, E.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4nxp.cif.gz 4nxp.cif.gz | 30.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4nxp.ent.gz pdb4nxp.ent.gz | 19.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4nxp.json.gz 4nxp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nx/4nxp https://data.pdbj.org/pub/pdb/validation_reports/nx/4nxp ftp://data.pdbj.org/pub/pdb/validation_reports/nx/4nxp ftp://data.pdbj.org/pub/pdb/validation_reports/nx/4nxp | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 10192.402 Da / 分子数: 1 / 断片: PDZ domain / 変異: L911M, K912E, L915F, L920V / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 株: human Homo sapiens (ヒト) / 株: human ヒト / 遺伝子: TIAM1 / プラスミド: PET21A-6HIS-rTEV / 発現宿主: ヒト / 遺伝子: TIAM1 / プラスミド: PET21A-6HIS-rTEV / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q13009 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q13009 |
|---|---|
| #2: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.12 Å3/Da / 溶媒含有率: 42.08 % |
|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.8 詳細: 1.5M Ammonium Sulfate, 0.1M Tris, pH=8.5, 12% Glycerol, pH 6.8, VAPOR DIFFUSION, HANGING DROP, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418 Å | |||||||||||||||
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE / 日付: 2011年7月21日 | |||||||||||||||
| 放射 | モノクロメーター: GRAPHITE / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 | |||||||||||||||
| Reflection twin |
| |||||||||||||||
| 反射 | 解像度: 2.3→19.85 Å / Num. all: 4117 / Num. obs: 3866 / % possible obs: 93.9 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 3.18 % / Biso Wilson estimate: 34.3 Å2 / Rmerge(I) obs: 0.104 / Net I/σ(I): 7.7 | |||||||||||||||
| 反射 シェル | 解像度: 2.3→2.38 Å / 冗長度: 3.17 % / Rmerge(I) obs: 0.421 / Mean I/σ(I) obs: 2.1 / % possible all: 97.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB entry 3KZD 解像度: 2.3→19.85 Å / Cor.coef. Fo:Fc: 0.933 / Cor.coef. Fo:Fc free: 0.903 / SU B: 7.454 / SU ML: 0.166 / 交差検証法: THROUGHOUT / ESU R: 0.102 / ESU R Free: 0.058 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 31.409 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.3→19.85 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj








