+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4mqs | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of active human M2 muscarinic acetylcholine receptor bound to the agonist iperoxo | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  G protein-coupled receptor (Gタンパク質共役受容体) / G protein-coupled receptor (Gタンパク質共役受容体) /  Muscarinic acetylcholine receptor Muscarinic acetylcholine receptor | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 Muscarinic acetylcholine receptors / symmetric synapse / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled acetylcholine receptor activity / regulation of smooth muscle contraction / cholinergic synapse / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled serotonin receptor activity / arrestin family protein binding / Muscarinic acetylcholine receptors / symmetric synapse / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled acetylcholine receptor activity / regulation of smooth muscle contraction / cholinergic synapse / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled serotonin receptor activity / arrestin family protein binding /  regulation of heart contraction ... regulation of heart contraction ... Muscarinic acetylcholine receptors / symmetric synapse / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled acetylcholine receptor activity / regulation of smooth muscle contraction / cholinergic synapse / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled serotonin receptor activity / arrestin family protein binding / Muscarinic acetylcholine receptors / symmetric synapse / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled acetylcholine receptor activity / regulation of smooth muscle contraction / cholinergic synapse / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / G protein-coupled serotonin receptor activity / arrestin family protein binding /  regulation of heart contraction / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / asymmetric synapse / axon terminus / presynaptic modulation of chemical synaptic transmission / clathrin-coated endocytic vesicle membrane / response to virus / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / G protein-coupled acetylcholine receptor signaling pathway / Cargo recognition for clathrin-mediated endocytosis / regulation of heart contraction / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / asymmetric synapse / axon terminus / presynaptic modulation of chemical synaptic transmission / clathrin-coated endocytic vesicle membrane / response to virus / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / G protein-coupled acetylcholine receptor signaling pathway / Cargo recognition for clathrin-mediated endocytosis /  Clathrin-mediated endocytosis / Clathrin-mediated endocytosis /  presynaptic membrane / presynaptic membrane /  nervous system development / G alpha (i) signalling events / chemical synaptic transmission / nervous system development / G alpha (i) signalling events / chemical synaptic transmission /  postsynaptic membrane / G protein-coupled receptor signaling pathway / neuronal cell body / glutamatergic synapse / postsynaptic membrane / G protein-coupled receptor signaling pathway / neuronal cell body / glutamatergic synapse /  シナプス / シナプス /  樹状突起 / 樹状突起 /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Lama glama (ラマ) Lama glama (ラマ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.5 Å 分子置換 / 解像度: 3.5 Å | ||||||
 データ登録者 データ登録者 | Kruse, A.C. / Ring, A.M. / Manglik, A. / Hu, J. / Hu, K. / Eitel, K. / Huebner, H. / Pardon, E. / Valant, C. / Sexton, P.M. ...Kruse, A.C. / Ring, A.M. / Manglik, A. / Hu, J. / Hu, K. / Eitel, K. / Huebner, H. / Pardon, E. / Valant, C. / Sexton, P.M. / Christopoulos, A. / Felder, C.C. / Gmeiner, P. / Steyaert, J. / Weis, W.I. / Garcia, K.C. / Wess, J. / Kobilka, B.K. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2013 ジャーナル: Nature / 年: 2013タイトル: Activation and allosteric modulation of a muscarinic acetylcholine receptor. 著者: Kruse, A.C. / Ring, A.M. / Manglik, A. / Hu, J. / Hu, K. / Eitel, K. / Hubner, H. / Pardon, E. / Valant, C. / Sexton, P.M. / Christopoulos, A. / Felder, C.C. / Gmeiner, P. / Steyaert, J. / ...著者: Kruse, A.C. / Ring, A.M. / Manglik, A. / Hu, J. / Hu, K. / Eitel, K. / Hubner, H. / Pardon, E. / Valant, C. / Sexton, P.M. / Christopoulos, A. / Felder, C.C. / Gmeiner, P. / Steyaert, J. / Weis, W.I. / Garcia, K.C. / Wess, J. / Kobilka, B.K. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR DETERMINED |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4mqs.cif.gz 4mqs.cif.gz | 93.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4mqs.ent.gz pdb4mqs.ent.gz | 67.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4mqs.json.gz 4mqs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mq/4mqs https://data.pdbj.org/pub/pdb/validation_reports/mq/4mqs ftp://data.pdbj.org/pub/pdb/validation_reports/mq/4mqs ftp://data.pdbj.org/pub/pdb/validation_reports/mq/4mqs | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  分子量: 39951.734 Da / 分子数: 1 / 断片: UNP residues 1-232, 373-466 / 変異: N0D, N1D, N4D, N7D, A373T, K374R / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: CHRM2 / プラスミド: PVL1392 Homo sapiens (ヒト) / 遺伝子: CHRM2 / プラスミド: PVL1392発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P08172 |
|---|---|
| #2: 抗体 | 分子量: 13428.396 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Lama glama (ラマ) / プラスミド: pMAL3CBN / 発現宿主: Lama glama (ラマ) / プラスミド: pMAL3CBN / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) |
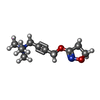
| #3: 化合物 | ChemComp-IXO / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.76 Å3/Da / 溶媒含有率: 67.31 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 詳細: Reconstituted in 10:1 monoolein:cholesterol mix. Precipitant solution: 10 - 20% PEG300, 100 mM HEPES, pH 7.2 - 7.9, 1.2% 1,2,3-heptanetriol, and 20 - 80 mM EDTA, pH 8.0, Lipidic cubic phase, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 77 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 23-ID-D / 波長: 1.033 Å / ビームライン: 23-ID-D / 波長: 1.033 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2013年2月23日 / 詳細: mirrors |
| 放射 | モノクロメーター: Double crystal cryo-cooled Si(111) プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.033 Å / 相対比: 1 : 1.033 Å / 相対比: 1 |
| 反射 | 解像度: 3.5→33 Å / Num. all: 10386 / Num. obs: 10386 / % possible obs: 95.9 % / Observed criterion σ(I): -3 / 冗長度: 4.8 % / Rmerge(I) obs: 0.188 |
| 反射 シェル | 解像度: 3.5→3.6 Å / 冗長度: 2.5 % / Rmerge(I) obs: 0.744 / Num. unique all: 871 / % possible all: 83.1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB entry 3UON 解像度: 3.5→32.522 Å / SU ML: 0.57 / 交差検証法: THROUGHOUT / σ(F): 1.38 / 位相誤差: 35.28 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.5→32.522 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj