登録情報 データベース : PDB / ID : 4eagタイトル Co-crystal structure of an chimeric AMPK core with ATP (5'-AMP-activated protein kinase subunit ...) x 2 EG:132E8.2 protein キーワード / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Drosophila melanogaster (キイロショウジョウバエ)Rattus norvegicus (ドブネズミ)手法 / / / 解像度 : 2.701 Å データ登録者 Chen, L. / Wang, J. / Zhang, Y.-Y. / Yan, S.F. / Neumann, D. / Schlattner, U. / Wang, Z.-X. / Wu, J.-W. ジャーナル : Nat.Struct.Mol.Biol. / 年 : 2012タイトル : AMP-activated protein kinase undergoes nucleotide-dependent conformational changes著者 : Chen, L. / Wang, J. / Zhang, Y.-Y. / Yan, S.F. / Neumann, D. / Schlattner, U. / Wang, Z.-X. / Wu, J.-W. 履歴 登録 2012年3月22日 登録サイト / 処理サイト 改定 1.0 2012年6月6日 Provider / タイプ 改定 1.1 2014年3月12日 Group 改定 1.2 2023年11月8日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) / AMPK
TRANSFERASE (転移酵素) / AMPK 機能・相同性情報
機能・相同性情報 regulation of digestive system process / AMPK inhibits chREBP transcriptional activation activity / Energy dependent regulation of mTOR by LKB1-AMPK / Carnitine metabolism / Regulation of TP53 Activity through Phosphorylation / TP53 Regulates Metabolic Genes /
regulation of digestive system process / AMPK inhibits chREBP transcriptional activation activity / Energy dependent regulation of mTOR by LKB1-AMPK / Carnitine metabolism / Regulation of TP53 Activity through Phosphorylation / TP53 Regulates Metabolic Genes /  Gタンパク質共役受容体キナーゼ / behavioral response to starvation /
Gタンパク質共役受容体キナーゼ / behavioral response to starvation /  オートファジー / Energy dependent regulation of mTOR by LKB1-AMPK ...
オートファジー / Energy dependent regulation of mTOR by LKB1-AMPK ... regulation of digestive system process / AMPK inhibits chREBP transcriptional activation activity / Energy dependent regulation of mTOR by LKB1-AMPK / Carnitine metabolism / Regulation of TP53 Activity through Phosphorylation / TP53 Regulates Metabolic Genes /
regulation of digestive system process / AMPK inhibits chREBP transcriptional activation activity / Energy dependent regulation of mTOR by LKB1-AMPK / Carnitine metabolism / Regulation of TP53 Activity through Phosphorylation / TP53 Regulates Metabolic Genes /  Gタンパク質共役受容体キナーゼ / behavioral response to starvation /
Gタンパク質共役受容体キナーゼ / behavioral response to starvation /  オートファジー / Energy dependent regulation of mTOR by LKB1-AMPK / basolateral part of cell / Regulation of TP53 Activity through Phosphorylation /
オートファジー / Energy dependent regulation of mTOR by LKB1-AMPK / basolateral part of cell / Regulation of TP53 Activity through Phosphorylation /  オートファジー / TP53 Regulates Metabolic Genes / nail development / G protein-coupled receptor kinase activity /
オートファジー / TP53 Regulates Metabolic Genes / nail development / G protein-coupled receptor kinase activity /  転移酵素; リンを含む基を移すもの; タンパク質-セリン/スレオニンキナーゼ / sequestering of triglyceride / import into nucleus / protein kinase regulator activity / negative regulation of cell size / establishment or maintenance of epithelial cell apical/basal polarity / nucleotide-activated protein kinase complex / positive regulation of axon guidance / dendrite morphogenesis / positive regulation of lipophagy / AMP-activated protein kinase activity / AMP binding / cellular response to nutrient levels / negative regulation of TORC1 signaling / cellular response to starvation /
転移酵素; リンを含む基を移すもの; タンパク質-セリン/スレオニンキナーゼ / sequestering of triglyceride / import into nucleus / protein kinase regulator activity / negative regulation of cell size / establishment or maintenance of epithelial cell apical/basal polarity / nucleotide-activated protein kinase complex / positive regulation of axon guidance / dendrite morphogenesis / positive regulation of lipophagy / AMP-activated protein kinase activity / AMP binding / cellular response to nutrient levels / negative regulation of TORC1 signaling / cellular response to starvation /  regulation of cell growth /
regulation of cell growth /  ADP binding / lipid metabolic process / fatty acid biosynthetic process / mitotic cell cycle / positive regulation of cold-induced thermogenesis /
ADP binding / lipid metabolic process / fatty acid biosynthetic process / mitotic cell cycle / positive regulation of cold-induced thermogenesis /  エンドソーム /
エンドソーム /  protein kinase activity / intracellular signal transduction /
protein kinase activity / intracellular signal transduction /  protein phosphorylation / protein serine/threonine kinase activity / protein-containing complex binding / positive regulation of gene expression /
protein phosphorylation / protein serine/threonine kinase activity / protein-containing complex binding / positive regulation of gene expression /  protein kinase binding /
protein kinase binding /  シグナル伝達 / protein-containing complex /
シグナル伝達 / protein-containing complex /  ATP binding /
ATP binding /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Drosophila melanogaster (キイロショウジョウバエ)
Drosophila melanogaster (キイロショウジョウバエ)
 Rattus norvegicus (ドブネズミ)
Rattus norvegicus (ドブネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.701 Å
分子置換 / 解像度: 2.701 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2012
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4eag.cif.gz
4eag.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4eag.ent.gz
pdb4eag.ent.gz PDB形式
PDB形式 4eag.json.gz
4eag.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ea/4eag
https://data.pdbj.org/pub/pdb/validation_reports/ea/4eag ftp://data.pdbj.org/pub/pdb/validation_reports/ea/4eag
ftp://data.pdbj.org/pub/pdb/validation_reports/ea/4eag リンク
リンク 集合体
集合体
 要素
要素
 Drosophila melanogaster (キイロショウジョウバエ)
Drosophila melanogaster (キイロショウジョウバエ)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) 転移酵素; リンを含む基を移すもの; タンパク質-セリン/スレオニンキナーゼ,
転移酵素; リンを含む基を移すもの; タンパク質-セリン/スレオニンキナーゼ,  Gタンパク質共役受容体キナーゼ
Gタンパク質共役受容体キナーゼ
 Rattus norvegicus (ドブネズミ) / 遺伝子: Prkab1 / 発現宿主:
Rattus norvegicus (ドブネズミ) / 遺伝子: Prkab1 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P80386
Escherichia coli (大腸菌) / 参照: UniProt: P80386
 Rattus norvegicus (ドブネズミ) / 遺伝子: Prkag1 / 発現宿主:
Rattus norvegicus (ドブネズミ) / 遺伝子: Prkag1 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P80385
Escherichia coli (大腸菌) / 参照: UniProt: P80385
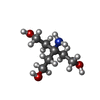



 アデノシン三リン酸
アデノシン三リン酸 トリスヒドロキシメチルアミノメタン
トリスヒドロキシメチルアミノメタン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 0.99583 Å
/ ビームライン: BL17U / 波長: 0.99583 Å : 0.99583 Å / 相対比: 1
: 0.99583 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj


