+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4dkl | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the mu-opioid receptor bound to a morphinan antagonist | ||||||
 要素 要素 | Mu-type opioid receptor, lysozyme chimera | ||||||
 キーワード キーワード |  SIGNALING PROTEIN/ANTAGONIST / SIGNALING PROTEIN/ANTAGONIST /  G-protein coupled receptor (Gタンパク質共役受容体) / G-protein coupled receptor (Gタンパク質共役受容体) /  7 transmembrane receptor (Gタンパク質共役受容体) / 7 transmembrane receptor (Gタンパク質共役受容体) /  SIGNALING PROTEIN-ANTAGONIST complex SIGNALING PROTEIN-ANTAGONIST complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Opioid Signalling /  spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity ...Opioid Signalling / spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity ...Opioid Signalling /  spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity / spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity /  filamin binding / G protein-coupled opioid receptor signaling pathway / regulation of cellular response to stress / G alpha (i) signalling events / behavioral response to ethanol / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / negative regulation of nitric oxide biosynthetic process / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / filamin binding / G protein-coupled opioid receptor signaling pathway / regulation of cellular response to stress / G alpha (i) signalling events / behavioral response to ethanol / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / negative regulation of nitric oxide biosynthetic process / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway /  neuropeptide binding / negative regulation of cAMP-mediated signaling / neuropeptide binding / negative regulation of cAMP-mediated signaling /  regulation of NMDA receptor activity / positive regulation of neurogenesis / regulation of NMDA receptor activity / positive regulation of neurogenesis /  eating behavior / negative regulation of cytosolic calcium ion concentration / transmission of nerve impulse / G-protein alpha-subunit binding / neuropeptide signaling pathway / GABA-ergic synapse / eating behavior / negative regulation of cytosolic calcium ion concentration / transmission of nerve impulse / G-protein alpha-subunit binding / neuropeptide signaling pathway / GABA-ergic synapse /  voltage-gated calcium channel activity / positive regulation of cAMP-mediated signaling / viral release from host cell by cytolysis / sensory perception of pain / dendrite membrane / presynaptic modulation of chemical synaptic transmission / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / voltage-gated calcium channel activity / positive regulation of cAMP-mediated signaling / viral release from host cell by cytolysis / sensory perception of pain / dendrite membrane / presynaptic modulation of chemical synaptic transmission / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway /  excitatory postsynaptic potential / dendrite cytoplasm / peptidoglycan catabolic process / locomotory behavior / G protein-coupled receptor activity / adenylate cyclase-activating dopamine receptor signaling pathway / G-protein beta-subunit binding / positive regulation of nitric oxide biosynthetic process / cell wall macromolecule catabolic process / excitatory postsynaptic potential / dendrite cytoplasm / peptidoglycan catabolic process / locomotory behavior / G protein-coupled receptor activity / adenylate cyclase-activating dopamine receptor signaling pathway / G-protein beta-subunit binding / positive regulation of nitric oxide biosynthetic process / cell wall macromolecule catabolic process /  リゾチーム / リゾチーム /  presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway /  lysozyme activity / positive regulation of cytosolic calcium ion concentration / lysozyme activity / positive regulation of cytosolic calcium ion concentration /  postsynaptic membrane / postsynaptic membrane /  perikaryon / response to ethanol / host cell cytoplasm / positive regulation of ERK1 and ERK2 cascade / perikaryon / response to ethanol / host cell cytoplasm / positive regulation of ERK1 and ERK2 cascade /  エンドソーム / defense response to bacterium / neuron projection / G protein-coupled receptor signaling pathway / protein domain specific binding / エンドソーム / defense response to bacterium / neuron projection / G protein-coupled receptor signaling pathway / protein domain specific binding /  神経繊維 / 神経繊維 /  focal adhesion / focal adhesion /  樹状突起 / 樹状突起 /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Manglik, A. / Kruse, A.C. / Kobilka, T.S. / Thian, F.S. / Mathiesen, J.M. / Sunahara, R.K. / Pardo, L. / Weis, W.I. / Kobilka, B.K. / Granier, S. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2012 ジャーナル: Nature / 年: 2012タイトル: Crystal structure of the {mu}-opioid receptor bound to a morphinan antagonist. 著者: Manglik, A. / Kruse, A.C. / Kobilka, T.S. / Thian, F.S. / Mathiesen, J.M. / Sunahara, R.K. / Pardo, L. / Weis, W.I. / Kobilka, B.K. / Granier, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4dkl.cif.gz 4dkl.cif.gz | 201.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4dkl.ent.gz pdb4dkl.ent.gz | 158.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4dkl.json.gz 4dkl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dk/4dkl https://data.pdbj.org/pub/pdb/validation_reports/dk/4dkl ftp://data.pdbj.org/pub/pdb/validation_reports/dk/4dkl ftp://data.pdbj.org/pub/pdb/validation_reports/dk/4dkl | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  3oduS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 52778.922 Da / 分子数: 1 / 断片: SEE REMARK 999 / 変異: D1020N, C1054T, C1097A / 由来タイプ: 組換発現 由来: (組換発現)   Mus musculus (ハツカネズミ), (組換発現) Mus musculus (ハツカネズミ), (組換発現)   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ)遺伝子: Mor, Oprm, Oprm1, E / プラスミド: pFastBac 発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)株 (発現宿主): Sf9 / 参照: UniProt: P42866, UniProt: P00720,  リゾチーム リゾチーム |
|---|
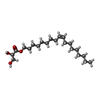
-非ポリマー , 7種, 55分子 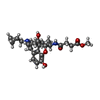











| #2: 化合物 | ChemComp-BF0 /  Beta-Funaltrexamine Beta-Funaltrexamine | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| #3: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#4: 化合物 | ChemComp-CLR / |  コレステロール コレステロール#5: 化合物 | #6: 化合物 | ChemComp-1PE / |  ポリエチレングリコール ポリエチレングリコール#7: 化合物 | ChemComp-CL / |  塩化物 塩化物#8: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 配列の詳細 | CHAIN A IS AN INTERNAL FUSION OF LYSOZYME (RESIDUES 2-161 OF UNP P00720) BETWEEN RESIDUES 52-263 ...CHAIN A IS AN INTERNAL FUSION OF LYSOZYME (RESIDUES 2-161 OF UNP P00720) BETWEEN RESIDUES 52-263 AND RESIDUES 270-352 OF MU-TYPE OPIOID RECEPTOR (UNP P42866). AN OFFSET OF 1000 HAS BEEN ADDED TO LYSOZYME RESIDUE NUMBERS WITHIN THE COORDINATE |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 25 X線回折 / 使用した結晶の数: 25 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.82 Å3/Da / 溶媒含有率: 67.78 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 / pH: 7 詳細: 100 mM HEPES, pH 7.0, 300 mM lithium sulfate, 7.5% DMSO, 30-38% PEG400 in monoolein:cholesterol mixed in a 10:1 ratio, LIPIDIC CUBIC PHASE, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 78 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 23-ID-D / 波長: 1.033 Å / ビームライン: 23-ID-D / 波長: 1.033 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2011年11月30日 / 詳細: Mirrors |
| 放射 | モノクロメーター: Double crystal cryo-cooled Si(111) プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.033 Å / 相対比: 1 : 1.033 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→30.5 Å / Num. obs: 19145 / % possible obs: 99 % / Observed criterion σ(I): -3 / 冗長度: 5.8 % / Rmerge(I) obs: 0.144 / Net I/σ(I): 10.6 |
| 反射 シェル | 解像度: 2.8→2.9 Å / 冗長度: 5.5 % / Rmerge(I) obs: 0.788 / Mean I/σ(I) obs: 1.8 / Num. unique all: 1907 / % possible all: 99.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 3ODU 解像度: 2.8→30.488 Å / SU ML: 0.86 / 交差検証法: THROUGHOUT / σ(F): 1.37 / 位相誤差: 29.73 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 59.885 Å2 / ksol: 0.319 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→30.488 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

















