| 登録構造単位 | A: SEPF-LIKE PROTEIN
B: SEPF-LIKE PROTEIN
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 18,992 | 2 |
|---|
| ポリマ- | 18,992 | 2 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 522 | 29 |
|---|
|
|---|
| 1 | A: SEPF-LIKE PROTEIN
A: SEPF-LIKE PROTEIN
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 18,992 | 2 |
|---|
| ポリマ- | 18,992 | 2 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 36 | 2 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 | | crystal symmetry operation | 12_574 | x,x-y+2,-z-1/6 | 1 |
| Buried area | 1680 Å2 |
|---|
| ΔGint | -12 kcal/mol |
|---|
| Surface area | 8850 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 2 | B: SEPF-LIKE PROTEIN
B: SEPF-LIKE PROTEIN
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 18,992 | 2 |
|---|
| ポリマ- | 18,992 | 2 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 36 | 2 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 | | crystal symmetry operation | 12_574 | x,x-y+2,-z-1/6 | 1 |
| Buried area | 1710 Å2 |
|---|
| ΔGint | -10 kcal/mol |
|---|
| Surface area | 9270 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 単位格子 | | Length a, b, c (Å) | 59.327, 59.327, 177.484 |
|---|
| Angle α, β, γ (deg.) | 90.00, 90.00, 120.00 |
|---|
| Int Tables number | 179 |
|---|
| Space group name H-M | P6522 |
|---|
|
|---|
| Components on special symmetry positions | | ID | モデル | 要素 |
|---|
| 1 | 1 | A-113-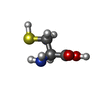 CYS | | 2 | 1 | A-2014- HOH | | 3 | 1 | B-2009- HOH |
|
|---|
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: | Dom-ID | Component-ID | Ens-ID | Selection details |
|---|
| 1 | 1 | 1 | CHAIN A AND (RESSEQ 51:112 )| 2 | 1 | 1 | CHAIN B AND (RESSEQ 51:112 )| 1 | 1 | 2 | CHAIN A AND (RESSEQ 114:128 )| 2 | 1 | 2 | CHAIN B AND (RESSEQ 114:128 ) | | | |
NCSアンサンブル:
NCS oper: | ID | Code | Matrix | ベクター |
|---|
| 1 | given(0.877306, 0.198287, -0.437054), (0.166965, 0.727675, 0.665291), (0.449952, -0.656637, 0.605286)9.3564, 46.3102, 42.7424| 2 | given(0.892401, 0.183658, -0.412176), (0.167554, 0.713255, 0.680583), (0.418981, -0.676415, 0.605738)| 11.223, 47.1033, 41.8967 | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード CELL CYCLE (細胞周期)
CELL CYCLE (細胞周期) 機能・相同性情報
機能・相同性情報

 PYROCOCCUS FURIOSUS COM1 (ピュロコックス・フリオスス)
PYROCOCCUS FURIOSUS COM1 (ピュロコックス・フリオスス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2.5 Å
多波長異常分散 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3zig.cif.gz
3zig.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3zig.ent.gz
pdb3zig.ent.gz PDB形式
PDB形式 3zig.json.gz
3zig.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/zi/3zig
https://data.pdbj.org/pub/pdb/validation_reports/zi/3zig ftp://data.pdbj.org/pub/pdb/validation_reports/zi/3zig
ftp://data.pdbj.org/pub/pdb/validation_reports/zi/3zig リンク
リンク 集合体
集合体


 要素
要素

 PYROCOCCUS FURIOSUS COM1 (ピュロコックス・フリオスス)
PYROCOCCUS FURIOSUS COM1 (ピュロコックス・フリオスス)
 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C41 / 参照: UniProt: I6V3Q6
ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C41 / 参照: UniProt: I6V3Q6 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID23-1 / 波長: 0.9793
/ ビームライン: ID23-1 / 波長: 0.9793  : 0.9793 Å / 相対比: 1
: 0.9793 Å / 相対比: 1  解析
解析 :
:  多波長異常分散
多波長異常分散 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

