| 登録情報 | データベース: PDB / ID: 3m6f
|
|---|
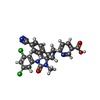
| タイトル | CD11A I-domain complexed with 6-((5S,9R)-9-(4-CYANOPHENYL)-3-(3,5-DICHLOROPHENYL)-1-METHYL-2,4-DIOXO-1,3,7- TRIAZASPIRO[4.4]NON-7-YL)NICOTINIC ACID |
|---|
 要素 要素 | Integrin alpha-L |
|---|
 キーワード キーワード |  CELL ADHESION (細胞接着) / CELL ADHESION (細胞接着) /  LFA1 / LFA1 /  INHIBITOR (酵素阻害剤) / INHIBITOR (酵素阻害剤) /  Alternative splicing (選択的スプライシング) / Alternative splicing (選択的スプライシング) /  Calcium (カルシウム) / Calcium (カルシウム) /  Disulfide bond (ジスルフィド) / Disulfide bond (ジスルフィド) /  Glycoprotein (糖タンパク質) / Glycoprotein (糖タンパク質) /  Integrin (インテグリン) / Integrin (インテグリン) /  Magnesium (マグネシウム) / Magnesium (マグネシウム) /  Membrane (生体膜) / Polymorphism / Receptor / Membrane (生体膜) / Polymorphism / Receptor /  Transmembrane (膜貫通型タンパク質) Transmembrane (膜貫通型タンパク質) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.85 Å 分子置換 / 解像度: 1.85 Å |
|---|
 データ登録者 データ登録者 | Sheriff, S. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2010タイトル: Small molecule antagonist of leukocyte function associated antigen-1 (LFA-1): structure-activity relationships leading to the identification of 6-((5S,9R)-9-(4-cyanophenyl)-3-(3,5- ...タイトル: Small molecule antagonist of leukocyte function associated antigen-1 (LFA-1): structure-activity relationships leading to the identification of 6-((5S,9R)-9-(4-cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro[4.4]nonan-7-yl)nicotinic acid (BMS-688521).著者: Watterson, S.H. / Xiao, Z. / Dodd, D.S. / Tortolani, D.R. / Vaccaro, W. / Potin, D. / Launay, M. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Lee, D. / Yang, X. / McIntyre, K.W. / Balimane, P. ...著者: Watterson, S.H. / Xiao, Z. / Dodd, D.S. / Tortolani, D.R. / Vaccaro, W. / Potin, D. / Launay, M. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Lee, D. / Yang, X. / McIntyre, K.W. / Balimane, P. / Patel, K. / Yang, Z. / Marathe, P. / Kadiyala, P. / Tebben, A.J. / Sheriff, S. / Chang, C.Y. / Ziemba, T. / Zhang, H. / Chen, B.C. / DelMonte, A.J. / Aranibar, N. / McKinnon, M. / Barrish, J.C. / Suchard, S.J. / Murali Dhar, T.G. ジャーナル: J.Med.Chem. / 年: 2010タイトル: Small molecule antagonist of leukocyte function associated antigen-1 (LFA-1): structure-activity relationships leading to the identification of 6-((5S,9R)-9-(4-cyanophenyl)-3-(3,5- ...タイトル: Small molecule antagonist of leukocyte function associated antigen-1 (LFA-1): structure-activity relationships leading to the identification of 6-((5S,9R)-9-(4-cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro[4.4]nonan-7-yl)nicotinic acid (BMS-688521).著者: Watterson, S.H. / Xiao, Z. / Dodd, D.S. / Tortolani, D.R. / Vaccaro, W. / Potin, D. / Launay, M. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Lee, D. / Yang, X. / McIntyre, K.W. / Balimane, P. ...著者: Watterson, S.H. / Xiao, Z. / Dodd, D.S. / Tortolani, D.R. / Vaccaro, W. / Potin, D. / Launay, M. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Lee, D. / Yang, X. / McIntyre, K.W. / Balimane, P. / Patel, K. / Yang, Z. / Marathe, P. / Kadiyala, P. / Tebben, A.J. / Sheriff, S. / Chang, C.Y. / Ziemba, T. / Zhang, H. / Chen, B.C. / DelMonte, A.J. / Aranibar, N. / McKinnon, M. / Barrish, J.C. / Suchard, S.J. / Murali Dhar, T.G.#1:  ジャーナル: J.Med.Chem. / 年: 2006タイトル: Discovery and Development of 5-[(5S,9R)-9-(4-Cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro [4.4]non-7-yl]methyl]-3-thiophenecarboxylicacid (BMS-587101) -- ...タイトル: Discovery and Development of 5-[(5S,9R)-9-(4-Cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro [4.4]non-7-yl]methyl]-3-thiophenecarboxylicacid (BMS-587101) -- Small Molecule Antagonist of LFA-1著者: Potin, D. / Launay, M. / Monatlik, F. / Malabre, P. / Fabreguettes, M. / Fouquet, A. / Maillet, M. / Nicolai, E. / Dorgeret, L. / Chevallier, F. / Besse, D. / Dufort, M. / Caussade, F. / ...著者: Potin, D. / Launay, M. / Monatlik, F. / Malabre, P. / Fabreguettes, M. / Fouquet, A. / Maillet, M. / Nicolai, E. / Dorgeret, L. / Chevallier, F. / Besse, D. / Dufort, M. / Caussade, F. / Ahmad, S.Z. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Balimane, P. / Patel, K. / Yang, Z. / Marathe, P. / Postelneck, J. / Townsend, R.M. / Sheriff, S. / Einspahr, H. / Kish, K. / Malley, M.F. / Gougoutas, J.Z. / Kadiyala, P. / Cheney, D.L. / Tejwani, R.W. / Murphy, D.K. / Mcintyre, K.W. / Yang, X. / Chao, S. / Leith, L. / Xiao, Z. / Mathur, A. / Chen, B.-C. / Wu, D.-R. / Traeger, S.C. / McKinnon, M. / Barrish, J.C. / Robl, J.A. / Iwanowicz, E.J. / Suchard, S.J. / Dhar, T.G.M. ジャーナル: J.Med.Chem. / 年: 2006タイトル: Discovery and Development of 5-[(5S,9R)-9-(4-Cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro [4.4]non-7-yl]methyl]-3-thiophenecarboxylicacid (BMS-587101) -- ...タイトル: Discovery and Development of 5-[(5S,9R)-9-(4-Cyanophenyl)-3-(3,5-dichlorophenyl)-1-methyl-2,4-dioxo-1,3,7-triazaspiro [4.4]non-7-yl]methyl]-3-thiophenecarboxylicacid (BMS-587101) -- Small Molecule Antagonist of LFA-1著者: Potin, D. / Launay, M. / Monatlik, F. / Malabre, P. / Fabreguettes, M. / Fouquet, A. / Maillet, M. / Nicolai, E. / Dorgeret, L. / Chevallier, F. / Besse, D. / Dufort, M. / Caussade, F. / ...著者: Potin, D. / Launay, M. / Monatlik, F. / Malabre, P. / Fabreguettes, M. / Fouquet, A. / Maillet, M. / Nicolai, E. / Dorgeret, L. / Chevallier, F. / Besse, D. / Dufort, M. / Caussade, F. / Ahmad, S.Z. / Stetsko, D.K. / Skala, S. / Davis, P.M. / Balimane, P. / Patel, K. / Yang, Z. / Marathe, P. / Postelneck, J. / Townsend, R.M. / Sheriff, S. / Einspahr, H. / Kish, K. / Malley, M.F. / Gougoutas, J.Z. / Kadiyala, P. / Cheney, D.L. / Tejwani, R.W. / Murphy, D.K. / Mcintyre, K.W. / Yang, X. / Chao, S. / Leith, L. / Xiao, Z. / Mathur, A. / Chen, B.-C. / Wu, D.-R. / Traeger, S.C. / McKinnon, M. / Barrish, J.C. / Robl, J.A. / Iwanowicz, E.J. / Suchard, S.J. / Dhar, T.G.M. |
|---|
| 履歴 | | 登録 | 2010年3月15日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2010年5月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2018年4月4日 | Group: Advisory / Data collection / カテゴリ: diffrn_source / pdbx_unobs_or_zero_occ_atoms / Item: _diffrn_source.type |
|---|
| 改定 1.3 | 2018年4月11日 | Group: Data collection / カテゴリ: diffrn_source / Item: _diffrn_source.source |
|---|
| 改定 1.4 | 2023年9月6日 | Group: Advisory / Data collection ...Advisory / Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_unobs_or_zero_occ_atoms / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード CELL ADHESION (細胞接着) /
CELL ADHESION (細胞接着) /  LFA1 /
LFA1 /  INHIBITOR (酵素阻害剤) /
INHIBITOR (酵素阻害剤) /  Alternative splicing (選択的スプライシング) /
Alternative splicing (選択的スプライシング) /  Calcium (カルシウム) /
Calcium (カルシウム) /  Disulfide bond (ジスルフィド) /
Disulfide bond (ジスルフィド) /  Glycoprotein (糖タンパク質) /
Glycoprotein (糖タンパク質) /  Integrin (インテグリン) /
Integrin (インテグリン) /  Magnesium (マグネシウム) /
Magnesium (マグネシウム) /  Membrane (生体膜) / Polymorphism / Receptor /
Membrane (生体膜) / Polymorphism / Receptor /  Transmembrane (膜貫通型タンパク質)
Transmembrane (膜貫通型タンパク質) 機能・相同性情報
機能・相同性情報 integrin complex / heterophilic cell-cell adhesion via plasma membrane cell adhesion molecules / cell adhesion mediated by integrin / leukocyte cell-cell adhesion / receptor clustering ...memory T cell extravasation / integrin alphaL-beta2 complex / T cell activation via T cell receptor contact with antigen bound to MHC molecule on antigen presenting cell / ICAM-3 receptor activity / RUNX3 Regulates Immune Response and Cell Migration /
integrin complex / heterophilic cell-cell adhesion via plasma membrane cell adhesion molecules / cell adhesion mediated by integrin / leukocyte cell-cell adhesion / receptor clustering ...memory T cell extravasation / integrin alphaL-beta2 complex / T cell activation via T cell receptor contact with antigen bound to MHC molecule on antigen presenting cell / ICAM-3 receptor activity / RUNX3 Regulates Immune Response and Cell Migration /  integrin complex / heterophilic cell-cell adhesion via plasma membrane cell adhesion molecules / cell adhesion mediated by integrin / leukocyte cell-cell adhesion / receptor clustering / Integrin cell surface interactions / specific granule membrane /
integrin complex / heterophilic cell-cell adhesion via plasma membrane cell adhesion molecules / cell adhesion mediated by integrin / leukocyte cell-cell adhesion / receptor clustering / Integrin cell surface interactions / specific granule membrane /  食作用 /
食作用 /  cell adhesion molecule binding / cell-matrix adhesion / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall /
cell adhesion molecule binding / cell-matrix adhesion / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall /  細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell /
細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell /  integrin binding /
integrin binding /  細胞接着 /
細胞接着 /  炎症 / external side of plasma membrane / Neutrophil degranulation /
炎症 / external side of plasma membrane / Neutrophil degranulation /  細胞膜 /
細胞膜 /  シグナル伝達 / extracellular exosome /
シグナル伝達 / extracellular exosome /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.85 Å
分子置換 / 解像度: 1.85 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2010
ジャーナル: J.Med.Chem. / 年: 2010 ジャーナル: J.Med.Chem. / 年: 2006
ジャーナル: J.Med.Chem. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3m6f.cif.gz
3m6f.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3m6f.ent.gz
pdb3m6f.ent.gz PDB形式
PDB形式 3m6f.json.gz
3m6f.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/m6/3m6f
https://data.pdbj.org/pub/pdb/validation_reports/m6/3m6f ftp://data.pdbj.org/pub/pdb/validation_reports/m6/3m6f
ftp://data.pdbj.org/pub/pdb/validation_reports/m6/3m6f
 リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: ITGAL, CD11A / プラスミド: PET11C / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ITGAL, CD11A / プラスミド: PET11C / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P20701
Escherichia coli (大腸菌) / 参照: UniProt: P20701 硝酸塩
硝酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E / 波長: 1.54178 Å
回転陽極 / タイプ: RIGAKU FR-E / 波長: 1.54178 Å : 1.54178 Å / 相対比: 1
: 1.54178 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj