| 登録情報 | データベース: PDB / ID: 3kqi
|
|---|
| タイトル | crystal structure of PHF2 PHD domain complexed with H3K4Me3 peptide |
|---|
 要素 要素 | - H3K4Me3 peptide
- PHD finger protein 2

|
|---|
 キーワード キーワード |  NUCLEAR PROTEIN / NUCLEAR PROTEIN /  PHD finger / Metal-binding / PHD finger / Metal-binding /  Zinc-finger (ジンクフィンガー) / Histone-binding Zinc-finger (ジンクフィンガー) / Histone-binding |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
histone H4K20 demethylase activity / negative regulation of rDNA heterochromatin formation /  酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization ...histone H4K20 demethylase activity / negative regulation of rDNA heterochromatin formation / 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization ...histone H4K20 demethylase activity / negative regulation of rDNA heterochromatin formation /  酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication / 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / liver development / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / liver development / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape /  transcription coregulator activity / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / transcription coregulator activity / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines /  動原体 / RMTs methylate histone arginines / 動原体 / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / 遺伝的組換え / Pre-NOTCH Transcription and Translation / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /  ヌクレオソーム / ヌクレオソーム /  nucleosome assembly / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production / nucleosome assembly / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production /  遺伝子発現 / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression / 遺伝子発現 / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /  transcription coactivator activity / transcription coactivator activity /  cadherin binding / iron ion binding / protein heterodimerization activity / Amyloid fiber formation / cadherin binding / iron ion binding / protein heterodimerization activity / Amyloid fiber formation /  核小体 / regulation of transcription by RNA polymerase II / protein-containing complex / 核小体 / regulation of transcription by RNA polymerase II / protein-containing complex /  DNA binding / extracellular exosome / zinc ion binding / extracellular region / DNA binding / extracellular exosome / zinc ion binding / extracellular region /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核類似検索 - 分子機能 細胞核類似検索 - 分子機能 Jumonji, helical domain / Jumonji helical domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / JmjC domain, hydroxylase / A domain family that is part of the cupin metalloenzyme superfamily. / JmjC domain / JmjC domain profile. /  Zinc finger, PHD-type, conserved site / PHD-finger ...Jumonji, helical domain / Jumonji helical domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / JmjC domain, hydroxylase / A domain family that is part of the cupin metalloenzyme superfamily. / JmjC domain / JmjC domain profile. / Zinc finger, PHD-type, conserved site / PHD-finger ...Jumonji, helical domain / Jumonji helical domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / JmjC domain, hydroxylase / A domain family that is part of the cupin metalloenzyme superfamily. / JmjC domain / JmjC domain profile. /  Zinc finger, PHD-type, conserved site / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-type, conserved site / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. /  Zinc finger, PHD-finger / Histone H3 signature 1. / Zinc finger, PHD-finger / Histone H3 signature 1. /  Zinc finger, PHD-type / PHD zinc finger / Histone H3 signature 2. / Zinc finger, PHD-type / PHD zinc finger / Histone H3 signature 2. /  ヒストンH3 / Histone H3/CENP-A / Histone H2A/H2B/H3 / Core histone H2A/H2B/H3/H4 / ヒストンH3 / Histone H3/CENP-A / Histone H2A/H2B/H3 / Core histone H2A/H2B/H3/H4 /  Zinc finger, FYVE/PHD-type / Histone-fold / Zinc finger, FYVE/PHD-type / Histone-fold /  Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.78 Å 単波長異常分散 / 解像度: 1.78 Å |
|---|
 データ登録者 データ登録者 | Wen, H. / Li, J.Z. / Song, T. / Lu, M. / Lee, M. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2010 ジャーナル: J.Biol.Chem. / 年: 2010
タイトル: Recognition of histone H3K4 trimethylation by the plant homeodomain of PHF2 modulates histone demethylation.
著者: Wen, H. / Li, J. / Song, T. / Lu, M. / Kan, P.Y. / Lee, M.G. / Sha, B. / Shi, X. |
|---|
| 履歴 | | 登録 | 2009年11月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2010年2月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Advisory / Version format compliance |
|---|
| 改定 1.2 | 2019年2月13日 | Group: Advisory / Data collection / Derived calculations
カテゴリ: pdbx_distant_solvent_atoms / pdbx_struct_assembly ...pdbx_distant_solvent_atoms / pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop / pdbx_struct_oper_list / struct_conn
Item: _pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details ..._pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_assembly_prop.value / _pdbx_struct_oper_list.matrix[1][1] / _pdbx_struct_oper_list.matrix[1][2] / _pdbx_struct_oper_list.matrix[2][1] / _pdbx_struct_oper_list.matrix[2][2] / _pdbx_struct_oper_list.matrix[3][3] / _pdbx_struct_oper_list.name / _pdbx_struct_oper_list.symmetry_operation / _pdbx_struct_oper_list.type / _pdbx_struct_oper_list.vector[1] / _pdbx_struct_oper_list.vector[2] / _pdbx_struct_oper_list.vector[3] / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード NUCLEAR PROTEIN /
NUCLEAR PROTEIN /  PHD finger / Metal-binding /
PHD finger / Metal-binding /  Zinc-finger (ジンクフィンガー) / Histone-binding
Zinc-finger (ジンクフィンガー) / Histone-binding 機能・相同性情報
機能・相同性情報 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization ...histone H4K20 demethylase activity / negative regulation of rDNA heterochromatin formation /
酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization ...histone H4K20 demethylase activity / negative regulation of rDNA heterochromatin formation /  酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /
酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む / protein demethylation / histone H3K9 demethylase activity / Chromatin modifying enzymes / epigenetic regulation of gene expression / transcription initiation-coupled chromatin remodeling / methylated histone binding / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / liver development / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape /
DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / liver development / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape /  transcription coregulator activity / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines /
transcription coregulator activity / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines /  動原体 / RMTs methylate histone arginines /
動原体 / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /
遺伝的組換え / Pre-NOTCH Transcription and Translation / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /  ヌクレオソーム /
ヌクレオソーム /  nucleosome assembly / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production /
nucleosome assembly / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production /  遺伝子発現 / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /
遺伝子発現 / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /  transcription coactivator activity /
transcription coactivator activity /  cadherin binding / iron ion binding / protein heterodimerization activity / Amyloid fiber formation /
cadherin binding / iron ion binding / protein heterodimerization activity / Amyloid fiber formation /  核小体 / regulation of transcription by RNA polymerase II / protein-containing complex /
核小体 / regulation of transcription by RNA polymerase II / protein-containing complex /  DNA binding / extracellular exosome / zinc ion binding / extracellular region /
DNA binding / extracellular exosome / zinc ion binding / extracellular region /  核質 /
核質 /  生体膜 /
生体膜 /  細胞核
細胞核
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 1.78 Å
単波長異常分散 / 解像度: 1.78 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2010
ジャーナル: J.Biol.Chem. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3kqi.cif.gz
3kqi.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3kqi.ent.gz
pdb3kqi.ent.gz PDB形式
PDB形式 3kqi.json.gz
3kqi.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/kq/3kqi
https://data.pdbj.org/pub/pdb/validation_reports/kq/3kqi ftp://data.pdbj.org/pub/pdb/validation_reports/kq/3kqi
ftp://data.pdbj.org/pub/pdb/validation_reports/kq/3kqi リンク
リンク 集合体
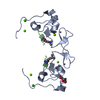
集合体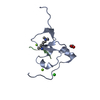
 要素
要素 / GRC5
/ GRC5
 Homo sapiens (ヒト) / 遺伝子: KIAA0662, PHF2 / プラスミド: PGEX6P1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: KIAA0662, PHF2 / プラスミド: PGEX6P1 / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O75151
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O75151








 グリセリン
グリセリン 塩化物
塩化物 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-ID / 波長: 1 Å
/ ビームライン: 22-ID / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  単波長異常分散 / 解像度: 1.78→7.99 Å / Cor.coef. Fo:Fc: 0.955 / Cor.coef. Fo:Fc free: 0.943 / SU B: 3.07 / SU ML: 0.059 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.101 / ESU R Free: 0.1 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
単波長異常分散 / 解像度: 1.78→7.99 Å / Cor.coef. Fo:Fc: 0.955 / Cor.coef. Fo:Fc free: 0.943 / SU B: 3.07 / SU ML: 0.059 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.101 / ESU R Free: 0.1 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj












