| 登録情報 | データベース: PDB / ID: 2vh1
|
|---|
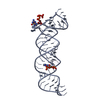
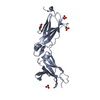
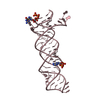
| タイトル | Crystal structure of bacterial cell division protein FtsQ from E.coli |
|---|
 要素 要素 | CELL DIVISION PROTEIN FTSQ 細胞分裂 細胞分裂 |
|---|
 キーワード キーワード |  CELL CYCLE (細胞周期) / FTSQ / CELL CYCLE (細胞周期) / FTSQ /  POTRA / POTRA /  MEMBRANE (生体膜) / MEMBRANE (生体膜) /  SEPTATION / SEPTATION /  CELL DIVISION (細胞分裂) / CELL DIVISION (細胞分裂) /  TRANSMEMBRANE (膜貫通型タンパク質) / INNER MEMBRANE TRANSMEMBRANE (膜貫通型タンパク質) / INNER MEMBRANE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
FtsQBL complex /  divisome complex / FtsZ-dependent cytokinesis / division septum assembly / cell division site / divisome complex / FtsZ-dependent cytokinesis / division septum assembly / cell division site /  細胞分裂 / identical protein binding / 細胞分裂 / identical protein binding /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能  Cell division protein FtsQ/DivIB / Cell division protein FtsQ/DivIB /  Cell division protein FtsQ, C-terminal / Cell division protein FtsQ, C-terminal /  Cell division protein FtsQ / Cell division protein FtsQ /  Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type / Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type /  Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type / Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type /  membrane protein fhac / membrane protein fhac /  POTRA domain / POTRA domain profile. ... POTRA domain / POTRA domain profile. ... Cell division protein FtsQ/DivIB / Cell division protein FtsQ/DivIB /  Cell division protein FtsQ, C-terminal / Cell division protein FtsQ, C-terminal /  Cell division protein FtsQ / Cell division protein FtsQ /  Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type / Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type /  Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type / Cell division protein FtsQ/DivIB, C-terminal / POTRA domain, FtsQ-type /  membrane protein fhac / membrane protein fhac /  POTRA domain / POTRA domain profile. / Ubiquitin-like (UB roll) / Roll / POTRA domain / POTRA domain profile. / Ubiquitin-like (UB roll) / Roll /  ロスマンフォールド / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 ロスマンフォールド / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |    ESCHERICHIA COLI (大腸菌) ESCHERICHIA COLI (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.7 Å 多波長異常分散 / 解像度: 2.7 Å |
|---|
 データ登録者 データ登録者 | van den Ent, F. / Vinkenvleugel, T. / Ind, A. / West, P. / Veprintsev, D. / Naninga, N. / den Blaauwen, T. / Lowe, J. |
|---|
 引用 引用 |  ジャーナル: Mol.Microbiol. / 年: 2008 ジャーナル: Mol.Microbiol. / 年: 2008
タイトル: Structural and Mutational Analysis of Cell Division Protein Ftsq
著者: van den Ent, F. / Vinkenvleugel, T. / Ind, A. / West, P. / Veprintsev, D. / Naninga, N. / den Blaauwen, T. / Lowe, J. |
|---|
| 履歴 | | 登録 | 2007年11月16日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2008年3月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Advisory / Version format compliance |
|---|
| 改定 1.2 | 2018年1月24日 | Group: Database references / Source and taxonomy / カテゴリ: citation_author / entity_src_gen
Item: _citation_author.name / _entity_src_gen.pdbx_host_org_ncbi_taxonomy_id ..._citation_author.name / _entity_src_gen.pdbx_host_org_ncbi_taxonomy_id / _entity_src_gen.pdbx_host_org_scientific_name / _entity_src_gen.pdbx_host_org_strain / _entity_src_gen.pdbx_host_org_variant |
|---|
| 改定 1.3 | 2024年5月8日 | Group: Data collection / Database references ...Data collection / Database references / Other / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / struct_ncs_dom_lim
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _struct_ncs_dom_lim.beg_auth_comp_id / _struct_ncs_dom_lim.beg_label_asym_id / _struct_ncs_dom_lim.beg_label_comp_id / _struct_ncs_dom_lim.beg_label_seq_id / _struct_ncs_dom_lim.end_auth_comp_id / _struct_ncs_dom_lim.end_label_asym_id / _struct_ncs_dom_lim.end_label_comp_id / _struct_ncs_dom_lim.end_label_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 細胞分裂
細胞分裂  キーワード
キーワード CELL CYCLE (細胞周期) / FTSQ /
CELL CYCLE (細胞周期) / FTSQ /  POTRA /
POTRA /  MEMBRANE (生体膜) /
MEMBRANE (生体膜) /  SEPTATION /
SEPTATION /  CELL DIVISION (細胞分裂) /
CELL DIVISION (細胞分裂) /  TRANSMEMBRANE (膜貫通型タンパク質) / INNER MEMBRANE
TRANSMEMBRANE (膜貫通型タンパク質) / INNER MEMBRANE 機能・相同性情報
機能・相同性情報 divisome complex / FtsZ-dependent cytokinesis / division septum assembly / cell division site /
divisome complex / FtsZ-dependent cytokinesis / division septum assembly / cell division site /  細胞分裂 / identical protein binding /
細胞分裂 / identical protein binding /  細胞膜
細胞膜
 ESCHERICHIA COLI (大腸菌)
ESCHERICHIA COLI (大腸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2.7 Å
多波長異常分散 / 解像度: 2.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Mol.Microbiol. / 年: 2008
ジャーナル: Mol.Microbiol. / 年: 2008 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2vh1.cif.gz
2vh1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2vh1.ent.gz
pdb2vh1.ent.gz PDB形式
PDB形式 2vh1.json.gz
2vh1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/vh/2vh1
https://data.pdbj.org/pub/pdb/validation_reports/vh/2vh1 ftp://data.pdbj.org/pub/pdb/validation_reports/vh/2vh1
ftp://data.pdbj.org/pub/pdb/validation_reports/vh/2vh1 リンク
リンク 集合体
集合体


 :
:  要素
要素 細胞分裂 / FTSQ
細胞分裂 / FTSQ
 ESCHERICHIA COLI (大腸菌) / プラスミド: PHIS17 / 発現宿主:
ESCHERICHIA COLI (大腸菌) / プラスミド: PHIS17 / 発現宿主: 
 ESCHERICHIA COLI BL21(DE3) (大腸菌) / Variant (発現宿主): C41 / 参照: UniProt: P06136
ESCHERICHIA COLI BL21(DE3) (大腸菌) / Variant (発現宿主): C41 / 参照: UniProt: P06136 X線回折
X線回折 試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID23-1 / 波長: 0.9392
/ ビームライン: ID23-1 / 波長: 0.9392  : 0.9392 Å / 相対比: 1
: 0.9392 Å / 相対比: 1  解析
解析 :
:  多波長異常分散
多波長異常分散 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj