+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2kht | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | NMR Structure of human alpha defensin HNP-1 | ||||||
 要素 要素 | Neutrophil defensin 1 | ||||||
 キーワード キーワード |  ANTIMICROBIAL PROTEIN (抗微生物ペプチド) / ANTIMICROBIAL PROTEIN (抗微生物ペプチド) /  microcrystalline protein / microcrystalline protein /  human alpha defensin / human alpha defensin /  Defensin (ディフェンシン) / Defensin (ディフェンシン) /  Secreted (分泌) / Secreted (分泌) /  Antibiotic (抗生物質) / Antiviral defense / Antibiotic (抗生物質) / Antiviral defense /  Fungicide (殺菌剤 (農薬その他)) / Fungicide (殺菌剤 (農薬その他)) /  Phosphoprotein Phosphoprotein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pore-forming activity /  ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus / ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus /  innate immune response in mucosa ...pore-forming activity / innate immune response in mucosa ...pore-forming activity /  ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus / ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus /  innate immune response in mucosa / Golgi lumen / innate immune response in mucosa / Golgi lumen /  走化性 / azurophil granule lumen / antimicrobial humoral immune response mediated by antimicrobial peptide / antibacterial humoral response / collagen-containing extracellular matrix / defense response to virus / killing of cells of another organism / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / defense response to Gram-positive bacterium / 走化性 / azurophil granule lumen / antimicrobial humoral immune response mediated by antimicrobial peptide / antibacterial humoral response / collagen-containing extracellular matrix / defense response to virus / killing of cells of another organism / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / defense response to Gram-positive bacterium /  免疫応答 / Neutrophil degranulation / 免疫応答 / Neutrophil degranulation /  extracellular space / extracellular exosome / extracellular region extracellular space / extracellular exosome / extracellular region類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 個体NMR / DGSA-distance geometry simulated annealing,  simulated annealing simulated annealing | ||||||
| Model details | minimized average, model 1 | ||||||
 データ登録者 データ登録者 | Zhang, Y. / Li, S. / Doherty, T.F. / Lubkowski, J. / Lu, W. / Li, J. / Barinka, C. / Hong, M. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2010 ジャーナル: J.Mol.Biol. / 年: 2010タイトル: Resonance assignment and three-dimensional structure determination of a human alpha-defensin, HNP-1, by solid-state NMR. 著者: Zhang, Y. / Doherty, T. / Li, J. / Lu, W. / Barinka, C. / Lubkowski, J. / Hong, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2kht.cif.gz 2kht.cif.gz | 98.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2kht.ent.gz pdb2kht.ent.gz | 79.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2kht.json.gz 2kht.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kh/2kht https://data.pdbj.org/pub/pdb/validation_reports/kh/2kht ftp://data.pdbj.org/pub/pdb/validation_reports/kh/2kht ftp://data.pdbj.org/pub/pdb/validation_reports/kh/2kht | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 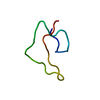
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 3452.111 Da / 分子数: 1 / 断片: Residues 65-94 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: DEF1, DEFA1, DEFA2, MRS / プラスミド: pGEX-2T / 発現宿主: Homo sapiens (ヒト) / 遺伝子: DEF1, DEFA1, DEFA2, MRS / プラスミド: pGEX-2T / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P59665 Escherichia coli (大腸菌) / 参照: UniProt: P59665 |
|---|
-実験情報
-実験
| 実験 | 手法: 個体NMR 詳細: HNP-1 structure determination through 2D & 3D CC and NC correlation experiments by solid state NMR. | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: Polycrystalline sample grown from 60% w/v PEG400, 30 mM Cacodylate, 60 mM Lithium Sulfate | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: DGSA-distance geometry simulated annealing,  simulated annealing simulated annealingソフトェア番号: 1 | ||||||||||||||||||||
| NMR constraints | Protein chi angle constraints total count: 0 / Protein other angle constraints total count: 0 / Protein phi angle constraints total count: 28 / Protein psi angle constraints total count: 28 | ||||||||||||||||||||
| 代表構造 | 選択基準: minimized average | ||||||||||||||||||||
| NMRアンサンブル | Average torsion angle constraint violation: 12.41 ° コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 10 / Maximum lower distance constraint violation: 0.53 Å / Maximum torsion angle constraint violation: 22.622 ° / Maximum upper distance constraint violation: 2.68 Å / Torsion angle constraint violation method: TALOS | ||||||||||||||||||||
| NMR ensemble rms | Distance rms dev: 0.58 Å |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


