| 登録情報 | データベース: PDB / ID: 1oyi
|
|---|
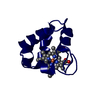
| タイトル | Solution structure of the Z-DNA binding domain of the vaccinia virus gene E3L |
|---|
 要素 要素 | double-stranded RNA-binding protein |
|---|
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / (alpha+beta) helix-turn-helix VIRAL PROTEIN (ウイルスタンパク質) / (alpha+beta) helix-turn-helix |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |    Vaccinia virus (ワクチニアウイルス) Vaccinia virus (ワクチニアウイルス) |
|---|
| 手法 |  溶液NMR / torsion angle dynamics 溶液NMR / torsion angle dynamics |
|---|
 データ登録者 データ登録者 | Kahmann, J.D. / Wecking, D.A. / Putter, V. / Lowenhaupt, K. / Kim, Y.-G. / Schmieder, P. / Oschkinat, H. / Rich, A. / Schade, M. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2004 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2004
タイトル: The solution structure of the N-terminal domain of E3L shows a tyrosine conformation that may explain its reduced affinity to Z-DNA in vitro.
著者: Kahmann, J.D. / Wecking, D.A. / Putter, V. / Lowenhaupt, K. / Kim, Y.-G. / Schmieder, P. / Oschkinat, H. / Rich, A. / Schade, M. |
|---|
| 履歴 | | 登録 | 2003年4月4日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2004年3月9日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月29日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年2月23日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
| Remark 999 | SEQUENCE THESE RESIDUES ARE VECTOR-ENCODED. |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード VIRAL PROTEIN (ウイルスタンパク質) / (alpha+beta) helix-turn-helix
VIRAL PROTEIN (ウイルスタンパク質) / (alpha+beta) helix-turn-helix 機能・相同性情報
機能・相同性情報 RNA binding
RNA binding
 Vaccinia virus (ワクチニアウイルス)
Vaccinia virus (ワクチニアウイルス) 溶液NMR / torsion angle dynamics
溶液NMR / torsion angle dynamics  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2004
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1oyi.cif.gz
1oyi.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1oyi.ent.gz
pdb1oyi.ent.gz PDB形式
PDB形式 1oyi.json.gz
1oyi.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/oy/1oyi
https://data.pdbj.org/pub/pdb/validation_reports/oy/1oyi ftp://data.pdbj.org/pub/pdb/validation_reports/oy/1oyi
ftp://data.pdbj.org/pub/pdb/validation_reports/oy/1oyi リンク
リンク 集合体
集合体
 要素
要素
 Vaccinia virus (ワクチニアウイルス)
Vaccinia virus (ワクチニアウイルス) / 遺伝子: E3L / プラスミド: pET28 / 生物種 (発現宿主): Escherichia coli / 発現宿主:
/ 遺伝子: E3L / プラスミド: pET28 / 生物種 (発現宿主): Escherichia coli / 発現宿主: 
 Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q86638
Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q86638 溶液NMR
溶液NMR 試料調製
試料調製
 : DRX / 磁場強度: 600 MHz
: DRX / 磁場強度: 600 MHz 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
