+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1h4o | ||||||
|---|---|---|---|---|---|---|---|
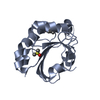
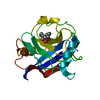
| タイトル | Monoclinic form of human peroxiredoxin 5 | ||||||
 要素 要素 | PEROXIREDOXIN 5 ペルオキシレドキシン ペルオキシレドキシン | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / ANTIOXIDANT ENZYME / THIOREDOXIN PEROTHIOREDOXIN / FOLDXIDASE OXIDOREDUCTASE (酸化還元酵素) / ANTIOXIDANT ENZYME / THIOREDOXIN PEROTHIOREDOXIN / FOLDXIDASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peroxynitrite reductase activity / reactive nitrogen species metabolic process / negative regulation of transcription by RNA polymerase III / negative regulation of oxidoreductase activity / NADPH oxidation / regulation of apoptosis involved in tissue homeostasis / RNA polymerase III transcription regulatory region sequence-specific DNA binding / thioredoxin-dependent peroxiredoxin / thioredoxin peroxidase activity / Detoxification of Reactive Oxygen Species ...peroxynitrite reductase activity / reactive nitrogen species metabolic process / negative regulation of transcription by RNA polymerase III / negative regulation of oxidoreductase activity / NADPH oxidation / regulation of apoptosis involved in tissue homeostasis / RNA polymerase III transcription regulatory region sequence-specific DNA binding / thioredoxin-dependent peroxiredoxin / thioredoxin peroxidase activity / Detoxification of Reactive Oxygen Species /  antioxidant activity / cysteine-type endopeptidase inhibitor activity involved in apoptotic process / peroxisomal matrix / positive regulation of collagen biosynthetic process / cell redox homeostasis / hydrogen peroxide catabolic process / TP53 Regulates Metabolic Genes / antioxidant activity / cysteine-type endopeptidase inhibitor activity involved in apoptotic process / peroxisomal matrix / positive regulation of collagen biosynthetic process / cell redox homeostasis / hydrogen peroxide catabolic process / TP53 Regulates Metabolic Genes /  peroxidase activity / cellular response to reactive oxygen species / peroxidase activity / cellular response to reactive oxygen species /  ペルオキシソーム / cellular response to oxidative stress / cytoplasmic vesicle / response to oxidative stress / ペルオキシソーム / cellular response to oxidative stress / cytoplasmic vesicle / response to oxidative stress /  ミトコンドリアマトリックス / ミトコンドリアマトリックス /  炎症 / 炎症 /  signaling receptor binding / intracellular membrane-bounded organelle / negative regulation of apoptotic process / perinuclear region of cytoplasm / signaling receptor binding / intracellular membrane-bounded organelle / negative regulation of apoptotic process / perinuclear region of cytoplasm /  ミトコンドリア / ミトコンドリア /  extracellular space / extracellular exosome / identical protein binding / extracellular space / extracellular exosome / identical protein binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単一同系置換 / 解像度: 1.95 Å 単一同系置換 / 解像度: 1.95 Å | ||||||
 データ登録者 データ登録者 | Declercq, J.P. / Evrard, C. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2001 ジャーナル: J.Mol.Biol. / 年: 2001タイトル: Crystal Structure of Human Peroxiredoxin 5, a Novel Type of Mammalian Peroxiredoxin at 1.5 A Resolution 著者: Declercq, J.P. / Evrard, C. / Clippe, A. / Stricht, D.V. / Bernard, A. / Knoops, B. #1: ジャーナル: J.Biol.Chem. / 年: 1999 タイトル: Cloning and Characterization of Aoeb166, a Novel Mammalian Antioxidant Enzyme of the Peroxiredoxin Family 著者: Knoops, B. / Clippe, A. / Bogard, C. / Arsalane, K. / Wattiez, R. / Hermans, C. / Duconseille, E. / Falmagne, P. / Bernard, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1h4o.cif.gz 1h4o.cif.gz | 266.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1h4o.ent.gz pdb1h4o.ent.gz | 218.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1h4o.json.gz 1h4o.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h4/1h4o https://data.pdbj.org/pub/pdb/validation_reports/h4/1h4o ftp://data.pdbj.org/pub/pdb/validation_reports/h4/1h4o ftp://data.pdbj.org/pub/pdb/validation_reports/h4/1h4o | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



