+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1eyx | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF R-PHYCOERYTHRIN AT 2.2 ANGSTROMS | ||||||
 要素 要素 | (R-PHYCOERYTHRIN) x 3 | ||||||
 キーワード キーワード |  PHOTOSYNTHESIS (光合成) / R-PHYCOERYTHRIN / MACROSEEDING / PHOTOSYNTHESIS (光合成) / R-PHYCOERYTHRIN / MACROSEEDING /  TWIN (双生児) / TWIN (双生児) /  PROTEIN STRUCTURE (タンパク質構造) / SEQUENCES / PHYCOBILIPROTEIN / PROTEIN STRUCTURE (タンパク質構造) / SEQUENCES / PHYCOBILIPROTEIN /  RED ALGAE (紅藻) RED ALGAE (紅藻) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Gracilaria chilensis (真核生物) Gracilaria chilensis (真核生物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.25 Å 分子置換 / 解像度: 2.25 Å | ||||||
 データ登録者 データ登録者 | Contreras-Martel, C. / Legrand, P. / Piras, C. / Vernede, X. / Martinez-Oyanedel, J. / Bunster, M. / Fontecilla-Camps, J.C. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2001 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2001タイトル: Crystallization and 2.2 A resolution structure of R-phycoerythrin from Gracilaria chilensis: a case of perfect hemihedral twinning. 著者: Contreras-Martel, C. / Martinez-Oyanedel, J. / Bunster, M. / Legrand, P. / Piras, C. / Vernede, X. / Fontecilla-Camps, J.C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1eyx.cif.gz 1eyx.cif.gz | 155.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1eyx.ent.gz pdb1eyx.ent.gz | 123.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1eyx.json.gz 1eyx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ey/1eyx https://data.pdbj.org/pub/pdb/validation_reports/ey/1eyx ftp://data.pdbj.org/pub/pdb/validation_reports/ey/1eyx ftp://data.pdbj.org/pub/pdb/validation_reports/ey/1eyx | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1liaS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 4分子 AKBL
| #1: タンパク質 | 分子量: 17770.857 Da / 分子数: 2 / 断片: ALPHA CHAIN / 由来タイプ: 天然 / 由来: (天然)  Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST 葉緑体 / 参照: UniProt: Q7SIG0 葉緑体 / 参照: UniProt: Q7SIG0#2: タンパク質 | 分子量: 18634.182 Da / 分子数: 2 / 断片: BETA CHAIN / 由来タイプ: 天然 / 由来: (天然)  Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST 葉緑体 / 参照: UniProt: Q7SIF9 葉緑体 / 参照: UniProt: Q7SIF9 |
|---|
-タンパク質・ペプチド , 1種, 2分子 GH
| #3: タンパク質・ペプチド | 分子量: 606.694 Da / 分子数: 2 / 断片: GAMMA CHAIN / 由来タイプ: 天然 / 由来: (天然)  Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST Gracilaria chilensis (真核生物) / Organelle: CHLOROPLAST 葉緑体 葉緑体 |
|---|
-非ポリマー , 5種, 122分子 
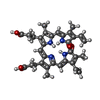

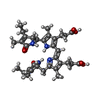





| #4: 化合物 |  硫酸塩 硫酸塩#5: 化合物 | ChemComp-BLA / | #6: 化合物 | ChemComp-CYC /  フィコシアノビリン フィコシアノビリン#7: 化合物 |  Phycourobilin Phycourobilin#8: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.88 Å3/Da / 溶媒含有率: 34.7 % 解説: A PERFECT HEMIHEDRAL TWINNED CRYSTAL. STANLEY DISTRIBUTION, 1.44. TWIN FRACTION, 0.48. | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: 200 MM HEPES, 500 MM SODIUM CHLORIDE, 50 MM POTASIUM CHLORIDE, 15 MM SODIUM AZIDE, 16% AMMONIUM SULPHATE, pH 7.0, VAPOR DIFFUSION, SITTING DROP, temperature 293K | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 294 K | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: BM14 / 波長: 0.9783 / ビームライン: BM14 / 波長: 0.9783 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1998年8月31日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9783 Å / 相対比: 1 : 0.9783 Å / 相対比: 1 |
| 反射 | 解像度: 2.16→42.55 Å / Num. obs: 38751 / % possible obs: 98.7 % / Observed criterion σ(I): 0 / 冗長度: 3.1 % / Rmerge(I) obs: 0.101 / Rsym value: 0.101 / Net I/σ(I): 7.3 |
| 反射 シェル | 解像度: 2.16→2.28 Å / 冗長度: 2 % / Rmerge(I) obs: 0.039 / Mean I/σ(I) obs: 2.8 / Rsym value: 0.039 / % possible all: 89.4 |
| 反射 | *PLUS Num. measured all: 123143 |
| 反射 シェル | *PLUS % possible obs: 89.4 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1LIA 解像度: 2.25→999 Å / Num. parameters: 2290 / Num. restraintsaints: 3005 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| Refine analyze | Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 5654 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.25→999 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor Rwork : 0.18 : 0.18 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj



