+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cwq | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | M INTERMEDIATE STRUCTURE OF THE WILD TYPE BACTERIORHODOPSIN IN COMBINATION WITH THE GROUND STATE STRUCTURE | ||||||||||||
 要素 要素 | BACTERIORHODOPSIN ("M" STATE INTERMEDIATE IN COMBINATION WITH GROUND STATE) | ||||||||||||
 キーワード キーワード |  ION TRANSPORT / PHOTO CYCLE INTERMEDIATE / 7-HELICAL MEMBRANE PROTEIN / ION TRANSPORT / PHOTO CYCLE INTERMEDIATE / 7-HELICAL MEMBRANE PROTEIN /  PROTON TRANSPORT (プロトンポンプ) / PROTON TRANSPORT (プロトンポンプ) /  RETINAL PROTEIN (レチナール) RETINAL PROTEIN (レチナール) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 photoreceptor activity / photoreceptor activity /  phototransduction / proton transmembrane transport / monoatomic ion channel activity / phototransduction / proton transmembrane transport / monoatomic ion channel activity /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.25 Å 分子置換 / 解像度: 2.25 Å | ||||||||||||
 データ登録者 データ登録者 | Sass, H.J. / Berendzen, J. / Neff, D. / Gessenich, R. / Ormos, P. / Bueldt, G. | ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2000 ジャーナル: Nature / 年: 2000タイトル: Structural alterations for proton translocation in the M state of wild-type bacteriorhodopsin. 著者: Sass, H.J. / Buldt, G. / Gessenich, R. / Hehn, D. / Neff, D. / Schlesinger, R. / Berendzen, J. / Ormos, P. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cwq.cif.gz 1cwq.cif.gz | 118.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cwq.ent.gz pdb1cwq.ent.gz | 93.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cwq.json.gz 1cwq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cw/1cwq https://data.pdbj.org/pub/pdb/validation_reports/cw/1cwq ftp://data.pdbj.org/pub/pdb/validation_reports/cw/1cwq ftp://data.pdbj.org/pub/pdb/validation_reports/cw/1cwq | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 26888.557 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: SCHIFF BASE LINKAGE BETWEEN LYS 216 (NZ) AND RET 601 (C15) DIETHER LIPID BILAYER 由来: (組換発現)   Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性)細胞内の位置: TRANSMEMBRANE  膜貫通型タンパク質 膜貫通型タンパク質発現宿主:   Halobacterium salinarum (好塩性) / 参照: UniProt: P02945 Halobacterium salinarum (好塩性) / 参照: UniProt: P02945 |
|---|
-非ポリマー , 6種, 198分子 
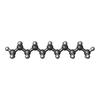
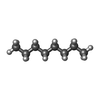
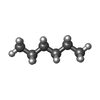







| #2: 化合物 |  レチナール レチナール#3: 化合物 | ChemComp-UND /  ウンデカン ウンデカン#4: 化合物 | ChemComp-OCT /  オクタン オクタン#5: 化合物 | ChemComp-HEX /  ヘキサン ヘキサン#6: 化合物 | ChemComp-TRD /  トリデカン トリデカン#7: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 構成要素の詳細 | THE ENTRY CONTAINS THE COORDINATES OF THE M INTERMEDIATE (CHAIN B + LIPID RESIDUES 1604-1624 AND ...THE ENTRY CONTAINS THE COORDINATE |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.25 Å3/Da / 解説: TWINNING OF 38% | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | pH: 5.6 / 詳細: CUBIC LIPID PHASE 2.5M PHOSPHATE, pH 5.60 | |||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ / pH: 5.6 / 手法: cubic lipid phase詳細: Landau, E.M., (1996) Proc.Natl.Acad.Sci.USA., 93, 14532. | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X8C / 波長: 1.214 / ビームライン: X8C / 波長: 1.214 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1998年11月9日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.214 Å / 相対比: 1 : 1.214 Å / 相対比: 1 |
| 反射 | 解像度: 2.25→20 Å / Num. obs: 10792 / % possible obs: 97.2 % / Observed criterion σ(I): 0 / 冗長度: 2 % / Biso Wilson estimate: 22.16 Å2 / Rsym value: 8.2 / Net I/σ(I): 12.05 |
| 反射 シェル | 解像度: 2.25→2.33 Å / 冗長度: 2 % / Mean I/σ(I) obs: 3.4 / Rsym value: 32.8 / % possible all: 99.4 |
| 反射 | *PLUS Rmerge(I) obs: 0.082 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.25→13 Å / 交差検証法: FREE R VALUE, MAXIMUM LIKELIHOOD / σ(F): 1 / 立体化学のターゲット値: ENGH & HUBER 分子置換 / 解像度: 2.25→13 Å / 交差検証法: FREE R VALUE, MAXIMUM LIKELIHOOD / σ(F): 1 / 立体化学のターゲット値: ENGH & HUBER詳細: THE TWIN RATIO OF 0.38 WAS CONSTANT DURING THE REFINEMENT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 138.6 Å2 / ksol: 0.264 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 22.24 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.25→13 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.25→2.33 Å / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 0.4 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 13 Å / % reflection Rfree: 5.3 % / Rfactor obs: 0.179 / Rfactor Rfree : 0.208 : 0.208 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.293 / Rfactor Rwork: 0.195 |
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj












