+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31984 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
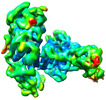
| タイトル | Cryo-EM structure of Machupo virus dimeric polymerase L | |||||||||
 マップデータ マップデータ | Cryo-EM structure of Machupo virus dimeric polymerase L | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-templated viral transcription / negative stranded viral RNA replication /  キャップスナッチング / キャップスナッチング /  virion component / host cell cytoplasm / virion component / host cell cytoplasm /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  hydrolase activity / hydrolase activity /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  nucleotide binding / nucleotide binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |  Machupo mammarenavirus (ウイルス) / Machupo mammarenavirus (ウイルス) /  Machupo virus (ウイルス) Machupo virus (ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.1 Å クライオ電子顕微鏡法 / 解像度: 5.1 Å | |||||||||
 データ登録者 データ登録者 | Zhang X / Ma J / Zhang S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of Machupo virus polymerase in complex with matrix protein Z. 著者: Jun Ma / Shuangyue Zhang / Xinzheng Zhang /  要旨: The Arenaviridae family includes several viruses that cause severe human hemorrhagic fevers with high mortality, with no effective countermeasures currently available. The arenavirus multi-domain L ...The Arenaviridae family includes several viruses that cause severe human hemorrhagic fevers with high mortality, with no effective countermeasures currently available. The arenavirus multi-domain L protein is involved in viral transcription and replication and represents a promising target for antiviral drugs. The arenavirus matrix protein Z is a small multi-functional protein that inhibits the activities of the L protein. Here we report the structure of Machupo virus L protein in complex with Z determined by cryo-electron microscopy. The Z protein acts as a staple and binds the L protein with 1:1 stoichiometry at the intersection between the PA-C-like region, RNA-dependent RNA polymerase and PB2-N-like region. Binding of the Z protein may lock the multiple domains of L into a fixed arrangement leading to loss of catalytic activity. These results further our understanding of the inhibitory mechanism of arenavirus replication machinery and provide a novel perspective to develop antiviral drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31984.map.gz emd_31984.map.gz | 1.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31984-v30.xml emd-31984-v30.xml emd-31984.xml emd-31984.xml | 11.4 KB 11.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31984.png emd_31984.png | 172.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31984 http://ftp.pdbj.org/pub/emdb/structures/EMD-31984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31984 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31984.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31984.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of Machupo virus dimeric polymerase L | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.64 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Machupo virus dimeric polymerase L
| 全体 | 名称: Machupo virus dimeric polymerase L |
|---|---|
| 要素 |
|
-超分子 #1: Machupo virus dimeric polymerase L
| 超分子 | 名称: Machupo virus dimeric polymerase L / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Machupo mammarenavirus (ウイルス) Machupo mammarenavirus (ウイルス) |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
|---|---|
| 由来(天然) | 生物種:  Machupo virus (ウイルス) Machupo virus (ウイルス) |
| 分子量 | 理論値: 253.456344 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDEYVQELKG LIRKHIPDRC EFAHQKVTFL SQVHPSPLLT EGFKLLSSLV ELESCEAHAC QANTDQRFVD VILSDNGILC PTLPKVIPD GFKLTGKTLI LLETFVRVNP DEFEKKWKAD MSKLLNLKHD LQKSGVTLVP IVDGRSNYNN RFVADWVIER M RWLLIEIL ...文字列: MDEYVQELKG LIRKHIPDRC EFAHQKVTFL SQVHPSPLLT EGFKLLSSLV ELESCEAHAC QANTDQRFVD VILSDNGILC PTLPKVIPD GFKLTGKTLI LLETFVRVNP DEFEKKWKAD MSKLLNLKHD LQKSGVTLVP IVDGRSNYNN RFVADWVIER M RWLLIEIL KASKSMLEID IEDQEYQRLI HSLSNVKNQS LGLENLEHLK RNSLDYDERL NESLFIGLKG DIRESTVREE LI KLKMWFK DEVFSKGLGK FKLTDRRELL ESLSSLGAHL DSDVSSCPFC NNKLMEIVYN VTFSSVERTD GAATVDQQFS TTH TNIEKH YLSVLSLCNK IKGLKVFNTR RNTLLFLDLI MVNLMVDISE SCQDAIESLR KSGLIVGQMV MLVNDRVLDI LEAI KLIRK KIGTNPNWVK NCSKILERSH PEIWLQLNTL IRQPDFNSLI SIAQYLVSDR PIMRYSVERG SDKICRHKLF QEMSS FEQM RLFKTLSSIS LSLINSMKTS FSSRLLVNER EFSKYFGNVR LRECYAQRFY LAESLVGFLF YQKTGERSRC YSVYLS DNG VMSEQGSFYC DPKRFFLPVF SDEVLAGMCE EMTSWLDFDT GLMNDTGPIL RLLVLAILCS PSKRNQTFLQ GLRYFLM AF ANQIHHIDLI SKLVVECKSS SEVVVQRLAV GLFIRLLGGE SDASSFFSRR FKYLLNVSYL CHLITKETPD RLTDQIKC F EKFIEPKVKF GCAVVNPSLN GKLTVDQEDI MINGLKKFFS KSLRDTEDVQ TPGVCKELLN YCVSLFNRGK LKVSGELKN NPFRPNITST ALDLSSNKSV VIPKLDELGN ILSTYDKEKL VSACVSSMAE RFKTKGRYNL DPESTDYLIL KNLTGLVSAG PKAKSSQEE LSLMYETLTE EQVESFNEIK YDVQVALAKM ADNSVNTRIK NLGRADNSVK NGNNPLDNLW SPFGVMKEIR A EVSLHEVK DFDPDVLPSD VYKELCDAVY KSSEKCNFFL EEVLDVCPLG LLLKNLTTSS YMEEEYFMCF KYLLIQGHFD QK LGSYEHK SRSRLGFTDE TLRLKDEVRL SIRESNSEAI ADKLDKSYFT NAALRNLCFY SEDSPTEFTS ISSNSGNLKF GLS YKEQVG SNRELYVGDL NTKLMTRLVE DFSEAVGNSM KYTCLNSEKE FERAICDMKM AVNNGDLSCS YDHSKWGPTM SPAL FLALL QMLELRTPVD RSKIDLDSVK SILKWHLHKV VEVPINVAEA YCIGKLKRSL GLMGCGSTSL SEEFFHQTMQ LSGQI PSHI MSVLDMGQGI LHNTSDLYGL ITEQFLCYAL DLLYDVIPVS YTSSDDQITL VKTPSLDIEG GSDAAEWLEM ICFHEF LSS KLNKFVSPKS VIGTFVAEFK SRFFVMGEET PLLTKFVSAA LHNVKCKTPT QLSETIDTIC DQCIANGVST KIVARIS KR VNQLIRYSGY GDTPFGAIED QDVKDWVDGS RGYRLQRKIE AIFYDDKETS FIRNCARKVF NDIKRGRIFE ENLINLIG R GGDEALTGFL QYAGCSEQEV NRVLNYRWVN LSSFGDLRLV LRTKLMTSRR VLEREEVPTL IKTLQSKLSR NFTKGVKKI LAESINKSAF QSSVASGFIG FCKSMGSKCV RDGKGGFLYI KEVYSGINVC ICEICALKPK IIYCNDSLNK VSQFSKPILW DYFSLVLTN ACELGEWVFS TVKEPQKPLV LNNQNFFWAV KPKVVRQIED QLGMNHVLQS IRRNYPVLFD EHLAPFMNDL Q VSRTMDSG RLKFLDVCIA LDMMNENLGI ISHLLKTRDN SVYIVKQSDC ALAHIRQSSY TDWELGLSPQ QICTNFKTQL VL SSMVNPL VLSTSCLKSF FWFNEVLELE DDSQIELAEL TDFALMVKNQ NVSRAMFVED IAMGYVVSNF EGVRISLSNV MVD GVQLPP KEKAPDVGVL FGLKAENVIV GLVVQIDHVR MSTKFKLRRK MVYSFSLECT MDVGDIQNKE VILKVVAVDQ SVSG SGGNH MLLDGVPVIA SLPLFTGQAS FDLAAMLIES NLAGSNDNFL MSNVTLDLGG FSPELSDKYS YRLSGPENQE DPLVL KDGA FYVGGERLST YKVELTGDLV VKALGALEDD EGVVSMLHQL WPYLKATSQV ILFQQEDFTI VHDLYKIQLT KSIESF GEW IEFTNFKVAY SKSLKELVIS DTQGSFRLKG VMCRPLANTL QVEDIEWSHP QFEKGGGSGG GSGGSSAWSH PQFEK |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 5.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 52947 |
 ムービー
ムービー コントローラー
コントローラー