+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of Inactive NTSR1 Bound to SR48692 and Nb6 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dynorphin receptor activity / response to acrylamide / regulation of saliva secretion / negative regulation of luteinizing hormone secretion / sensory perception of temperature stimulus / positive regulation of eating behavior / G protein-coupled neurotensin receptor activity / adenylate cyclase-inhibiting opioid receptor signaling pathway / inositol phosphate catabolic process / G protein-coupled opioid receptor activity ...dynorphin receptor activity / response to acrylamide / regulation of saliva secretion / negative regulation of luteinizing hormone secretion / sensory perception of temperature stimulus / positive regulation of eating behavior / G protein-coupled neurotensin receptor activity / adenylate cyclase-inhibiting opioid receptor signaling pathway / inositol phosphate catabolic process / G protein-coupled opioid receptor activity / symmetric synapse / G protein-coupled opioid receptor signaling pathway / D-aspartate import across plasma membrane / positive regulation of gamma-aminobutyric acid secretion /  conditioned place preference / positive regulation of dopamine secretion / conditioned place preference / positive regulation of dopamine secretion /  知覚 / L-glutamate import across plasma membrane / positive regulation of potassium ion transmembrane transport / positive regulation of arachidonic acid secretion / regulation of respiratory gaseous exchange / positive regulation of inhibitory postsynaptic potential / 知覚 / L-glutamate import across plasma membrane / positive regulation of potassium ion transmembrane transport / positive regulation of arachidonic acid secretion / regulation of respiratory gaseous exchange / positive regulation of inhibitory postsynaptic potential /  maternal behavior / maternal behavior /  neuropeptide binding / neuropeptide binding /  receptor serine/threonine kinase binding / negative regulation of systemic arterial blood pressure / negative regulation of release of sequestered calcium ion into cytosol / positive regulation of glutamate secretion / positive regulation of p38MAPK cascade / positive regulation of inositol phosphate biosynthetic process / receptor serine/threonine kinase binding / negative regulation of systemic arterial blood pressure / negative regulation of release of sequestered calcium ion into cytosol / positive regulation of glutamate secretion / positive regulation of p38MAPK cascade / positive regulation of inositol phosphate biosynthetic process /  temperature homeostasis / temperature homeostasis /  eating behavior / response to lipid / eating behavior / response to lipid /  regulation of membrane depolarization / behavioral response to cocaine / detection of temperature stimulus involved in sensory perception of pain / regulation of membrane depolarization / behavioral response to cocaine / detection of temperature stimulus involved in sensory perception of pain /  estrous cycle / neuropeptide signaling pathway / axon terminus / MECP2 regulates neuronal receptors and channels / sensory perception of pain / estrous cycle / neuropeptide signaling pathway / axon terminus / MECP2 regulates neuronal receptors and channels / sensory perception of pain /  横行小管 / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / Peptide ligand-binding receptors / adult locomotory behavior / positive regulation of release of sequestered calcium ion into cytosol / dendritic shaft / 横行小管 / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / Peptide ligand-binding receptors / adult locomotory behavior / positive regulation of release of sequestered calcium ion into cytosol / dendritic shaft /  学習 / 学習 /  筋小胞体 / locomotory behavior / G protein-coupled receptor activity / cellular response to glucose stimulus / response to nicotine / response to insulin / 筋小胞体 / locomotory behavior / G protein-coupled receptor activity / cellular response to glucose stimulus / response to nicotine / response to insulin /  terminal bouton / cytoplasmic side of plasma membrane / synaptic vesicle membrane / response to estrogen / phospholipase C-activating G protein-coupled receptor signaling pathway / terminal bouton / cytoplasmic side of plasma membrane / synaptic vesicle membrane / response to estrogen / phospholipase C-activating G protein-coupled receptor signaling pathway /  presynaptic membrane / G alpha (i) signalling events / G alpha (q) signalling events / chemical synaptic transmission / presynaptic membrane / G alpha (i) signalling events / G alpha (q) signalling events / chemical synaptic transmission /  postsynaptic membrane / postsynaptic membrane /  perikaryon / response to ethanol / defense response to virus / cellular response to lipopolysaccharide / perikaryon / response to ethanol / defense response to virus / cellular response to lipopolysaccharide /  樹状突起スパイン / neuron projection / 樹状突起スパイン / neuron projection /  免疫応答 / 免疫応答 /  脂質ラフト / positive regulation of apoptotic process / G protein-coupled receptor signaling pathway / 脂質ラフト / positive regulation of apoptotic process / G protein-coupled receptor signaling pathway /  樹状突起 / protein-containing complex binding / positive regulation of gene expression / negative regulation of apoptotic process / 樹状突起 / protein-containing complex binding / positive regulation of gene expression / negative regulation of apoptotic process /  ゴルジ体 / ゴルジ体 /  細胞膜 / 細胞膜 /  小胞体 / 小胞体 /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Lama glama (ラマ) Lama glama (ラマ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.4 Å クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Robertson MJ / Skiniotis G | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Structure determination of inactive-state GPCRs with a universal nanobody. 著者: Michael J Robertson / Makaía M Papasergi-Scott / Feng He / Alpay B Seven / Justin G Meyerowitz / Ouliana Panova / Maria Claudia Peroto / Tao Che / Georgios Skiniotis /  要旨: Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite ...Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite representing one of the largest classes of therapeutic targets, most inactive-state G protein-coupled receptors (GPCRs) have remained inaccessible for cryo-EM because their small size and membrane-embedded nature impedes projection alignment for high-resolution map reconstructions. Here we demonstrate that the same single-chain camelid antibody (nanobody) recognizing a grafted intracellular loop can be used to obtain cryo-EM structures of inactive-state GPCRs at resolutions comparable or better than those obtained by X-ray crystallography. Using this approach, we obtained structures of neurotensin 1 receptor bound to antagonist SR48692, μ-opioid receptor bound to alvimopan, apo somatostatin receptor 2 and histamine receptor 2 bound to famotidine. We expect this rapid, straightforward approach to facilitate the broad exploration of GPCR inactive states without the need for extensive engineering and crystallization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26589.map.gz emd_26589.map.gz | 118.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26589-v30.xml emd-26589-v30.xml emd-26589.xml emd-26589.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26589.png emd_26589.png | 102.4 KB | ||
| その他 |  emd_26589_additional_1.map.gz emd_26589_additional_1.map.gz emd_26589_half_map_1.map.gz emd_26589_half_map_1.map.gz emd_26589_half_map_2.map.gz emd_26589_half_map_2.map.gz | 1.5 MB 115.9 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26589 http://ftp.pdbj.org/pub/emdb/structures/EMD-26589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26589 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26589.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26589.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.8677 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_26589_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26589_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26589_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of NTSR1 and Nb6
| 全体 | 名称: Complex of NTSR1 and Nb6 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of NTSR1 and Nb6
| 超分子 | 名称: Complex of NTSR1 and Nb6 / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: NTSR1
| 超分子 | 名称: NTSR1 / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Nb6
| 超分子 | 名称: Nb6 / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
-分子 #1: Neurotensin receptor 1
| 分子 | 名称: Neurotensin receptor 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.084391 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: DYKDDDDAMG QPGNGSAFLL APNRSHAPDH DVENLYFQGQ RAQAGLEEAL LAPGFGNASG NASERVLAAP SSELDVNTDI YSKVLVTAV YLALFVVGTV GNTVTLFTLA RKKSLQSLQS TVHYHLGSLA LSDLLTLLLA MPVELYNFIW VHHPWAFGDA G CRGYYFLR ...文字列: DYKDDDDAMG QPGNGSAFLL APNRSHAPDH DVENLYFQGQ RAQAGLEEAL LAPGFGNASG NASERVLAAP SSELDVNTDI YSKVLVTAV YLALFVVGTV GNTVTLFTLA RKKSLQSLQS TVHYHLGSLA LSDLLTLLLA MPVELYNFIW VHHPWAFGDA G CRGYYFLR DACTYATALN VASLSVERYL AICHPFKAKT LMSRSRTKKF ISAIWLASAL LAVPMLFTMG EQNRSADGQH AG GLVCTPT IHTATVKVVI QVNTFMSFIF PMVVISVLYT LMILRLKSVR LLSGSREKDR NLRRITRLVL AVVIAFVVCW LPY HVRRLM FCYISDEQWT PFLYDFYHYF YMVTNALFYV SSTINPILYN LVSANFRHIF LATLACLCPV WRRRRKRPAF SRKA DSVSS NHTLSSNATR ETLY |
-分子 #2: Nanobody 6
| 分子 | 名称: Nanobody 6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
| 分子量 | 理論値: 14.730255 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAQVQLQESG GGLVQAGESL RLSCAASGTI FRLYDMGWYR RVSGNQRELV ASITSGGSTK YGDSVKGRFT ISRDNAKNTV YLQMSSLKP EDTAVYYCNA EYRTGIWEEL LDGWGQGTQV TVSSHHHHHH EPEA |
-分子 #3: 2-[[1-(7-chloranylquinolin-4-yl)-5-(2,6-dimethoxyphenyl)pyrazol-3...
| 分子 | 名称: 2-[[1-(7-chloranylquinolin-4-yl)-5-(2,6-dimethoxyphenyl)pyrazol-3-yl]carbonylamino]adamantane-2-carboxylic acid タイプ: ligand / ID: 3 / コピー数: 1 / 式: Q6Q |
|---|---|
| 分子量 | 理論値: 587.065 Da |
| Chemical component information | 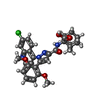 ChemComp-Q6Q: |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 13 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.82 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: OTHER |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 372987 |
 ムービー
ムービー コントローラー
コントローラー






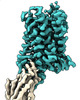

















 Z
Z Y
Y X
X