+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25377 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of mouse agonist ML-SA1-bound TRPML1 channel at 2.32 Angstrom resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transferrin endocytosis and recycling / positive regulation of lysosome organization / calcium ion export / intracellularly phosphatidylinositol-3,5-bisphosphate-gated monatomic cation channel activity / phagosome maturation / NAADP-sensitive calcium-release channel activity / cellular response to pH /  TRPチャネル / endosomal transport / intracellular vesicle ...Transferrin endocytosis and recycling / positive regulation of lysosome organization / calcium ion export / intracellularly phosphatidylinositol-3,5-bisphosphate-gated monatomic cation channel activity / phagosome maturation / NAADP-sensitive calcium-release channel activity / cellular response to pH / TRPチャネル / endosomal transport / intracellular vesicle ...Transferrin endocytosis and recycling / positive regulation of lysosome organization / calcium ion export / intracellularly phosphatidylinositol-3,5-bisphosphate-gated monatomic cation channel activity / phagosome maturation / NAADP-sensitive calcium-release channel activity / cellular response to pH /  TRPチャネル / endosomal transport / intracellular vesicle / monoatomic cation transmembrane transport / autophagosome maturation / phagocytic cup / monoatomic cation channel activity / release of sequestered calcium ion into cytosol / cellular response to calcium ion / cell projection / phagocytic vesicle membrane / late endosome / late endosome membrane / protein homotetramerization / TRPチャネル / endosomal transport / intracellular vesicle / monoatomic cation transmembrane transport / autophagosome maturation / phagocytic cup / monoatomic cation channel activity / release of sequestered calcium ion into cytosol / cellular response to calcium ion / cell projection / phagocytic vesicle membrane / late endosome / late endosome membrane / protein homotetramerization /  獲得免疫系 / 獲得免疫系 /  リソソーム / リソソーム /  receptor complex / lysosomal membrane / receptor complex / lysosomal membrane /  lipid binding / lipid binding /  ゴルジ体 / ゴルジ体 /  核質 / 核質 /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.32 Å クライオ電子顕微鏡法 / 解像度: 2.32 Å | |||||||||
 データ登録者 データ登録者 | Gan N / Han Y / Jiang Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structural mechanism of allosteric activation of TRPML1 by PI(3,5)P and rapamycin. 著者: Ninghai Gan / Yan Han / Weizhong Zeng / Yan Wang / Jing Xue / Youxing Jiang /  要旨: Transient receptor potential mucolipin 1 (TRPML1) is a Ca-permeable, nonselective cation channel ubiquitously expressed in the endolysosomes of mammalian cells and its loss-of-function mutations are ...Transient receptor potential mucolipin 1 (TRPML1) is a Ca-permeable, nonselective cation channel ubiquitously expressed in the endolysosomes of mammalian cells and its loss-of-function mutations are the direct cause of type IV mucolipidosis (MLIV), an autosomal recessive lysosomal storage disease. TRPML1 is a ligand-gated channel that can be activated by phosphatidylinositol 3,5-bisphosphate [PI(3,5)P] as well as some synthetic small-molecule agonists. Recently, rapamycin has also been shown to directly bind and activate TRPML1. Interestingly, both PI(3,5)P and rapamycin have low efficacy in channel activation individually but together they work cooperatively and activate the channel with high potency. To reveal the structural basis underlying the synergistic activation of TRPML1 by PI(3,5)P and rapamycin, we determined the high-resolution cryoelectron microscopy (cryo-EM) structures of the mouse TRPML1 channel in various states, including apo closed, PI(3,5)P-bound closed, and PI(3,5)P/temsirolimus (a rapamycin analog)-bound open states. These structures, combined with electrophysiology, elucidate the molecular details of ligand binding and provide structural insight into how the TRPML1 channel integrates two distantly bound ligand stimuli and facilitates channel opening. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25377.map.gz emd_25377.map.gz | 84.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25377-v30.xml emd-25377-v30.xml emd-25377.xml emd-25377.xml | 11 KB 11 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25377.png emd_25377.png | 50 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25377 http://ftp.pdbj.org/pub/emdb/structures/EMD-25377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25377 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25377.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25377.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mouse Transient receptor potential-mucolipin 1 apo closed state
| 全体 | 名称: Mouse Transient receptor potential-mucolipin 1 apo closed state |
|---|---|
| 要素 |
|
-超分子 #1: Mouse Transient receptor potential-mucolipin 1 apo closed state
| 超分子 | 名称: Mouse Transient receptor potential-mucolipin 1 apo closed state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Mucolipin-1
| 分子 | 名称: Mucolipin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 65.573617 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATPAGRRAS ETERLLTPNP GYGTQVGTSP APTTPTEEED LRRRLKYFFM SPCDKFRAKG RKPCKLMLQV VKILVVTVQL ILFGLSNQL VVTFREENTI AFRHLFLLGY SDGSDDTFAA YTQEQLYQAI FYAVDQYLIL PEISLGRYAY VRGGGGPWAN G SALALCQR ...文字列: MATPAGRRAS ETERLLTPNP GYGTQVGTSP APTTPTEEED LRRRLKYFFM SPCDKFRAKG RKPCKLMLQV VKILVVTVQL ILFGLSNQL VVTFREENTI AFRHLFLLGY SDGSDDTFAA YTQEQLYQAI FYAVDQYLIL PEISLGRYAY VRGGGGPWAN G SALALCQR YYHRGHVDPA NDTFDIDPRV VTDCIQVDPP DRPPDIPSED LDFLDGSASY KNLTLKFHKL INVTIHFQLK TI NLQSLIN NEIPDCYTFS ILITFDNKAH SGRIPIRLET KTHIQECKHP SVSRHGDNSF RLLFDVVVIL TCSLSFLLCA RSL LRGFLL QNEFVVFMWR RRGREISLWE RLEFVNGWYI LLVTSDVLTI SGTVMKIGIE AKNLASYDVC SILLGTSTLL VWVG VIRYL TFFHKYNILI ATLRVALPSV MRFCCCVAVI YLGYCFCGWI VLGPYHVKFR SLSMVSECLF SLINGDDMFV TFAAM QAQQ GHSSLVWLFS QLYLYSFISL FIYMVLSLFI ALITGAYDTI KHPGGTGTEK SELQAYIEQC QDSPTSGKFR RGSGSA CSL FCCCGRDSPE DHSLLVN |
-分子 #3: 2-{2-oxo-2-[(4S)-2,2,4-trimethyl-3,4-dihydroquinolin-1(2H)-yl]eth...
| 分子 | 名称: 2-{2-oxo-2-[(4S)-2,2,4-trimethyl-3,4-dihydroquinolin-1(2H)-yl]ethyl}-1H-isoindole-1,3(2H)-dione タイプ: ligand / ID: 3 / コピー数: 4 / 式: AQV |
|---|---|
| 分子量 | 理論値: 362.422 Da |
| Chemical component information | 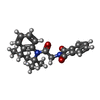 ChemComp-AQV: |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 12 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.9 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
|---|---|
| 最終 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.32 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 41980 |
 ムービー
ムービー コントローラー
コントローラー