+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13173 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Proton-powered peptide transporter SbmA in lipid nanodisc | ||||||||||||||||||||||||
 マップデータ マップデータ | SbmA peptide transporter in nanodisc | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報secondary active transmembrane transporter activity / microcin transmembrane transporter activity / microcin B17 transport / microcin transport / : / toxin transmembrane transporter activity / peptide transport / peptide transmembrane transporter activity / plasma membrane => GO:0005886 / ATPase-coupled transmembrane transporter activity ...secondary active transmembrane transporter activity / microcin transmembrane transporter activity / microcin B17 transport / microcin transport / : / toxin transmembrane transporter activity / peptide transport / peptide transmembrane transporter activity / plasma membrane => GO:0005886 / ATPase-coupled transmembrane transporter activity /  protein transport / response to antibiotic / protein transport / response to antibiotic /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||||||||||||||||||||
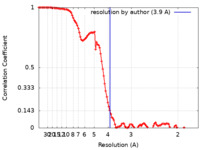
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Ghilarov D / Beis K | ||||||||||||||||||||||||
| 資金援助 |  ポーランド, ポーランド,  英国, 英国,  日本, 日本,  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Molecular mechanism of SbmA, a promiscuous transporter exploited by antimicrobial peptides. 著者: Dmitry Ghilarov / Satomi Inaba-Inoue / Piotr Stepien / Feng Qu / Elizabeth Michalczyk / Zuzanna Pakosz / Norimichi Nomura / Satoshi Ogasawara / Graham Charles Walker / Sylvie Rebuffat / So ...著者: Dmitry Ghilarov / Satomi Inaba-Inoue / Piotr Stepien / Feng Qu / Elizabeth Michalczyk / Zuzanna Pakosz / Norimichi Nomura / Satoshi Ogasawara / Graham Charles Walker / Sylvie Rebuffat / So Iwata / Jonathan Gardiner Heddle / Konstantinos Beis /      要旨: Antibiotic metabolites and antimicrobial peptides mediate competition between bacterial species. Many of them hijack inner and outer membrane proteins to enter cells. Sensitivity of enteric bacteria ...Antibiotic metabolites and antimicrobial peptides mediate competition between bacterial species. Many of them hijack inner and outer membrane proteins to enter cells. Sensitivity of enteric bacteria to multiple peptide antibiotics is controlled by the single inner membrane protein SbmA. To establish the molecular mechanism of peptide transport by SbmA and related BacA, we determined their cryo–electron microscopy structures at 3.2 and 6 Å local resolution, respectively. The structures show a previously unknown fold, defining a new class of secondary transporters named SbmA-like peptide transporters. The core domain includes conserved glutamates, which provide a pathway for proton translocation, powering transport. The structures show an outward-open conformation with a large cavity that can accommodate diverse substrates. We propose a molecular mechanism for antibacterial peptide uptake paving the way for creation of narrow-targeted therapeutics. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13173.map.gz emd_13173.map.gz | 3.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13173-v30.xml emd-13173-v30.xml emd-13173.xml emd-13173.xml | 13.2 KB 13.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13173_fsc.xml emd_13173_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13173.png emd_13173.png | 121 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13173 http://ftp.pdbj.org/pub/emdb/structures/EMD-13173 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13173 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13173 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7p34C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10763 (タイトル: Proton-powered peptide transporter SbmA in lipid nanodisc EMPIAR-10763 (タイトル: Proton-powered peptide transporter SbmA in lipid nanodiscData size: 2.7 TB Data #1: Unaligned multi-frame movies of SbmA in lipid nanodisc [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13173.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13173.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SbmA peptide transporter in nanodisc | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Proton-driven peptide transporter SbmA in lipid nanodisc
| 全体 | 名称: Proton-driven peptide transporter SbmA in lipid nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: Proton-driven peptide transporter SbmA in lipid nanodisc
| 超分子 | 名称: Proton-driven peptide transporter SbmA in lipid nanodisc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: C43 Escherichia coli (大腸菌) / 組換株: C43 |
-分子 #1: SbmA
| 分子 | 名称: SbmA / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MFKSFFPKPG TFFLSAFVW A LIAVIFWQ AG GGDWVAR ITG ASGQIP ISAA RFWSL DFLIF YAYY IVCVGL FAL FWFIYSP HR WQYWSILG T ALIIFVTWF LVEVGVAVNA WYAPFYDLI Q TALSSPHK VT IEQFYRE VGV FLGIAL IAVV ISVLN ...文字列: MFKSFFPKPG TFFLSAFVW A LIAVIFWQ AG GGDWVAR ITG ASGQIP ISAA RFWSL DFLIF YAYY IVCVGL FAL FWFIYSP HR WQYWSILG T ALIIFVTWF LVEVGVAVNA WYAPFYDLI Q TALSSPHK VT IEQFYRE VGV FLGIAL IAVV ISVLN NFFVS HYVF RWRTAM NEY YMANWQQ LR HIEGAAQR V QEDTMRFAS TLENMGVSFI NAIMTLIAF L PVLVTLSA HV PELPIIG HIP YGLVIA AIVW SLMGT GLLAV VGIK LPGLEF KNQ RVEAAYR KE LVYGEDDA T RATPPTVRE LFSAVRKNYF RLYFHYMYF N IARILYLQ VD NVFGLFL LFP SIVAGT ITLG LMTQI TNVFG QVRG AFQYLI NSW TTLVELM SI YKRLRSFE H ELDGDKIQE VTHTLS |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial model generated by Buccaneer and refined by PHENIX |
|---|
 ムービー
ムービー コントローラー
コントローラー