+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10799 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a BcsB pentamer in the context of an assembled Bcs macrocomplex | ||||||||||||||||||
 マップデータ マップデータ | composite map made from two experimental sharpened maps of the same sample data | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellulose biosynthetic process / UDP-glucose metabolic process / membrane => GO:0016020 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.0 Å クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zouhir S / Krasteva PV | ||||||||||||||||||
| 資金援助 | 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Architecture and regulation of an enterobacterial cellulose secretion system. 著者: Wiem Abidi / Samira Zouhir / Meryem Caleechurn / Stéphane Roche / Petya Violinova Krasteva /  要旨: Many free-living and pathogenic enterobacteria secrete biofilm-promoting cellulose using a multicomponent, envelope-embedded Bcs secretion system under the control of intracellular second messenger c- ...Many free-living and pathogenic enterobacteria secrete biofilm-promoting cellulose using a multicomponent, envelope-embedded Bcs secretion system under the control of intracellular second messenger c-di-GMP. The molecular understanding of system assembly and cellulose secretion has been largely limited to the crystallographic studies of a distantly homologous BcsAB synthase tandem and a low-resolution reconstruction of an assembled macrocomplex that encompasses most of the inner membrane and cytosolic subunits and features an atypical layered architecture. Here, we present cryo-EM structures of the assembled Bcs macrocomplex, as well as multiple crystallographic snapshots of regulatory Bcs subcomplexes. The structural and functional data uncover the mechanism of asymmetric secretion system assembly and periplasmic crown polymerization and reveal unexpected subunit stoichiometry, multisite c-di-GMP recognition, and ATP-dependent regulation. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10799.map.gz emd_10799.map.gz | 215.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10799-v30.xml emd-10799-v30.xml emd-10799.xml emd-10799.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10799.png emd_10799.png | 195.9 KB | ||
| その他 |  emd_10799_additional_1.map.gz emd_10799_additional_1.map.gz emd_10799_additional_2.map.gz emd_10799_additional_2.map.gz emd_10799_additional_3.map.gz emd_10799_additional_3.map.gz emd_10799_additional_4.map.gz emd_10799_additional_4.map.gz | 215.5 MB 218.5 MB 126.4 MB 126.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10799 http://ftp.pdbj.org/pub/emdb/structures/EMD-10799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10799 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10799.map.gz / 形式: CCP4 / 大きさ: 262.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10799.map.gz / 形式: CCP4 / 大きさ: 262.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | composite map made from two experimental sharpened maps of the same sample data | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05248 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: 2.9A locally refined and sharpened map used for...
| ファイル | emd_10799_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 2.9A locally refined and sharpened map used for the main composite map | ||||||||||||
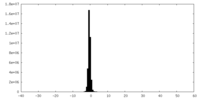
| 投影像・断面図 |
| ||||||||||||
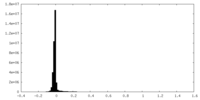
| 密度ヒストグラム |
-追加マップ: 3.1A average resolution sharpened map used for the main composite map
| ファイル | emd_10799_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3.1A average resolution sharpened map used for the main composite map | ||||||||||||
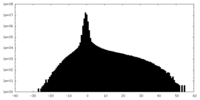
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Non sharpened map
| ファイル | emd_10799_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non sharpened map | ||||||||||||
| 投影像・断面図 |
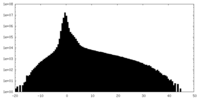
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Non sharpened map
| ファイル | emd_10799_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non sharpened map | ||||||||||||
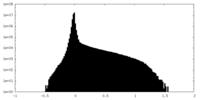
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Co-expression of Bcs subunits R, Q, A, B, E, F and G lead to the ...
| 全体 | 名称: Co-expression of Bcs subunits R, Q, A, B, E, F and G lead to the purification of a membrane complex assembly |
|---|---|
| 要素 |
|
-超分子 #1: Co-expression of Bcs subunits R, Q, A, B, E, F and G lead to the ...
| 超分子 | 名称: Co-expression of Bcs subunits R, Q, A, B, E, F and G lead to the purification of a membrane complex assembly タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: periplasm Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: periplasm |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCDF-duet Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCDF-duet |
| 分子量 | 理論値: 734 KDa |
-超分子 #2: Homopentameric assembly of the BcsB protein
| 超分子 | 名称: Homopentameric assembly of the BcsB protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: periplasm Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: periplasm |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCDF-duet Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCDF-duet |
-分子 #1: Bacterial cellulose secretion regulator BcsB
| 分子 | 名称: Bacterial cellulose secretion regulator BcsB / タイプ: protein_or_peptide / ID: 1 / 詳細: Bacterial cellulose synthesis subunit B / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: 1094 Escherichia coli (大腸菌) / 株: 1094 |
| 分子量 | 理論値: 86.184383 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MKRKLFWICA VAMGMSAFPS FMTQATPATQ PLINAEPAVA AQTEQNPQVG QVMPGVQGAD APVVAQNGPS RDVKLTFAQI APPPGSMVL RGINPNGSIE FGMRSDEVVT KAMLNLEYTP SPSLLPVQSQ LKVYLNDELM GVLPVTKEQL GKKTLAQMPI N PLFITDFN ...文字列: MKRKLFWICA VAMGMSAFPS FMTQATPATQ PLINAEPAVA AQTEQNPQVG QVMPGVQGAD APVVAQNGPS RDVKLTFAQI APPPGSMVL RGINPNGSIE FGMRSDEVVT KAMLNLEYTP SPSLLPVQSQ LKVYLNDELM GVLPVTKEQL GKKTLAQMPI N PLFITDFN RVRLEFVGHY QDVCENPAST TLWLDVGRSS GLDLTYQTLN VKNDLSHFPV PFFDPRDNRT NTLPMVFAGA PD VGLQQAS AIVASWFGSR SGWRGQNFPV LYNQLPDRNA IVFATNDKRP DFLRDHPAVK APVIEMINHP QNPYVKLLVV FGR DDKDLL QAAKGIAQGN ILFRGESVVV NEVKPLLPRK PYDAPNWVRT DRPVTFGELK TYEEQLQSSG LEPAAINVSL NLPP DLYLM RSTGIDMDIN YRYTMPPVKD SSRMDISLNN QFLQSFNLSS KQEANRLLLR IPVLQGLLDG KTDVSIPALK LGATN QLRF DFEYMNPMPG GSVDNCITFQ PVQNHVVIGD DSTIDFSKYY HFIPMPDLRA FANAGFPFSR MADLSQTITV MPKAPN EAQ METLLNTVGF IGAQTGFPAI NLTVTDDGST IQGKDADIMI IGGIPDKLKD DKQIDLLVQA TESWVKTPMR QTPFPGI VP DESDRAAETR STLTSSGAMA AVIGFQSPYN DQRSVIALLA DSPRGYEMLN DAVNDSGKRA TMFGSVAVIR ESGINSLR V GDVYYVGHLP WFERLWYALA NHPILLAVLA AISVILLAWV LWRLLRIISR RRLNPDNE |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.75 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.75 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: Gctf / 詳細: Gctf through the cryoSPARC v2 interface. |
|---|---|
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: Ab Initio model generation in cryosparc V2 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. V2) 詳細: All 2D classification and particle curation was performed in cryoSPARC v2 |
| 最終 3次元分類 | クラス数: 1 / ソフトウェア - 名称: cryoSPARC (ver. V2) 詳細: Final refinement using non-uniform refinement in cryoSPARC v2, followed by density segmation and mask creation in UCSF Chimera, particle subtraction for removal of noisy/unresolved regions ...詳細: Final refinement using non-uniform refinement in cryoSPARC v2, followed by density segmation and mask creation in UCSF Chimera, particle subtraction for removal of noisy/unresolved regions and local refinement of a BcsB pentamer in cryoSPARC v2. |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. V2) |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. v2) / 使用した粒子像数: 576455 |
| 詳細 | A total of 9,129 movies recorded on the CM01 Titan Krios transmission electron microscope (Thermo Fisher Scientific) at the ESRF Grenoble operated at 300 kV and equipped with a Gatan K2 Summit direct electron detector and a GIF Quantum LS imaging filter |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6yg8: |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X