+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDE52 |
|---|---|
 試料 試料 | Ribonuclease E from Escherichia coli リボヌクレアーゼE リボヌクレアーゼE
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 regulation of RNA helicase activity / rRNA 5'-end processing / regulation of RNA helicase activity / rRNA 5'-end processing /  リボヌクレアーゼE / リボヌクレアーゼE /  ribonuclease E activity / bacterial degradosome / ribonuclease E activity / bacterial degradosome /  endoribonuclease complex / DEAD/H-box RNA helicase binding / 7S RNA binding / RNA catabolic process / tRNA processing ... endoribonuclease complex / DEAD/H-box RNA helicase binding / 7S RNA binding / RNA catabolic process / tRNA processing ... regulation of RNA helicase activity / rRNA 5'-end processing / regulation of RNA helicase activity / rRNA 5'-end processing /  リボヌクレアーゼE / リボヌクレアーゼE /  ribonuclease E activity / bacterial degradosome / ribonuclease E activity / bacterial degradosome /  endoribonuclease complex / DEAD/H-box RNA helicase binding / 7S RNA binding / RNA catabolic process / tRNA processing / mRNA catabolic process / RNA nuclease activity / endoribonuclease complex / DEAD/H-box RNA helicase binding / 7S RNA binding / RNA catabolic process / tRNA processing / mRNA catabolic process / RNA nuclease activity /  転写後修飾 / RNA endonuclease activity / cytoplasmic side of plasma membrane / rRNA processing / protein complex oligomerization / protein homotetramerization / 転写後修飾 / RNA endonuclease activity / cytoplasmic side of plasma membrane / rRNA processing / protein complex oligomerization / protein homotetramerization /  tRNA binding / tRNA binding /  rRNA binding / molecular adaptor activity / magnesium ion binding / rRNA binding / molecular adaptor activity / magnesium ion binding /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞質 細胞質類似検索 - 分子機能 |
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
 引用 引用 |  日付: 2019 Dec 日付: 2019 Decタイトル: A structural and biochemical comparison of Ribonuclease E homologues from pathogenic bacteria highlights species-specific properties 著者: Mardle C / Shakespeare T / Butt L / Goddard L / Gowers D / Atkins H / Vincent H |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #2185 |  タイプ: dummy / ソフトウェア: (5) / ダミー原子の半径: 5.00 A / 対称性  : P222 / カイ2乗値: 1.054 / P-value: 0.000144 : P222 / カイ2乗値: 1.054 / P-value: 0.000144 Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|
- 試料
試料
 試料 試料 | 名称: Ribonuclease E from Escherichia coli リボヌクレアーゼE リボヌクレアーゼE試料濃度: 6.58 mg/ml |
|---|---|
| バッファ | 名称: 10 mM DTT, 10 mM MgCl2, 0.5 M NaCl, 20 mM Tris / pH: 8 |
| 要素 #1203 | 名称: RNase E / タイプ: protein / 記述: Endoribonuclease E / 分子量: 61.84 / 分子数: 4 / 由来: Escherichia coli / 参照: UniProt: P21513 配列: HHHHHHHHHH SSGHIEGRHM KRMLINATQQ EELRVALVDG QRLYDLDIES PGHEQKKANI YKGKITRIEP SLEAAFVDYG AERHGFLPLK EIAREYFPAN YSAHGRPNIK DVLREGQEVI VQIDKEERGN KGAALTTFIS LAGSYLVLMP NNPRAGGISR RIEGDDRTEL ...配列: HHHHHHHHHH SSGHIEGRHM KRMLINATQQ EELRVALVDG QRLYDLDIES PGHEQKKANI YKGKITRIEP SLEAAFVDYG AERHGFLPLK EIAREYFPAN YSAHGRPNIK DVLREGQEVI VQIDKEERGN KGAALTTFIS LAGSYLVLMP NNPRAGGISR RIEGDDRTEL KEALASLELP EGMGLIVRTA GVGKSAEALQ WDLSFRLKHW EAIKKAAESR PAPFLIHQES NVIVRAFRDY LRQDIGEILI DNPKVLELAR QHIAALGRPD FSSKIKLYTG EIPLFSHYQI ESQIESAFQR EVRLPSGGSI VIDSTEALTA IDINSARATR GGDIEETAFN TNLEAADEIA RQLRLRDLGG LIVIDFIDMT PVRHQRAVEN RLREAVRQDR ARIQISHISR FGLLEMSRQR LSPSLGESSH HVCPRCSGTG TVRDNESLSL SILRLIEEEA LKENTQEVHA IVPVPIASYL LNEKRSAVNA IETRQDGVRC VIVPNDQMET PHYHVLRVRK GEETPTLSYM LPKLHEEAMA LPSEEEFAER KRPEQPAL |
-実験情報
| ビーム | 設備名称: Diamond Light Source B21 / 地域: Oxfordshire / 国: UK  / 形状 / 形状 : 1 x 5 mm / 線源: X-ray synchrotron : 1 x 5 mm / 線源: X-ray synchrotron シンクロトロン / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 4.014 mm シンクロトロン / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 4.014 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | ||||||||||||||||||||||||||||||
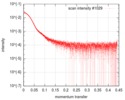
| スキャン |
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
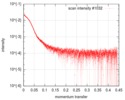
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー


 SASDE52
SASDE52