+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8csv | ||||||
|---|---|---|---|---|---|---|---|
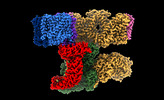
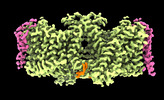
| タイトル | Local refinement of Anykyrin-1 (N-terminal half of membrane binding domain) in Class 2 of erythrocyte ankyrin-1 complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSPORT PROTEIN/STRUCTURAL PROTEIN /  Membrane Protein (膜タンパク質) / Membrane Protein (膜タンパク質) /  Anion Exchange (イオン交換) / Anion Exchange (イオン交換) /  Erythrocyte (赤血球) / Erythrocyte (赤血球) /  Glycoprotein (糖タンパク質) / TRANSPORT PROTEIN-STRUCTURAL PROTEIN complex Glycoprotein (糖タンパク質) / TRANSPORT PROTEIN-STRUCTURAL PROTEIN complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spectrin-associated cytoskeleton / positive regulation of organelle organization / maintenance of epithelial cell apical/basal polarity / pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / NrCAM interactions / Bicarbonate transporters / Neurofascin interactions / intracellular monoatomic ion homeostasis ...spectrin-associated cytoskeleton / positive regulation of organelle organization / maintenance of epithelial cell apical/basal polarity / pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / NrCAM interactions / Bicarbonate transporters / Neurofascin interactions / intracellular monoatomic ion homeostasis /  ankyrin-1 complex / CHL1 interactions / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / cytoskeletal anchor activity / solute:inorganic anion antiporter activity / bicarbonate transport / bicarbonate transmembrane transporter activity / monoatomic anion transport / ankyrin-1 complex / CHL1 interactions / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / cytoskeletal anchor activity / solute:inorganic anion antiporter activity / bicarbonate transport / bicarbonate transmembrane transporter activity / monoatomic anion transport /  M band / Interaction between L1 and Ankyrins / chloride transport / chloride transmembrane transporter activity / M band / Interaction between L1 and Ankyrins / chloride transport / chloride transmembrane transporter activity /  ankyrin binding / negative regulation of glycolytic process through fructose-6-phosphate / ankyrin binding / negative regulation of glycolytic process through fructose-6-phosphate /  spectrin binding / spectrin binding /  hemoglobin binding / cortical cytoskeleton / hemoglobin binding / cortical cytoskeleton /  エキソサイトーシス / erythrocyte development / エキソサイトーシス / erythrocyte development /  axolemma / endoplasmic reticulum to Golgi vesicle-mediated transport / COPI-mediated anterograde transport / protein-membrane adaptor activity / chloride transmembrane transport / cytoskeleton organization / axolemma / endoplasmic reticulum to Golgi vesicle-mediated transport / COPI-mediated anterograde transport / protein-membrane adaptor activity / chloride transmembrane transport / cytoskeleton organization /  筋小胞体 / protein localization to plasma membrane / 筋小胞体 / protein localization to plasma membrane /  regulation of intracellular pH / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / transmembrane transport / structural constituent of cytoskeleton / regulation of intracellular pH / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / transmembrane transport / structural constituent of cytoskeleton /  筋鞘 / cytoplasmic side of plasma membrane / Z disc / 筋鞘 / cytoplasmic side of plasma membrane / Z disc /  凝固・線溶系 / 凝固・線溶系 /  ATPase binding / basolateral plasma membrane / ATPase binding / basolateral plasma membrane /  postsynaptic membrane / postsynaptic membrane /  protein phosphatase binding / transmembrane transporter binding / blood microparticle / protein phosphatase binding / transmembrane transporter binding / blood microparticle /  細胞骨格 / neuron projection / structural molecule activity / 細胞骨格 / neuron projection / structural molecule activity /  enzyme binding / enzyme binding /  シグナル伝達 / protein homodimerization activity / extracellular exosome / シグナル伝達 / protein homodimerization activity / extracellular exosome /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Vallese, F. / Kim, K. / Yen, L.Y. / Johnston, J.D. / Noble, A.J. / Cali, T. / Clarke, O.B. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Architecture of the human erythrocyte ankyrin-1 complex. 著者: Francesca Vallese / Kookjoo Kim / Laura Y Yen / Jake D Johnston / Alex J Noble / Tito Calì / Oliver Biggs Clarke /   要旨: The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association ...The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association with the band 3 anion exchanger and the Rhesus glycoproteins remains unknown. Here we present structures of ankyrin-1 complexes purified from human erythrocytes. We reveal the architecture of a core complex of ankyrin-1, the Rhesus proteins RhAG and RhCE, the band 3 anion exchanger, protein 4.2, glycophorin A and glycophorin B. The distinct T-shaped conformation of membrane-bound ankyrin-1 facilitates recognition of RhCE and, unexpectedly, the water channel aquaporin-1. Together, our results uncover the molecular details of ankyrin-1 association with the erythrocyte membrane, and illustrate the mechanism of ankyrin-mediated membrane protein clustering. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8csv.cif.gz 8csv.cif.gz | 152.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8csv.ent.gz pdb8csv.ent.gz | 88.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8csv.json.gz 8csv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cs/8csv https://data.pdbj.org/pub/pdb/validation_reports/cs/8csv ftp://data.pdbj.org/pub/pdb/validation_reports/cs/8csv ftp://data.pdbj.org/pub/pdb/validation_reports/cs/8csv | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  26972MC  7uz3C  7uzeC  7uzqC  7uzsC  7uzuC  7uzvC  7v07C  7v0kC  7v0mC  7v0qC  7v0sC  7v0tC  7v0uC  7v0xC  7v0yC  7v19C  8crqC  8crrC  8crtC  8cs9C  8cslC  8cswC  8csxC  8csyC  8ct2C  8ct3C  8cteC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 実験データセット #1 | データ参照:  10.6019/EMPIAR-11043 / データの種類: EMPIAR 10.6019/EMPIAR-11043 / データの種類: EMPIAR |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  / ANK-1 / Ankyrin-R / Erythrocyte ankyrin / ANK-1 / Ankyrin-R / Erythrocyte ankyrin分子量: 206522.625 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Homo sapiens (ヒト) / 参照: UniProt: P16157 Homo sapiens (ヒト) / 参照: UniProt: P16157 |
|---|---|
| #2: タンパク質 |  / Anion exchange protein 1 / AE 1 / Anion exchanger 1 / Solute carrier family 4 member 1 / Anion exchange protein 1 / AE 1 / Anion exchanger 1 / Solute carrier family 4 member 1分子量: 101883.859 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Homo sapiens (ヒト) / 参照: UniProt: P02730 Homo sapiens (ヒト) / 参照: UniProt: P02730 |
| #3: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Class 2 of the erythrocyte ankyrin-1 complex (local refinement of N-terminal half of membrane binding domain, bound to N-terminal peptide of band3) タイプ: COMPLEX / Entity ID: #1-#2 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.4 詳細: Final gel filtration buffer contained 0.05% w/v digitonin, 130 mM KCl, 20 mM HEPES, pH 7.4, 1 mM ATP, 1 mM MgCl2, 1 mM PMSF. Peak fractions were concentrated to 8 mg/mL, and 0.01% w/v ...詳細: Final gel filtration buffer contained 0.05% w/v digitonin, 130 mM KCl, 20 mM HEPES, pH 7.4, 1 mM ATP, 1 mM MgCl2, 1 mM PMSF. Peak fractions were concentrated to 8 mg/mL, and 0.01% w/v glycyrrhizic acid was added immediately prior to vitrification. |
| 試料 | 濃度: 8 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: Ankyrin complex mixture purified from digitonin-solubilized erythrocyte ghost membranes |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: 4-6 seconds, wait time 30 seconds |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 500 nm / Cs Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 500 nm / Cs : 2.7 mm / アライメント法: COMA FREE : 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 2.5 sec. / 電子線照射量: 58 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 14464 / 詳細: Two grids were imaged in a single session. |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20.1_4487: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||
CTF補正 | 詳細: Patch CTF (cryoSPARC v3) followed by per particle defocus refinement and refinement of higher order aberrations (cryoSPARC v3) タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||
3次元再構成 | 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 145645 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



























 PDBj
PDBj












