+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8ce5 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
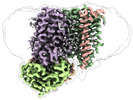
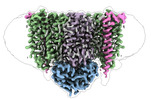
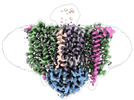
| タイトル | Cytochrome c maturation complex CcmABCD, E154Q, ATP-bound | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / Cytochrome c maturation MEMBRANE PROTEIN (膜タンパク質) / Cytochrome c maturation | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome c biosynthetic process / heme import across plasma membrane / ABC-type heme transporter / ABC-type heme transporter activity / heme transmembrane transporter activity / cytochrome complex assembly / ATP-binding cassette (ABC) transporter complex /  heme binding / heme binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.62 Å クライオ電子顕微鏡法 / 解像度: 3.62 Å | |||||||||||||||
 データ登録者 データ登録者 | Ilcu, L. / Zhang, L. / Einsle, O. | |||||||||||||||
| 資金援助 | European Union,  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Architecture of the Heme-translocating CcmABCD/E complex required for Cytochrome c maturation. 著者: Lorena Ilcu / Lukas Denkhaus / Anton Brausemann / Lin Zhang / Oliver Einsle /  要旨: Mono- and multiheme cytochromes c are post-translationally matured by the covalent attachment of heme. For this, Escherichia coli employs the most complex type of maturation machineries, the Ccm- ...Mono- and multiheme cytochromes c are post-translationally matured by the covalent attachment of heme. For this, Escherichia coli employs the most complex type of maturation machineries, the Ccm-system (for cytochrome c maturation). It consists of two membrane protein complexes, one of which shuttles heme across the membrane to a mobile chaperone that then delivers the cofactor to the second complex, an apoprotein:heme lyase, for covalent attachment. Here we report cryo-electron microscopic structures of the heme translocation complex CcmABCD from E. coli, alone and bound to the heme chaperone CcmE. CcmABCD forms a heterooctameric complex centered around the ABC transporter CcmAB that does not by itself transport heme. Our data suggest that the complex flops a heme group from the inner to the outer leaflet at its CcmBC interfaces, driven by ATP hydrolysis at CcmA. A conserved heme-handling motif (WxWD) at the periplasmic side of CcmC rotates the heme by 90° for covalent attachment to the heme chaperone CcmE that we find interacting exclusively with the CcmB subunit. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8ce5.cif.gz 8ce5.cif.gz | 206.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8ce5.ent.gz pdb8ce5.ent.gz | 163 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8ce5.json.gz 8ce5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ce/8ce5 https://data.pdbj.org/pub/pdb/validation_reports/ce/8ce5 ftp://data.pdbj.org/pub/pdb/validation_reports/ce/8ce5 ftp://data.pdbj.org/pub/pdb/validation_reports/ce/8ce5 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 Aa
| #1: タンパク質 | 分子量: 24410.729 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: ccmA, yejW, b2201, JW5366 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: ccmA, yejW, b2201, JW5366 / 発現宿主:   Escherichia coli K-12 (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C43 / 参照: UniProt: P33931, ABC-type heme transporter Escherichia coli K-12 (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C43 / 参照: UniProt: P33931, ABC-type heme transporter |
|---|
-Heme exporter protein ... , 3種, 4分子 BbCD
| #2: タンパク質 | 分子量: 23632.676 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: ccmB, yejV, b2200, JW2188 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: ccmB, yejV, b2200, JW2188 / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABL8 Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABL8#3: タンパク質 | | 分子量: 27911.264 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: ccmC, yejT, yejU, b2199, JW2187 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: ccmC, yejT, yejU, b2199, JW2187 / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABM1 Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABM1#4: タンパク質 | | 分子量: 7753.103 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: ccmD, yojM, b2198, JW2186 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: ccmD, yojM, b2198, JW2186 / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABM5 Escherichia coli K-12 (大腸菌) / 参照: UniProt: P0ABM5 |
|---|
-非ポリマー , 2種, 4分子 


| #5: 化合物 | | #6: 化合物 |  アデノシン三リン酸 アデノシン三リン酸 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex of cytochrome c maturation sysmtem I, Ccm(AB)2CD タイプ: COMPLEX / Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 由来(組換発現) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: TFS GLACIOS |
|---|---|
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20.1_4487: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
3次元再構成 | 解像度: 3.62 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 355914 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj



