+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8c4e | ||||||
|---|---|---|---|---|---|---|---|
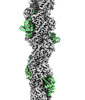
| タイトル | F-actin decorated by SipA426-685 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | CELL INVASION / Salmonella invasion | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Striated Muscle Contraction / skeletal muscle thin filament assembly / striated muscle thin filament / skeletal muscle fiber development /  stress fiber / stress fiber /  マイクロフィラメント / マイクロフィラメント /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 /  actin binding / actin binding /  hydrolase activity / extracellular region / hydrolase activity / extracellular region /  ATP binding ATP binding類似検索 - 分子機能 | ||||||
| 生物種 |   Salmonella (サルモネラ菌) Salmonella (サルモネラ菌)  Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) | ||||||
| 手法 |  電子顕微鏡法 / らせん対称体再構成法 / 電子顕微鏡法 / らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å クライオ電子顕微鏡法 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Yuan, B. / Wald, J. / Marlovits, T.C. | ||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Structural basis for subversion of host cell actin cytoskeleton during infection. 著者: Biao Yuan / Jonas Scholz / Jiri Wald / Roland Thuenauer / Rory Hennell James / Irina Ellenberg / Sabine Windhorst / Jan Faix / Thomas C Marlovits /  要旨: Secreted bacterial type III secretion system (T3SS) proteins are essential for successful infection by many human pathogens. Both T3SS translocator SipC and effector SipA are critical for infection ...Secreted bacterial type III secretion system (T3SS) proteins are essential for successful infection by many human pathogens. Both T3SS translocator SipC and effector SipA are critical for infection by subversion of the host cell cytoskeleton, but the precise molecular interplay between them remains unknown. Here, using cryo-electron microscopy, we show that SipA binds along the F-actin grooves with a unique binding pattern. SipA stabilizes F-actin through charged interface residues and appears to prevent inorganic phosphate release through closure of the "back door" of adenosine 5'-triphosphate pocket. We also show that SipC enhances the binding of SipA to F-actin, thus demonstrating that a sequential presence of T3SS proteins in host cells is associated with a sequence of infection events-starting with actin nucleation, filament growth, and stabilization. Together, our data explain the coordinated interplay of a precisely tuned and highly effective mechanism during infection and provide a blueprint for interfering with effectors acting on actin. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8c4e.cif.gz 8c4e.cif.gz | 1.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8c4e.ent.gz pdb8c4e.ent.gz | 1.1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8c4e.json.gz 8c4e.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c4/8c4e https://data.pdbj.org/pub/pdb/validation_reports/c4/8c4e ftp://data.pdbj.org/pub/pdb/validation_reports/c4/8c4e ftp://data.pdbj.org/pub/pdb/validation_reports/c4/8c4e | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  16425MC  8c4cC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  / Alpha-actin-1 / Alpha-actin-1分子量: 42109.973 Da / 分子数: 8 / 由来タイプ: 天然 / 由来: (天然)   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ)参照: UniProt: P68139,  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与#2: タンパク質 | 分子量: 28413.857 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   Salmonella (サルモネラ菌) / 遺伝子: sipA, sspA / 発現宿主: Salmonella (サルモネラ菌) / 遺伝子: sipA, sspA / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q8VQB5 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q8VQB5#3: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸#4: 化合物 | ChemComp-PO4 /  リン酸塩 リン酸塩#5: 化合物 | ChemComp-MG / 研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: らせん対称体再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 |
| ||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Escherichia coli (大腸菌) / 株: BL21(DE3) Escherichia coli (大腸菌) / 株: BL21(DE3) | ||||||||||||||||||||||||||||
| 緩衝液 | pH: 8 詳細: 5 mM Tris buffer, pH 7.5, 0.1 mM DTT, 0.2 mM ATP, 0.2 mM EGTA and 0.05 mM MgCl2 | ||||||||||||||||||||||||||||
| 試料 | 濃度: 0.5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 800 nm / Cs Bright-field microscopy / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 800 nm / Cs : 2.7 mm / C2レンズ絞り径: 70 µm : 2.7 mm / C2レンズ絞り径: 70 µm |
| 撮影 | 平均露光時間: 3 sec. / 電子線照射量: 70 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: RELION / バージョン: 3.1 / カテゴリ: 3次元再構成 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| らせん対称 | 回転角度/サブユニット: -166.48 ° / 軸方向距離/サブユニット: 28.15 Å / らせん対称軸の対称性: C1 | ||||||||||||||||||||||||
3次元再構成 | 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 120864 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: HELICAL | ||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER | ||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 59.12 Å2 | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj








