+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pom34-Pom152 membrane attachment site yeast NPC | |||||||||||||||
 マップデータ マップデータ | c8 ring reconstructed from multibody volume | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  nuclear pore complex (核膜孔) / nucleocytoplasmic transport / nuclear pore complex (核膜孔) / nucleocytoplasmic transport /  nucleoporin (ヌクレオポリン) / nucleoporin (ヌクレオポリン) /  membrane protein (膜タンパク質) / membrane protein (膜タンパク質) /  translocase (輸送酵素) / translocase (輸送酵素) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear pore transmembrane ring / spindle pole body duplication / nuclear pore organization / structural constituent of nuclear pore / nuclear outer membrane / nucleocytoplasmic transport / nuclear envelope lumen / mRNA transport /  核膜孔 / protein-membrane adaptor activity ...nuclear pore transmembrane ring / spindle pole body duplication / nuclear pore organization / structural constituent of nuclear pore / nuclear outer membrane / nucleocytoplasmic transport / nuclear envelope lumen / mRNA transport / 核膜孔 / protein-membrane adaptor activity ...nuclear pore transmembrane ring / spindle pole body duplication / nuclear pore organization / structural constituent of nuclear pore / nuclear outer membrane / nucleocytoplasmic transport / nuclear envelope lumen / mRNA transport /  核膜孔 / protein-membrane adaptor activity / nuclear periphery / cell periphery / protein import into nucleus / 核膜孔 / protein-membrane adaptor activity / nuclear periphery / cell periphery / protein import into nucleus /  核膜 / 核膜 /  核膜 / 核膜 /  ミトコンドリア ミトコンドリア類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||||||||
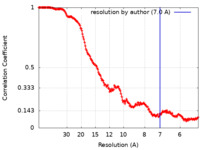
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.0 Å クライオ電子顕微鏡法 / 解像度: 7.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Akey CW / Echeverria I / Ouch C / Fernandez-Martinez J / Rout MP | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Implications of a multiscale structure of the yeast nuclear pore complex. 著者: Christopher W Akey / Ignacia Echeverria / Christna Ouch / Ilona Nudelman / Yi Shi / Junjie Wang / Brian T Chait / Andrej Sali / Javier Fernandez-Martinez / Michael P Rout /   要旨: Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from ...Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from cryogenic electron microscopy and AlphaFold2 models. Key features of the inner and outer rings were integrated into a comprehensive model. We resolved flexible connectors that tie together the core scaffold, along with equatorial transmembrane complexes and a lumenal ring that anchor this channel within the pore membrane. The organization of the nuclear double outer ring reveals an architecture that may be shared with ancestral NPCs. Additional connections between the core scaffold and the central transporter suggest that under certain conditions, a degree of local organization is present at the periphery of the transport machinery. These connectors may couple conformational changes in the scaffold to the central transporter to modulate transport. Collectively, this analysis provides insights into assembly, transport, and NPC evolution. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41117.map.gz emd_41117.map.gz | 2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41117-v30.xml emd-41117-v30.xml emd-41117.xml emd-41117.xml | 36.5 KB 36.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41117_fsc.xml emd_41117_fsc.xml | 21.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41117.png emd_41117.png | 63.6 KB | ||
| Filedesc metadata |  emd-41117.cif.gz emd-41117.cif.gz | 8.5 KB | ||
| その他 |  emd_41117_additional_1.map.gz emd_41117_additional_1.map.gz emd_41117_additional_2.map.gz emd_41117_additional_2.map.gz emd_41117_additional_3.map.gz emd_41117_additional_3.map.gz emd_41117_additional_4.map.gz emd_41117_additional_4.map.gz emd_41117_additional_5.map.gz emd_41117_additional_5.map.gz emd_41117_half_map_1.map.gz emd_41117_half_map_1.map.gz emd_41117_half_map_2.map.gz emd_41117_half_map_2.map.gz | 2 MB 378.7 MB 49.9 MB 49.9 MB 4.3 MB 5.3 MB 5.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41117 http://ftp.pdbj.org/pub/emdb/structures/EMD-41117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41117 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8t9lMC  8tieC  8tj5C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41117.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41117.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | c8 ring reconstructed from multibody volume | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.66 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: extracted TM density for Ndc1
| ファイル | emd_41117_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | extracted TM density for Ndc1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
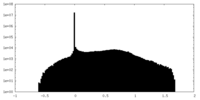
| 密度ヒストグラム |
-追加マップ: extracted region of micelle ring at lower resolution for the Poms.
| ファイル | emd_41117_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | extracted region of micelle ring at lower resolution for the Poms. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
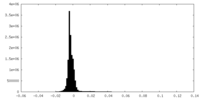
| 密度ヒストグラム |
-追加マップ: half map of multibody volume
| ファイル | emd_41117_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of multibody volume | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
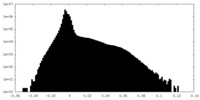
| 密度ヒストグラム |
-追加マップ: half map of multibody volume
| ファイル | emd_41117_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of multibody volume | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
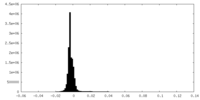
| 密度ヒストグラム |
-追加マップ: c2 map of multibody volume
| ファイル | emd_41117_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | c2 map of multibody volume | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map c8 ring from multibody
| ファイル | emd_41117_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map c8 ring from multibody | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map c8 ring from multibody
| ファイル | emd_41117_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map c8 ring from multibody | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nuclear Pore Complex
| 全体 | 名称: Nuclear Pore Complex 核膜孔 核膜孔 |
|---|---|
| 要素 |
|
-超分子 #1: Nuclear Pore Complex
| 超分子 | 名称: Nuclear Pore Complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Protein A tagged Mlp1 pullout of NPC |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)株: MATa ade2-1 ura3-1 his3-11,15 trp1-1 leu2-3,112 can1-100 MLP1-PPX-ProteinA::HIS5 Organelle: nucleus / 細胞中の位置: nuclear envelope |
-超分子 #2: Pom34-Pom152 Inner ring anchor complex
| 超分子 | 名称: Pom34-Pom152 Inner ring anchor complex / タイプ: complex / ID: 2 / 親要素: 1 詳細: native complex resolved by multibody image processing |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)株: MATa ade2-1 ura3-1 his3-11,15 trp1-1 leu2-3,112 can1-100 MLP1-PPX-ProteinA::HIS5 Organelle: nucleus / 細胞中の位置: nuclear envelope |
-分子 #1: Nucleoporin POM152
| 分子 | 名称: Nucleoporin POM152 / タイプ: protein_or_peptide / ID: 1 詳細: Pore outer membrane protein and anchor for the lumenal ring コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: MATa ade2-1 ura3-1 his3-11 Saccharomyces cerevisiae (パン酵母) / 株: MATa ade2-1 ura3-1 his3-11 |
| 分子量 | 理論値: 151.833891 KDa |
| 配列 | 文字列: MEHRYNVFND TPRGNHWMGS SVSGSPRPSY SSRPNVNTTR RFQYSDDEPA EKIRPLRSRS FKSTESNISD EKSRISERDS KDRYINGDK KVDIYSLPLI STDVLEISKQ RTFAVILFLI IQCYKIYDLV ILKSGLPLSG LLFKNYRFNF ISKYFIIDSF F LYVLPSFN ...文字列: MEHRYNVFND TPRGNHWMGS SVSGSPRPSY SSRPNVNTTR RFQYSDDEPA EKIRPLRSRS FKSTESNISD EKSRISERDS KDRYINGDK KVDIYSLPLI STDVLEISKQ RTFAVILFLI IQCYKIYDLV ILKSGLPLSG LLFKNYRFNF ISKYFIIDSF F LYVLPSFN IPRLTFKPWV VYLQILAMLL LNIFISSDHE FVLISLIMTT WRKLYTKELS VTGSAINHHR IFDSSAHFKG AL TIKILPE NTAMFNPLHE SYCLPMDTNL FKINSIDVPI RINSTEEIEY IELEYRDLYT NSVELRSLSK KDFKIIDNPK SFL KKDQSV LKSHSNDFEE GSTIRYLAVT LQDIGFYQIK KIVDSKKLNL KIHQSHLVVP YCPIASITGT GSNDRCIGDS DNVS FEIQG VPPMKLAYSK IVNGQTFSYV DSSLQPEYFE SPLQSSKSKQ SFTQGELNDL KWGRNQPVNI NLDSSITQDG KFAYK IDKI TDGLGNVVDF TSLPEELKKR YDLSYNFNVH EVPRAALEER FDPKSPTKRS IAIVFEEIKN WISDIPYVIS LSYTDA QDK SKKIMNVTTD SLTKVLQADL PGSYNLEYIE SKFCPGEIVG KSNVLVTMPV APTMEVKSFP ILDQCVGQVG LNFELSF TG APPYYYNTKI YKLENGERKL YDAKRYTSEG TRNRFSYSPP KEGNYEIVFD TVSNKLFTEP IKLEPVKEYT FKTSMRVK P SASLKLHHDL KLCLGDHSSV PVALKGQGPF TLTYDIIETF SSKRKTFEIK EIKTNEYVIK TPVFTTGGDY ILSLVSIKD STGCVVGLSQ PDAKIQVRRD IPSAAFNFFE PIKEAKIKHG SVTEIPLKLS GEGPFTVKFK HMDYDGNIVK EFENKFQNSY KPALKVSKE GLYQLVDIRD SSCQGNVIYR NSLYKVSFLE KPKFAIQDNH HITKVTENLF SKEEVCQGME GTVDLALFGS P PFILEYDL MAPNGHISTK KIQVATKYAS LKLPNQIPGE YITTIKAIFD GNYGESDIHF REHQSELIIK QTVHPIPDVA FA DGGKTLR ACAANVDQIS FLEPINLKFL QGESPFSITF SVYHESTSRT DQYTIDNIDS ENFSFEKLYE GMKLGNHAIT IDS VVDANG CVNSLISGPR NQILVSITDA PKIHILDPST EYCVGDYVAY QLNGVAPFMI KYEFNGIPLK SKERSSQFVR LASE PGIIS ITSLQDSSSQ CIVDFTNPKL KSEFDDLSLN IHPIPSVTVS QGNYVTEDIR EGDQAEVIFS FEGTPPFSLT YVRTE ETDG KHGKRRSQVV ETHKVTDIYS HEYKVITSLQ GTYEAIEITD AYCFAKNDLF FNN UniProtKB: Nucleoporin POM152 |
-分子 #2: Nucleoporin POM34
| 分子 | 名称: Nucleoporin POM34 / タイプ: protein_or_peptide / ID: 2 / 詳細: Pore membrane protein and inner ring anchor / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: 15 trp1-1 leu2-3 Saccharomyces cerevisiae (パン酵母) / 株: 15 trp1-1 leu2-3 |
| 分子量 | 理論値: 34.279301 KDa |
| 配列 | 文字列: MKIQAGQLGL DDNDVPGPLP DTDSKPSSQS QNDTPMFKLG NFESPVLKEL SRRTVNKEME TQRIMTNVIA FAFWNLLVKF IKFFWNNTH VGRQFCNRLS RIHLYMLTFH TLKKANIIYH TTFSWLNAEL LDYLFHLLIS LNILFSLWKL LSTVKVSDLN L TDRQKKLL ...文字列: MKIQAGQLGL DDNDVPGPLP DTDSKPSSQS QNDTPMFKLG NFESPVLKEL SRRTVNKEME TQRIMTNVIA FAFWNLLVKF IKFFWNNTH VGRQFCNRLS RIHLYMLTFH TLKKANIIYH TTFSWLNAEL LDYLFHLLIS LNILFSLWKL LSTVKVSDLN L TDRQKKLL GVDMQSSVDT GLQPQHPHYV STSKISQMAQ NKTHIPQTNL KNHPAYLFKG LETPLKARQR EMAEEQTKLQ SQ SLHTKNV FGTLQRHSGI SSTLVSANND NNSPHTPVTR KGYIPSSKYA YMMNSQSPRG KI UniProtKB: Nucleoporin POM34 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20mM HEPES,50mM Potassium acetate,20mM NaCl,2mM MgCl2,1mM DTT |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK III |
| 詳細 | One step affinity purified |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 37651 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | Preliminary grid screening done manually with individual images of low magnification montages of candidate meshes. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-40 / 撮影したグリッド数: 1 / 実像数: 4015 / 平均電子線量: 40.0 e/Å2 / 詳細: 3218 images retained after triage |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model / 詳細: none |
|---|---|
| 詳細 | Rigid body docking and manual building with Chimera and coot |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: cross correlation |
| 得られたモデル |  PDB-8t9l: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X