+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of hSlo1 in digitonin, Ca2+-free and EDTA-free | |||||||||
 マップデータ マップデータ | unsharpened cryo-EM map of hSlo1 in digitonin, Ca2 -free and EDTA-free | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Acetylcholine inhibits contraction of outer hair cells /  排尿 / Ca2+ activated K+ channels / large conductance calcium-activated potassium channel activity / response to carbon monoxide / 排尿 / Ca2+ activated K+ channels / large conductance calcium-activated potassium channel activity / response to carbon monoxide /  calcium-activated potassium channel activity / negative regulation of cell volume / intracellular potassium ion homeostasis / smooth muscle contraction involved in micturition / Sensory processing of sound by inner hair cells of the cochlea ...Acetylcholine inhibits contraction of outer hair cells / calcium-activated potassium channel activity / negative regulation of cell volume / intracellular potassium ion homeostasis / smooth muscle contraction involved in micturition / Sensory processing of sound by inner hair cells of the cochlea ...Acetylcholine inhibits contraction of outer hair cells /  排尿 / Ca2+ activated K+ channels / large conductance calcium-activated potassium channel activity / response to carbon monoxide / 排尿 / Ca2+ activated K+ channels / large conductance calcium-activated potassium channel activity / response to carbon monoxide /  calcium-activated potassium channel activity / negative regulation of cell volume / intracellular potassium ion homeostasis / smooth muscle contraction involved in micturition / Sensory processing of sound by inner hair cells of the cochlea / response to osmotic stress / calcium-activated potassium channel activity / negative regulation of cell volume / intracellular potassium ion homeostasis / smooth muscle contraction involved in micturition / Sensory processing of sound by inner hair cells of the cochlea / response to osmotic stress /  voltage-gated potassium channel activity / cGMP effects / potassium ion transmembrane transport / voltage-gated potassium channel activity / cGMP effects / potassium ion transmembrane transport /  voltage-gated potassium channel complex / voltage-gated potassium channel complex /  regulation of membrane potential / regulation of membrane potential /  カベオラ / potassium ion transport / response to calcium ion / カベオラ / potassium ion transport / response to calcium ion /  vasodilation / vasodilation /  actin binding / actin binding /  postsynaptic membrane / response to hypoxia / apical plasma membrane / positive regulation of apoptotic process / postsynaptic membrane / response to hypoxia / apical plasma membrane / positive regulation of apoptotic process /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Tao X / Zhao C / MacKinnon R | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Membrane protein isolation and structure determination in cell-derived membrane vesicles. 著者: Xiao Tao / Chen Zhao / Roderick MacKinnon /  要旨: Integral membrane protein structure determination traditionally requires extraction from cell membranes using detergents or polymers. Here, we describe the isolation and structure determination of ...Integral membrane protein structure determination traditionally requires extraction from cell membranes using detergents or polymers. Here, we describe the isolation and structure determination of proteins in membrane vesicles derived directly from cells. Structures of the ion channel Slo1 from total cell membranes and from cell plasma membranes were determined at 3.8 Å and 2.7 Å resolution, respectively. The plasma membrane environment stabilizes Slo1, revealing an alteration of global helical packing, polar lipid, and cholesterol interactions that stabilize previously unresolved regions of the channel and an additional ion binding site in the Ca regulatory domain. The two methods presented enable structural analysis of both internal and plasma membrane proteins without disrupting weakly interacting proteins, lipids, and cofactors that are essential to biological function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40045.map.gz emd_40045.map.gz | 32 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40045-v30.xml emd-40045-v30.xml emd-40045.xml emd-40045.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40045_fsc.xml emd_40045_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40045.png emd_40045.png | 176.9 KB | ||
| その他 |  emd_40045_additional_1.map.gz emd_40045_additional_1.map.gz emd_40045_half_map_1.map.gz emd_40045_half_map_1.map.gz emd_40045_half_map_2.map.gz emd_40045_half_map_2.map.gz | 59.5 MB 59.1 MB 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40045 http://ftp.pdbj.org/pub/emdb/structures/EMD-40045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40045 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40045.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40045.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened cryo-EM map of hSlo1 in digitonin, Ca2 -free and EDTA-free | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
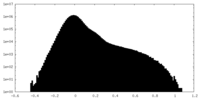
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: cryo-EM map of hSlo1 in digitonin, Ca2 -free...
| ファイル | emd_40045_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM map of hSlo1 in digitonin, Ca2 -free and EDTA-free, sharpened with a B-factor of 120.7 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
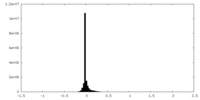
| 密度ヒストグラム |
-ハーフマップ: half map 1 of hSlo1 in digitonin, Ca2 -free and EDTA-free
| ファイル | emd_40045_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 of hSlo1 in digitonin, Ca2 -free and EDTA-free | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
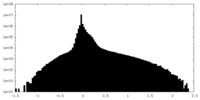
| 密度ヒストグラム |
-ハーフマップ: half map 2 of hSlo1 in digitonin, Ca2 -free and EDTA-free
| ファイル | emd_40045_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 of hSlo1 in digitonin, Ca2 -free and EDTA-free | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
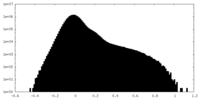
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ALFA-hSlo1 tetrameric channel
| 全体 | 名称: ALFA-hSlo1 tetrameric channel |
|---|---|
| 要素 |
|
-超分子 #1: ALFA-hSlo1 tetrameric channel
| 超分子 | 名称: ALFA-hSlo1 tetrameric channel / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Calcium-activated potassium channel subunit alpha-1
| 分子 | 名称: Calcium-activated potassium channel subunit alpha-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120.908125 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAPSRLEEEL RRRLTEPDAL IIPVTMEVPC DSRGQRMWWA FLASSMVTFF GGLFIILLWR TLKYLWTVCC HCGGKTKEAQ KINNGSSQA DGTLKPVDEK EEAVAAEVGW MTSVKDWAGV MISAQTLTGR VLVVLVFALS IGALVIYFID SSNPIESCQN F YKDFTLQI ...文字列: MAPSRLEEEL RRRLTEPDAL IIPVTMEVPC DSRGQRMWWA FLASSMVTFF GGLFIILLWR TLKYLWTVCC HCGGKTKEAQ KINNGSSQA DGTLKPVDEK EEAVAAEVGW MTSVKDWAGV MISAQTLTGR VLVVLVFALS IGALVIYFID SSNPIESCQN F YKDFTLQI DMAFNVFFLL YFGLRFIAAN DKLWFWLEVN SVVDFFTVPP VFVSVYLNRS WLGLRFLRAL RLIQFSEILQ FL NILKTSN SIKLVNLLSI FISTWLTAAG FIHLVENSGD PWENFQNNQA LTYWECVYLL MVTMSTVGYG DVYAKTTLGR LFM VFFILG GLAMFASYVP EIIELIGNRK KYGGSYSAVS GRKHIVVCGH ITLESVSNFL KDFLHKDRDD VNVEIVFLHN ISPN LELEA LFKRHFTQVE FYQGSVLNPH DLARVKIESA DACLILANKY CADPDAEDAS NIMRVISIKN YHPKIRIITQ MLQYH NKAH LLNIPSWNWK EGDDAICLAE LKLGFIAQSC LAQGLSTMLA NLFSMRSFIK IEEDTWQKYY LEGVSNEMYT EYLSSA FVG LSFPTVCELC FVKLKLLMIA IEYKSANRES RILINPGNHL KIQEGTLGFF IASDAKEVKR AFFYCKACHD DITDPKR IK KCGCKRLEDE QPSTLSPKKK QRNGGMRNSP NTSPKLMRHD PLLIPGNDQI DNMDSNVKKY DSTGMFHWCA PKEIEKVI L TRSEAAMTVL SGHVVVCIFG DVSSALIGLR NLVMPLRASN FHYHELKHIV FVGSIEYLKR EWETLHNFPK VSILPGTPL SRADLRAVNI NLCDMCVILS ANQNNIDDTS LQDKECILAS LNIKSMQFDD SIGVLQANSQ GFTPPGMDRS SPDNSPVHGM LRQPSITTG VNIPIITELV NDTNVQFLDQ DDDDDPDTEL YLTQPFACGT AFAVSVLDSL MSATYFNDNI LTLIRTLVTG G ATPELEAL IAEENALRGG YSTPQTLANR DRCRVAQLAL LDGPFADLGD GGCYGDLFCK ALKTYNMLCF GIYRLRDAHL ST PSQCTKR YVITNPPYEF ELVPTDLIFC LMQFD |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.001 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 0.001 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 18495 / 平均露光時間: 2.0 sec. / 平均電子線量: 51.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X