+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Conformation 2 of the ligand binding domain (LBDconf2) of GluA2 flip Q isoform of AMPA receptor in complex with gain-of-function TARP gamma-2, with 10mM CaCl2, 150mM NaCl, 1mM MgCl2, 330uM CTZ, and 100mM glutamate (Open-CaNaMg) | |||||||||
 マップデータ マップデータ | LBDconf2, Open-CaNaMg | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ionotropic glutamate receptors / AMPA receptors / ionotropic glutamate receptors / AMPA receptors /  ion channel (イオンチャネル) / ion channel (イオンチャネル) /  ligand-gated ion channel (リガンド依存性イオンチャネル) / auxiliary subunit / stargazing / TARP gamma2 / ligand-gated ion channel (リガンド依存性イオンチャネル) / auxiliary subunit / stargazing / TARP gamma2 /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spine synapse / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / response to lithium ion / perisynaptic space / cellular response to glycine / AMPA glutamate receptor activity / Trafficking of GluR2-containing AMPA receptors /  immunoglobulin binding ...spine synapse / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / response to lithium ion / perisynaptic space / cellular response to glycine / AMPA glutamate receptor activity / Trafficking of GluR2-containing AMPA receptors / immunoglobulin binding ...spine synapse / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / response to lithium ion / perisynaptic space / cellular response to glycine / AMPA glutamate receptor activity / Trafficking of GluR2-containing AMPA receptors /  immunoglobulin binding / AMPA glutamate receptor complex / kainate selective glutamate receptor activity / immunoglobulin binding / AMPA glutamate receptor complex / kainate selective glutamate receptor activity /  ionotropic glutamate receptor complex / extracellularly glutamate-gated ion channel activity / asymmetric synapse / regulation of receptor recycling / Unblocking of NMDA receptors, glutamate binding and activation / ionotropic glutamate receptor complex / extracellularly glutamate-gated ion channel activity / asymmetric synapse / regulation of receptor recycling / Unblocking of NMDA receptors, glutamate binding and activation /  glutamate receptor binding / positive regulation of synaptic transmission / glutamate-gated receptor activity / presynaptic active zone membrane / response to fungicide / glutamate receptor binding / positive regulation of synaptic transmission / glutamate-gated receptor activity / presynaptic active zone membrane / response to fungicide /  regulation of synaptic transmission, glutamatergic / cellular response to brain-derived neurotrophic factor stimulus / somatodendritic compartment / dendrite membrane / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / regulation of synaptic transmission, glutamatergic / cellular response to brain-derived neurotrophic factor stimulus / somatodendritic compartment / dendrite membrane / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential /  ionotropic glutamate receptor binding / ionotropic glutamate receptor signaling pathway / dendrite cytoplasm / ionotropic glutamate receptor binding / ionotropic glutamate receptor signaling pathway / dendrite cytoplasm /  cytoskeletal protein binding / cytoskeletal protein binding /  SNARE binding / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / dendritic shaft / SNARE binding / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / dendritic shaft /  synaptic membrane / synaptic membrane /  synaptic transmission, glutamatergic / synaptic transmission, glutamatergic /  PDZ domain binding / postsynaptic density membrane / protein tetramerization / modulation of chemical synaptic transmission / Schaffer collateral - CA1 synapse / establishment of protein localization / PDZ domain binding / postsynaptic density membrane / protein tetramerization / modulation of chemical synaptic transmission / Schaffer collateral - CA1 synapse / establishment of protein localization /  terminal bouton / terminal bouton /  receptor internalization / synaptic vesicle membrane / cerebral cortex development / receptor internalization / synaptic vesicle membrane / cerebral cortex development /  シナプス小胞 / presynapse / シナプス小胞 / presynapse /  signaling receptor activity / signaling receptor activity /  presynaptic membrane / presynaptic membrane /  amyloid-beta binding / amyloid-beta binding /  成長円錐 / 成長円錐 /  perikaryon / chemical synaptic transmission / perikaryon / chemical synaptic transmission /  scaffold protein binding / scaffold protein binding /  postsynaptic membrane / postsynaptic membrane /  樹状突起スパイン / 樹状突起スパイン /  postsynaptic density / neuron projection / postsynaptic density / neuron projection /  神経繊維 / neuronal cell body / 神経繊維 / neuronal cell body /  樹状突起 / 樹状突起 /  シナプス / glutamatergic synapse / protein-containing complex binding / endoplasmic reticulum membrane / シナプス / glutamatergic synapse / protein-containing complex binding / endoplasmic reticulum membrane /  protein kinase binding / protein kinase binding /  細胞膜 / 細胞膜 /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | |||||||||
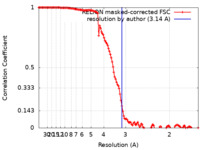
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.14 Å クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Nakagawa T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: The open gate of the AMPA receptor forms a Ca binding site critical in regulating ion transport. 著者: Terunaga Nakagawa / Xin-Tong Wang / Federico J Miguez-Cabello / Derek Bowie /   要旨: Alpha-amino-3-hydroxyl-5-methyl-4-isoxazole-propionic acid receptors (AMPARs) are cation-selective ion channels that mediate most fast excitatory neurotransmission in the brain. Although their gating ...Alpha-amino-3-hydroxyl-5-methyl-4-isoxazole-propionic acid receptors (AMPARs) are cation-selective ion channels that mediate most fast excitatory neurotransmission in the brain. Although their gating mechanism has been studied extensively, understanding how cations traverse the pore has remained elusive. Here we investigated putative ion and water densities in the open pore of Ca-permeable AMPARs (rat GRIA2 flip-Q isoform) at 2.3-2.6 Å resolution. We show that the ion permeation pathway attains an extracellular Ca binding site (site-G) when the channel gate moves into the open configuration. Site-G is highly selective for Ca over Na, favoring the movement of Ca into the selectivity filter of the pore. Seizure-related N619K mutation, adjacent to site-G, promotes channel opening but attenuates Ca binding and thus diminishes Ca permeability. Our work identifies the importance of site-G, which coordinates with the Q/R site of the selectivity filter to ensure the preferential transport of Ca through the channel pore. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29364.map.gz emd_29364.map.gz | 166.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29364-v30.xml emd-29364-v30.xml emd-29364.xml emd-29364.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29364_fsc.xml emd_29364_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29364.png emd_29364.png | 162.4 KB | ||
| マスクデータ |  emd_29364_msk_1.map emd_29364_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-29364.cif.gz emd-29364.cif.gz | 6.6 KB | ||
| その他 |  emd_29364_half_map_1.map.gz emd_29364_half_map_1.map.gz emd_29364_half_map_2.map.gz emd_29364_half_map_2.map.gz | 139 MB 139 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29364 http://ftp.pdbj.org/pub/emdb/structures/EMD-29364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29364 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fphMC  8fp4C  8fp9C  8fpcC  8fpgC  8fpkC  8fplC  8fpsC  8fpvC  8fpyC  8fpzC  8fq1C  8fq2C  8fq3C  8fq5C  8fq6C  8fq8C  8fqaC  8fqbC  8fqdC  8fqeC  8fqfC  8fqgC  8fqhC  8fr0C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29364.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29364.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LBDconf2, Open-CaNaMg | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8195 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_29364_msk_1.map emd_29364_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half1 map, LBDconf2, Open-CaNaMg
| ファイル | emd_29364_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half1 map, LBDconf2, Open-CaNaMg | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half2 map, LBDconf2, Open-CaNaMg
| ファイル | emd_29364_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half2 map, LBDconf2, Open-CaNaMg | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GluA2 flip-Q isoform in complex with TARP gamma2 (K52E, K53E)
| 全体 | 名称: GluA2 flip-Q isoform in complex with TARP gamma2 (K52E, K53E) |
|---|---|
| 要素 |
|
-超分子 #1: GluA2 flip-Q isoform in complex with TARP gamma2 (K52E, K53E)
| 超分子 | 名称: GluA2 flip-Q isoform in complex with TARP gamma2 (K52E, K53E) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 500 KDa |
-分子 #1: Glutamate receptor 2
| 分子 | 名称: Glutamate receptor 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 99.559461 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MQKIMHISVL LSPVLWGLIF GVSSNSIQIG GLFPRGADQE YSAFRVGMVQ FSTSEFRLTP HIDNLEVANS FAVTNAFCSQ FSRGVYAIF GFYDKKSVNT ITSFCGTLHV SFITPSFPTD GTHPFVIQMR PDLKGALLSL IEYYQWDKFA YLYDSDRGLS T LQAVLDSA ...文字列: MQKIMHISVL LSPVLWGLIF GVSSNSIQIG GLFPRGADQE YSAFRVGMVQ FSTSEFRLTP HIDNLEVANS FAVTNAFCSQ FSRGVYAIF GFYDKKSVNT ITSFCGTLHV SFITPSFPTD GTHPFVIQMR PDLKGALLSL IEYYQWDKFA YLYDSDRGLS T LQAVLDSA AEKKWQVTAI NVGNINNDKK DETYRSLFQD LELKKERRVI LDCERDKVND IVDQVITIGK HVKGYHYIIA NL GFTDGDL LKIQFGGANV SGFQIVDYDD SLVSKFIERW STLEEKEYPG AHTATIKYTS ALTYDAVQVM TEAFRNLRKQ RIE ISRRGN AGDCLANPAV PWGQGVEIER ALKQVQVEGL SGNIKFDQNG KRINYTINIM ELKTNGPRKI GYWSEVDKMV VTLT ELPSG NDTSGLENKT VVVTTILESP YVMMKKNHEM LEGNERYEGY CVDLAAEIAK HCGFKYKLTI VGDGKYGARD ADTKI WNGM VGELVYGKAD IAIAPLTITL VREEVIDFSK PFMSLGISIM IKKPQKSKPG VFSFLDPLAY EIWMCIVFAY IGVSVV LFL VSRFSPYEWH TEEFEDGRET QSSESTNEFG IFNSLWFSLG AFMQQGCDIS PRSLSGRIVG GVWWFFTLII ISSYTAN LA AFLTVERMVS PIESAEDLSK QTEIAYGTLD SGSTKEFFRR SKIAVFDKMW TYMRSAEPSV FVRTTAEGVA RVRKSKGK Y AYLLESTMNE YIEQRKPCDT MKVGGNLDSK GYGIATPKGS SLGTPVNLAV LKLSEQGVLD KLKNKWWYDK GECGAKDSG SKEKTSALSL SNVAGVFYIL VGGLGLAMLV ALIEFCYKSR AEAKRMKVAK NPQNINPSSS QNSQNFATDY KDDDDKEGYN VYGIESVKI UniProtKB:  GRIA2 GRIA2 |
-分子 #2: GLUTAMIC ACID
| 分子 | 名称: GLUTAMIC ACID / タイプ: ligand / ID: 2 / コピー数: 4 / 式: GLU |
|---|---|
| 分子量 | 理論値: 147.129 Da |
| Chemical component information |  ChemComp-GLU: |
-分子 #3: CYCLOTHIAZIDE
| 分子 | 名称: CYCLOTHIAZIDE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CYZ |
|---|---|
| 分子量 | 理論値: 389.878 Da |
| Chemical component information | 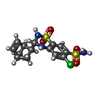 ChemComp-CYZ: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 6 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: L-glutamic acid (100 mM) and cyclothiazide (CTZ, 330 uM) were added before freezing. The 1 M L-glutamic acid stock solution was adjusted to pH 7.4 using NaOH. | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 32662 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

































 Z
Z Y
Y X
X