+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
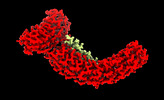
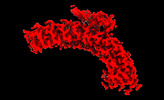
| タイトル | Protein 4.2 (local refinement from consensus reconstruction of ankyrin complex classes) | |||||||||
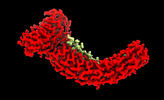
 マップデータ マップデータ | Main map used for model building/refinement. Density modified and cropped using phenix.resolve_cryo_em, resampled to better visualize high resolution features using relion_image_handler. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Membrane Protein (膜タンパク質) / Membrane Protein (膜タンパク質) /  Anion Exchange (イオン交換) / Anion Exchange (イオン交換) /  Erythrocyte (赤血球) / Erythrocyte (赤血球) /  Glycoprotein (糖タンパク質) / Glycoprotein (糖タンパク質) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hemoglobin metabolic process /  protein-glutamine gamma-glutamyltransferase activity / protein-glutamine gamma-glutamyltransferase activity /  ankyrin-1 complex / cortical cytoskeleton / erythrocyte maturation / spleen development / cell morphogenesis / structural constituent of cytoskeleton / multicellular organismal-level iron ion homeostasis / regulation of cell shape ...hemoglobin metabolic process / ankyrin-1 complex / cortical cytoskeleton / erythrocyte maturation / spleen development / cell morphogenesis / structural constituent of cytoskeleton / multicellular organismal-level iron ion homeostasis / regulation of cell shape ...hemoglobin metabolic process /  protein-glutamine gamma-glutamyltransferase activity / protein-glutamine gamma-glutamyltransferase activity /  ankyrin-1 complex / cortical cytoskeleton / erythrocyte maturation / spleen development / cell morphogenesis / structural constituent of cytoskeleton / multicellular organismal-level iron ion homeostasis / regulation of cell shape / ankyrin-1 complex / cortical cytoskeleton / erythrocyte maturation / spleen development / cell morphogenesis / structural constituent of cytoskeleton / multicellular organismal-level iron ion homeostasis / regulation of cell shape /  細胞骨格 / 細胞骨格 /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
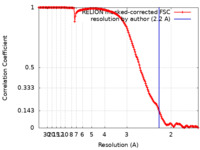
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.2 Å クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Vallese F / Kim K / Yen LY / Johnston JD / Noble AJ / Cali T / Clarke OB | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Architecture of the human erythrocyte ankyrin-1 complex. 著者: Francesca Vallese / Kookjoo Kim / Laura Y Yen / Jake D Johnston / Alex J Noble / Tito Calì / Oliver Biggs Clarke /   要旨: The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association ...The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association with the band 3 anion exchanger and the Rhesus glycoproteins remains unknown. Here we present structures of ankyrin-1 complexes purified from human erythrocytes. We reveal the architecture of a core complex of ankyrin-1, the Rhesus proteins RhAG and RhCE, the band 3 anion exchanger, protein 4.2, glycophorin A and glycophorin B. The distinct T-shaped conformation of membrane-bound ankyrin-1 facilitates recognition of RhCE and, unexpectedly, the water channel aquaporin-1. Together, our results uncover the molecular details of ankyrin-1 association with the erythrocyte membrane, and illustrate the mechanism of ankyrin-mediated membrane protein clustering. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26917.map.gz emd_26917.map.gz | 74.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26917-v30.xml emd-26917-v30.xml emd-26917.xml emd-26917.xml | 32.1 KB 32.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26917_fsc.xml emd_26917_fsc.xml | 15.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26917.png emd_26917.png | 53.8 KB | ||
| Filedesc metadata |  emd-26917.cif.gz emd-26917.cif.gz | 6.5 KB | ||
| その他 |  emd_26917_additional_1.map.gz emd_26917_additional_1.map.gz emd_26917_additional_2.map.gz emd_26917_additional_2.map.gz emd_26917_additional_3.map.gz emd_26917_additional_3.map.gz emd_26917_additional_4.map.gz emd_26917_additional_4.map.gz emd_26917_half_map_1.map.gz emd_26917_half_map_1.map.gz emd_26917_half_map_2.map.gz emd_26917_half_map_2.map.gz | 322.3 MB 322.3 MB 552.4 KB 328.2 MB 74 MB 74 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26917 http://ftp.pdbj.org/pub/emdb/structures/EMD-26917 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26917 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26917 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uzsMC  7uz3C  7uzeC  7uzqC  7uzuC  7uzvC  7v07C  7v0kC  7v0mC  7v0qC  7v0sC  7v0tC  7v0uC  7v0xC  7v0yC  7v19C  8crqC  8crrC  8crtC  8cs9C  8cslC  8csvC  8cswC  8csxC  8csyC  8ct2C  8ct3C  8cteC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26917.map.gz / 形式: CCP4 / 大きさ: 80.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26917.map.gz / 形式: CCP4 / 大きさ: 80.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map used for model building/refinement. Density modified and cropped using phenix.resolve_cryo_em, resampled to better visualize high resolution features using relion_image_handler. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.415 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Half map 1 (unmodified)
| ファイル | emd_26917_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
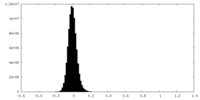
| 注釈 | Half map 1 (unmodified) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Half map 2 (unmodified)
| ファイル | emd_26917_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
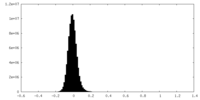
| 注釈 | Half map 2 (unmodified) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Mask used for FSC calculation.
| ファイル | emd_26917_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
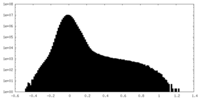
| 注釈 | Mask used for FSC calculation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharpened (B=56.5) map.
| ファイル | emd_26917_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
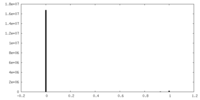
| 注釈 | Sharpened (B=56.5) map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 (cropped and resampled).
| ファイル | emd_26917_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 (cropped and resampled). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 (cropped and resampled).
| ファイル | emd_26917_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 (cropped and resampled). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ankyrin complex mixture purified from digitonin-solubilized eryth...
| 全体 | 名称: Ankyrin complex mixture purified from digitonin-solubilized erythrocyte ghost membranes |
|---|---|
| 要素 |
|
-超分子 #1: Ankyrin complex mixture purified from digitonin-solubilized eryth...
| 超分子 | 名称: Ankyrin complex mixture purified from digitonin-solubilized erythrocyte ghost membranes タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Protein 4.2 (local refinement from consensus refinement of ankyrin complex) |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 器官: Blood / 組織: Erythrocytes / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 器官: Blood / 組織: Erythrocytes / 細胞中の位置: Plasma membrane |
-分子 #1: Protein 4.2
| 分子 | 名称: Protein 4.2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 器官: Blood / 組織: Erythrocytes Homo sapiens (ヒト) / 器官: Blood / 組織: Erythrocytes |
| 分子量 | 理論値: 77.096914 KDa |
| 配列 | 文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKW WSAVVEERDA QSWTISVTTP ADAVIGHYSL LLQVSGRKQL LLGQFTLLFN PWNREDAVFL KNEAQRMEYL L NQNGLIYL ...文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKW WSAVVEERDA QSWTISVTTP ADAVIGHYSL LLQVSGRKQL LLGQFTLLFN PWNREDAVFL KNEAQRMEYL L NQNGLIYL GTADCIQAES WDFGQFEGDV IDLSLRLLSK DKQVEKWSQP VHVARVLGAL LHFLKEQRVL PTPQTQATQE GA LLNKRRG SVPILRQWLT GRGRPVYDGQ AWVLAAVACT VLRCLGIPAR VVTTFASAQG TGGRLLIDEY YNEEGLQNGE GQR GRIWIF QTSTECWMTR PALPQGYDGW QILHPSAPNG GGVLGSCDLV PVRAVKEGTL GLTPAVSDLF AAINASCVVW KCCE DGTLE LTDSNTKYVG NNISTKGVGS DRCEDITQNY KYPEGSLQEK EVLERVEKEK MEREKDNGIR PPSLETASPL YLLLK APSS LPLRGDAQIS VTLVNHSEQE KAVQLAIGVQ AVHYNGVLAA KLWRKKLHLT LSANLEKIIT IGLFFSNFER NPPENT FLR LTAMATHSES NLSCFAQEDI AICRPHLAIK MPEKAEQYQP LTASVSLQNS LDAPMEDCVI SILGRGLIHR ERSYRFR SV WPENTMCAKF QFTPTHVGLQ RLTVEVDCNM FQNLTNYKSV TVVAPELSA UniProtKB:  Protein 4.2 Protein 4.2 |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 193 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: Final gel filtration buffer contained 0.05% w/v digitonin, 130 mM KCl, 20 mM HEPES, pH 7.4, 1 mM ATP, 1 mM MgCl2, 1 mM PMSF. Peak fractions were concentrated to 8 mg/mL and 0.01% ( w/v ...詳細: Final gel filtration buffer contained 0.05% w/v digitonin, 130 mM KCl, 20 mM HEPES, pH 7.4, 1 mM ATP, 1 mM MgCl2, 1 mM PMSF. Peak fractions were concentrated to 8 mg/mL and 0.01% ( w/v glycyrrhizic acid was added immediately prior to vitrification. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 4-6 seconds, wait time 30 seconds. |
| 詳細 | Ankyrin complex mixture purified from digitonin-solubilized erythrocyte ghost membranes |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 14464 / 平均露光時間: 2.5 sec. / 平均電子線量: 58.0 e/Å2 / 詳細: Two grids were imaged in a single session. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




























 Z
Z Y
Y X
X