+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
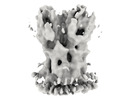
| タイトル | Subtomogram average of the Vaccinia virus (WR) A4/A10 palisade trimer in mature virions | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Palisade / A10 / A4 / p4a /  Vaccinia (ワクシニアウイルス) / Vaccinia (ワクシニアウイルス) /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 virion component / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / structural molecule activity / virion component / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / structural molecule activity /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Vaccinia virus WR (ワクシニアウイルス) Vaccinia virus WR (ワクシニアウイルス) | |||||||||
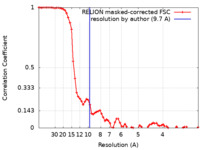
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 9.7 Å クライオ電子顕微鏡法 / 解像度: 9.7 Å | |||||||||
 データ登録者 データ登録者 | Calcraft T / Hernandez-Gonzalez M / Nans A / Rosenthal PB / Way M | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2024 ジャーナル: mBio / 年: 2024タイトル: Palisade structure in intact vaccinia virions. 著者: Miguel Hernandez-Gonzalez / Thomas Calcraft / Andrea Nans / Peter B Rosenthal / Michael Way /  要旨: Vaccinia virus assembly in the cytoplasm of infected cells involves the formation of a biconcave viral core inside the maturing viral particle. The boundary of the core is defined by a ...Vaccinia virus assembly in the cytoplasm of infected cells involves the formation of a biconcave viral core inside the maturing viral particle. The boundary of the core is defined by a pseudohexagonal palisade layer, composed of trimers projecting from an inner wall. To understand the assembly of this complex core architecture, we obtained a subnanometer structure of the palisade trimer by cryo-electron tomography and subtomogram averaging of purified intact virions. Using AlphaFold2 structure predictions, we determined that the palisade is formed from trimers of the proteolytically processed form of the viral protein A10. In addition, we found that each A10 protomer associates with an α-helix (residues 24-66) of A4. Cellular localization assays outside the context of infection demonstrate that the A4 N-terminus is necessary and sufficient to interact with A10. The interaction between A4 and A10 provides insights into how the palisade layer might become tightly associated with the viral membrane during virion maturation. Reconstruction of the palisade layer reveals that, despite local hexagonal ordering, the A10/A4 trimers are widely spaced, suggesting that additional components organize the lattice. This spacing would, however, allow the adoption of the characteristic biconcave shape of the viral core. Finally, we also found that the palisade incorporates multiple copies of a hexameric portal structure. We suggest that these portals are formed by E6, a viral protein that is essential for virion assembly and required to release viral mRNA from the core early in infection.IMPORTANCEPoxviruses such as variola virus (smallpox) and monkeypox cause diseases in humans. Other poxviruses, including vaccinia and modified vaccinia Ankara, are used as vaccine vectors. Given their importance, a greater structural understanding of poxvirus virions is needed. We now performed cryo-electron tomography of purified intact vaccinia virions to study the structure of the palisade, a protein lattice that defines the viral core boundary. We identified the main viral proteins that form the palisade and their interaction surfaces and provided new insights into the organization of the viral core. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18918.map.gz emd_18918.map.gz | 2.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18918-v30.xml emd-18918-v30.xml emd-18918.xml emd-18918.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18918_fsc.xml emd_18918_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18918.png emd_18918.png | 71 KB | ||
| マスクデータ |  emd_18918_msk_1.map emd_18918_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18918.cif.gz emd-18918.cif.gz | 6.5 KB | ||
| その他 |  emd_18918_half_map_1.map.gz emd_18918_half_map_1.map.gz emd_18918_half_map_2.map.gz emd_18918_half_map_2.map.gz | 13.3 MB 13.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18918 http://ftp.pdbj.org/pub/emdb/structures/EMD-18918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18918 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8r5iMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18918.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18918.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.56 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18918_msk_1.map emd_18918_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
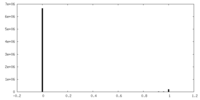
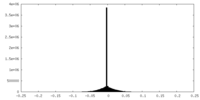
| 投影像・断面図 |
| ||||||||||||
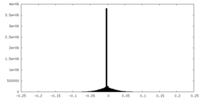
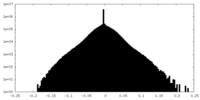
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18918_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
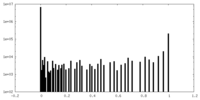
| 投影像・断面図 |
| ||||||||||||
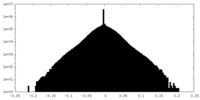
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18918_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vaccinia virus WR
| 全体 | 名称:   Vaccinia virus WR (ワクシニアウイルス) Vaccinia virus WR (ワクシニアウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Vaccinia virus WR
| 超分子 | 名称: Vaccinia virus WR / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: A36-YdF / NCBI-ID: 10254 / 生物種: Vaccinia virus WR / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Core protein A10
| 分子 | 名称: Core protein A10 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vaccinia virus WR (ワクシニアウイルス) Vaccinia virus WR (ワクシニアウイルス) |
| 分子量 | 理論値: 71.073477 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MMPIKSIVTL DQLEDSEYLF RIVSTVLPHL CLDYKVCDQL KTTFVHPFDI LLNNSLGSVT KQDELQAAIS KLGINYLIDT TSRELKLFN VTLNAGNIDI INTPINISSE TNPIINTHSF YDLPPFTQHL LNIRLTDTEY RARFIGGYIK PDGSDSMDVL A EKKYPDLN ...文字列: MMPIKSIVTL DQLEDSEYLF RIVSTVLPHL CLDYKVCDQL KTTFVHPFDI LLNNSLGSVT KQDELQAAIS KLGINYLIDT TSRELKLFN VTLNAGNIDI INTPINISSE TNPIINTHSF YDLPPFTQHL LNIRLTDTEY RARFIGGYIK PDGSDSMDVL A EKKYPDLN FDNTYLFNIL YKDVINAPIK EFKAKIVNGV LSRQDFDNLI GVRQYITIQD RPRFDDAYNI ADAARHYGVN LN TLPLPNV DLTTMPTYKH LIMFEQYFIY TYDRVDIYYN GNKMLFDDEI INFTISMRYQ SLIPRLVDFF PDIPVNNNIV LHT RDPQNA AVNVTVALPN VQFVDINRNN KFFINFFNLL AKEQRSTAIK VTKSMFWDGM DYEEYKSKNL QDMMFINSTC YVFG LYNHN NTTYCSILSD IISAEKTPIR VCLLPRVVGG KTVTNLISET LKSISSMTIR EFPRKDKSIM HIGLSETGFM RFFQL LRLM ADKPHETAIK EVVMAYVGIK LGDKGSPYYI RKESYQDFIY LLFASMGFKV TTRRSIMGSN NISIISIRPR VTKQYI VAT LMKTSCSKNE AEKLITSAFD LLNFMVSVSD FRDYQSYRQY RNYCPRYFYA G UniProtKB: Major core protein OPG136 precursor |
-分子 #2: Core protein A4
| 分子 | 名称: Core protein A4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vaccinia virus WR (ワクシニアウイルス) Vaccinia virus WR (ワクシニアウイルス) |
| 分子量 | 理論値: 30.95235 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDFFNKFSQG LAESSTPKSS IYYSEEKDPD TKKDEAIEIG LKSQESYYQR QLREQLARDN MTVASRQPIQ PLQPTIHITP QPVPTATPA PILLPSSTVP TPKPRQQTNT SSDMSNLFDW LSEDTDAPAS SLLPALTPSN AVQDIISKFN KDQKTTTPPS T QPSQTLPT ...文字列: MDFFNKFSQG LAESSTPKSS IYYSEEKDPD TKKDEAIEIG LKSQESYYQR QLREQLARDN MTVASRQPIQ PLQPTIHITP QPVPTATPA PILLPSSTVP TPKPRQQTNT SSDMSNLFDW LSEDTDAPAS SLLPALTPSN AVQDIISKFN KDQKTTTPPS T QPSQTLPT TTCTQQSDGN ISCTTPTVTP PQPPIVATVC TPTPTGGTVC TTAQQNPNPG AASQQNLDDM ALKDLMSNVE RD MHQLQAE TNDLVTNVYD AREYTRRAID QILQLVKGFE RFQK UniProtKB: 39kDa core protein OPG130 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 詳細: 45mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 平均電子線量: 2.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8r5i: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X