+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13144 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellular monoatomic cation homeostasis / xenobiotic detoxification by transmembrane export across the plasma membrane / ABC-type xenobiotic transporter activity / response to cycloheximide / cell periphery / response to xenobiotic stimulus / response to antibiotic /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  ATP binding ...intracellular monoatomic cation homeostasis / xenobiotic detoxification by transmembrane export across the plasma membrane / ABC-type xenobiotic transporter activity / response to cycloheximide / cell periphery / response to xenobiotic stimulus / response to antibiotic / ATP binding ...intracellular monoatomic cation homeostasis / xenobiotic detoxification by transmembrane export across the plasma membrane / ABC-type xenobiotic transporter activity / response to cycloheximide / cell periphery / response to xenobiotic stimulus / response to antibiotic /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.13 Å クライオ電子顕微鏡法 / 解像度: 3.13 Å | |||||||||
 データ登録者 データ登録者 | Szewczak-Harris A / Wagner M / Du D / Schmitt L / Luisi BF | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure and efflux mechanism of the yeast pleiotropic drug resistance transporter Pdr5. 著者: Andrzej Harris / Manuel Wagner / Dijun Du / Stefanie Raschka / Lea-Marie Nentwig / Holger Gohlke / Sander H J Smits / Ben F Luisi / Lutz Schmitt /    要旨: Pdr5, a member of the extensive ABC transporter superfamily, is representative of a clinically relevant subgroup involved in pleiotropic drug resistance. Pdr5 and its homologues drive drug efflux ...Pdr5, a member of the extensive ABC transporter superfamily, is representative of a clinically relevant subgroup involved in pleiotropic drug resistance. Pdr5 and its homologues drive drug efflux through uncoupled hydrolysis of nucleotides, enabling organisms such as baker's yeast and pathogenic fungi to survive in the presence of chemically diverse antifungal agents. Here, we present the molecular structure of Pdr5 solved with single particle cryo-EM, revealing details of an ATP-driven conformational cycle, which mechanically drives drug translocation through an amphipathic channel, and a clamping switch within a conserved linker loop that acts as a nucleotide sensor. One half of the transporter remains nearly invariant throughout the cycle, while its partner undergoes changes that are transmitted across inter-domain interfaces to support a peristaltic motion of the pumped molecule. The efflux model proposed here rationalises the pleiotropic impact of Pdr5 and opens new avenues for the development of effective antifungal compounds. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13144.map.gz emd_13144.map.gz | 500.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13144-v30.xml emd-13144-v30.xml emd-13144.xml emd-13144.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13144.png emd_13144.png | 49.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13144 http://ftp.pdbj.org/pub/emdb/structures/EMD-13144 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13144 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13144 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13144.map.gz / 形式: CCP4 / 大きさ: 548.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13144.map.gz / 形式: CCP4 / 大きさ: 548.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.652 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward...
| 全体 | 名称: Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward...
| 超分子 | 名称: Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)細胞中の位置: cell membrane |
-分子 #1: Pleiotropic ABC efflux transporter of multiple drugs
| 分子 | 名称: Pleiotropic ABC efflux transporter of multiple drugs タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
| 分子量 | 理論値: 170.61725 KDa |
| 配列 | 文字列: MPEAKLNNNV NDVTSYSSAS SSTENAADLH NYNGFDEHTE ARIQKLARTL TAQSMQNSTQ SAPNKSDAQS IFSSGVEGVN PIFSDPEAP GYDPKLDPNS ENFSSAAWVK NMAHLSAADP DFYKPYSLGC AWKNLSASGA SADVAYQSTV VNIPYKILKS G LRKFQRSK ...文字列: MPEAKLNNNV NDVTSYSSAS SSTENAADLH NYNGFDEHTE ARIQKLARTL TAQSMQNSTQ SAPNKSDAQS IFSSGVEGVN PIFSDPEAP GYDPKLDPNS ENFSSAAWVK NMAHLSAADP DFYKPYSLGC AWKNLSASGA SADVAYQSTV VNIPYKILKS G LRKFQRSK ETNTFQILKP MDGCLNPGEL LVVLGRPGSG CTTLLKSISS NTHGFDLGAD TKISYSGYSG DDIKKHFRGE VV YNAEADV HLPHLTVFET LVTVARLKTP QNRIKGVDRE SYANHLAEVA MATYGLSHTR NTKVGNDIVR GVSGGERKRV SIA EVSICG SKFQCWDNAT RGLDSATALE FIRALKTQAD ISNTSATVAI YQCSQDAYDL FNKVCVLDDG YQIYYGPADK AKKY FEDMG YVCPSRQTTA DFLTSVTSPS ERTLNKDMLK KGIHIPQTPK EMNDYWVKSP NYKELMKEVD QRLLNDDEAS REAIK EAHI AKQSKRARPS SPYTVSYMMQ VKYLLIRNMW RLRNNIGFTL FMILGNCSMA LILGSMFFKI MKKGDTSTFY FRGSAM FFA ILFNAFSSLL EIFSLYEARP ITEKHRTYSL YHPSADAFAS VLSEIPSKLI IAVCFNIIFY FLVDFRRNGG VFFFYLL IN IVAVFSMSHL FRCVGSLTKT LSEAMVPASM LLLALSMYTG FAIPKKKILR WSKWIWYINP LAYLFESLLI NEFHGIKF P CAEYVPRGPA YANISSTESV CTVVGAVPGQ DYVLGDDFIR GTYQYYHKDK WRGFGIGMAY VVFFFFVYLF LCEYNEGAK QKGEILVFPR SIVKRMKKRG VLTEKNANDP ENVGERSDLS SDRKMLQESS EEESDTYGEI GLSKSEAIFH WRNLCYEVQI KAETRRILN NVDGWVKPGT LTALMGASGA GKTTLLDCLA ERVTMGVITG DILVNGIPRD KSFPRSIGYC QQQDLHLKTA T VRESLRFS AYLRQPAEVS IEEKNRYVEE VIKILEMEKY ADAVVGVAGE GLNVEQRKRL TIGVELTAKP KLLVFLDEPT SG LDSQTAW SICQLMKKLA NHGQAILCTI HQPSAILMQE FDRLLFMQRG GKTVYFGDLG EGCKTMIDYF ESHGAHKCPA DAN PAEWML EVVGAAPGSH ANQDYYEVWR NSEEYRAVQS ELDWMERELP KKGSITAAED KHEFSQSIIY QTKLVSIRLF QQYW RSPDY LWSKFILTIF NQLFIGFTFF KAGTSLQGLQ NQMLAVFMFT VIFNPILQQY LPSFVQQRDL YEARERPSRT FSWIS FIFA QIFVEVPWNI LAGTIAYFIY YYPIGFYSNA SAAGQLHERG ALFWLFSCAF YVYVGSMGLL VISFNQVAES AANLAS LLF TMSLSFCGVM TTPSAMPRFW IFMYRVSPLT YFIQALLAVG VANVDVKCAD YELLEFTPPS GMTCGQYMEP YLQLAKT GY LTDENATDTC SFCQISTTND YLANVNSFYS ERWRNYGIFI CYIAFNYIAG VFFYWLARVP KKNGKLSKK |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: RHODAMINE 6G
| 分子 | 名称: RHODAMINE 6G / タイプ: ligand / ID: 4 / コピー数: 1 / 式: RHQ |
|---|---|
| 分子量 | 理論値: 443.557 Da |
| Chemical component information | 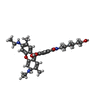 ChemComp-R6G: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: HELIUM |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 1.31 sec. / 平均電子線量: 47.73 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4.1) |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 再構成 | 使用したクラス数: 1 / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.13 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 76419 |
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7p05: |
 ムービー
ムービー コントローラー
コントローラー






















