+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10135 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of murine perforin-2 ectodomain in a pore form | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dendritic cell antigen processing and presentation / antigen processing and presentation of exogenous peptide antigen / phagolysosome membrane /  ファゴリソソーム / pore-forming activity / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane ...dendritic cell antigen processing and presentation / antigen processing and presentation of exogenous peptide antigen / phagolysosome membrane / ファゴリソソーム / pore-forming activity / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane ...dendritic cell antigen processing and presentation / antigen processing and presentation of exogenous peptide antigen / phagolysosome membrane /  ファゴリソソーム / pore-forming activity / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane / cytoplasmic vesicle / defense response to Gram-negative bacterium / ファゴリソソーム / pore-forming activity / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane / cytoplasmic vesicle / defense response to Gram-negative bacterium /  獲得免疫系 / defense response to Gram-positive bacterium / defense response to bacterium 獲得免疫系 / defense response to Gram-positive bacterium / defense response to bacterium類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.0 Å クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||
 データ登録者 データ登録者 | Ni T / Yu X / Gilbert RJC | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2020 ジャーナル: Sci Adv / 年: 2020タイトル: Structure and mechanism of bactericidal mammalian perforin-2, an ancient agent of innate immunity. 著者: Tao Ni / Fang Jiao / Xiulian Yu / Saša Aden / Lucy Ginger / Sophie I Williams / Fangfang Bai / Vojtěch Pražák / Dimple Karia / Phillip Stansfeld / Peijun Zhang / George Munson / Gregor ...著者: Tao Ni / Fang Jiao / Xiulian Yu / Saša Aden / Lucy Ginger / Sophie I Williams / Fangfang Bai / Vojtěch Pražák / Dimple Karia / Phillip Stansfeld / Peijun Zhang / George Munson / Gregor Anderluh / Simon Scheuring / Robert J C Gilbert /    要旨: Perforin-2 (MPEG1) is thought to enable the killing of invading microbes engulfed by macrophages and other phagocytes, forming pores in their membranes. Loss of perforin-2 renders individual ...Perforin-2 (MPEG1) is thought to enable the killing of invading microbes engulfed by macrophages and other phagocytes, forming pores in their membranes. Loss of perforin-2 renders individual phagocytes and whole organisms significantly more susceptible to bacterial pathogens. Here, we reveal the mechanism of perforin-2 activation and activity using atomic structures of pre-pore and pore assemblies, high-speed atomic force microscopy, and functional assays. Perforin-2 forms a pre-pore assembly in which its pore-forming domain points in the opposite direction to its membrane-targeting domain. Acidification then triggers pore formation, via a 180° conformational change. This novel and unexpected mechanism prevents premature bactericidal attack and may have played a key role in the evolution of all perforin family proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10135.map.gz emd_10135.map.gz | 18.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10135-v30.xml emd-10135-v30.xml emd-10135.xml emd-10135.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10135_fsc.xml emd_10135_fsc.xml | 12.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10135.png emd_10135.png | 84.5 KB | ||
| マスクデータ |  emd_10135_msk_1.map emd_10135_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10135_half_map_1.map.gz emd_10135_half_map_1.map.gz emd_10135_half_map_2.map.gz emd_10135_half_map_2.map.gz | 134.5 MB 134.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10135 http://ftp.pdbj.org/pub/emdb/structures/EMD-10135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10135 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10135.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10135.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.22 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
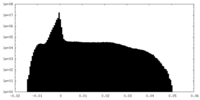
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10135_msk_1.map emd_10135_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
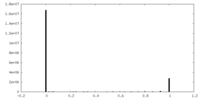
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10135_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
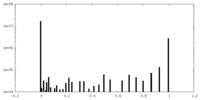
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10135_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
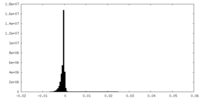
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Murine perforin-2 ecto domain
| 全体 | 名称: Murine perforin-2 ecto domain |
|---|---|
| 要素 |
|
-超分子 #1: Murine perforin-2 ecto domain
| 超分子 | 名称: Murine perforin-2 ecto domain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293T / 組換プラスミド: pHLsec-1D4 Homo sapiens (ヒト) / 組換細胞: HEK293T / 組換プラスミド: pHLsec-1D4 |
-分子 #1: Macrophage-expressed gene 1 protein
| 分子 | 名称: Macrophage-expressed gene 1 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 71.129531 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ETGDKPLGET GTTGFQICKN ALKLPVLEVL PGGGWDNLRN VDMGRVMDLT YTNCKTTEDG QYIIPDEVYT IPQKESNLEM NSEVLESWM NYQSTTSLSI NTELALFSRV NGKFSTEFQR MKTLQVKDQA VTTRVQVRNR IYTVKTTPTS ELSLGFTKAL M DICDQLEK ...文字列: ETGDKPLGET GTTGFQICKN ALKLPVLEVL PGGGWDNLRN VDMGRVMDLT YTNCKTTEDG QYIIPDEVYT IPQKESNLEM NSEVLESWM NYQSTTSLSI NTELALFSRV NGKFSTEFQR MKTLQVKDQA VTTRVQVRNR IYTVKTTPTS ELSLGFTKAL M DICDQLEK NQTKMATYLA ELLILNYGTH VITSVDAGAA LVQEDHVRSS FLLDNQNSQN TVTASAGIAF LNIVNFKVET DY ISQTSLT KDYLSNRTNS RVQSFGGVPF YPGITLETWQ KGITNHLVAI DRAGLPLHFF IKPDKLPGLP GPLVKKLSKT VET AVRHYY TFNTHPGCTN VDSPNFNFQA NMDDDSCDAK VTNFTFGGVY QECTELSGDV LCQNLEQKNL LTGDFSCPPG YSPV HLLSQ THEEGYSRLE CKKKCTLKIF CKTVCEDVFR VAKAEFRAYW CVAAGQVPDN SGLLFGGVFT DKTINPMTNA QSCPA GYIP LNLFESLKVC VSLDYELGFK FSVPFGGFFS CIMGNPLVNS DTAKDVRAPS LKKCPGGFSQ HLAVISDGCQ VSYCVK AGI FTGGSLLPVR LPPYTKPPLM SQVATNTVIV TNSETARSWI KDPQTNQWKL GEPLELRRAM TVIHGDSNGM SGGGTET SQ VAPA |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 32 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 5.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: FEI FALCON III (4k x 4k) #0 - 検出モード: COUNTING / #0 - 平均電子線量: 50.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: FEI FALCON III (4k x 4k) #1 - 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X