+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13119 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
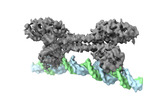
| タイトル | S. agalactiae BusR in complex with its busA-promotor DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報monoatomic cation transmembrane transporter activity / potassium ion transport / DNA-binding transcription factor activity 類似検索 - 分子機能 | |||||||||
| 生物種 |   Streptococcus agalactiae (バクテリア) Streptococcus agalactiae (バクテリア) | |||||||||
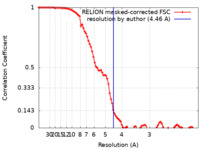
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.46 Å クライオ電子顕微鏡法 / 解像度: 4.46 Å | |||||||||
 データ登録者 データ登録者 | Bandera AM / Witte G | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2021 ジャーナル: Nucleic Acids Res / 年: 2021タイトル: BusR senses bipartite DNA binding motifs by a unique molecular ruler architecture. 著者: Adrian M Bandera / Joseph Bartho / Katja Lammens / David Jan Drexler / Jasmin Kleinschwärzer / Karl-Peter Hopfner / Gregor Witte /  要旨: The cyclic dinucleotide second messenger c-di-AMP is a major player in regulation of potassium homeostasis and osmolyte transport in a variety of bacteria. Along with various direct interactions with ...The cyclic dinucleotide second messenger c-di-AMP is a major player in regulation of potassium homeostasis and osmolyte transport in a variety of bacteria. Along with various direct interactions with proteins such as potassium channels, the second messenger also specifically binds to transcription factors, thereby altering the processes in the cell on the transcriptional level. We here describe the structural and biochemical characterization of BusR from the human pathogen Streptococcus agalactiae. BusR is a member of a yet structurally uncharacterized subfamily of the GntR family of transcription factors that downregulates transcription of the genes for the BusA (OpuA) glycine-betaine transporter upon c-di-AMP binding. We report crystal structures of full-length BusR, its apo and c-di-AMP bound effector domain, as well as cryo-EM structures of BusR bound to its operator DNA. Our structural data, supported by biochemical and biophysical data, reveal that BusR utilizes a unique domain assembly with a tetrameric coiled-coil in between the binding platforms, serving as a molecular ruler to specifically recognize a 22 bp separated bipartite binding motif. Binding of c-di-AMP to BusR induces a shift in equilibrium from an inactivated towards an activated state that allows BusR to bind the target DNA, leading to transcriptional repression. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13119.map.gz emd_13119.map.gz | 4.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13119-v30.xml emd-13119-v30.xml emd-13119.xml emd-13119.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13119_fsc.xml emd_13119_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13119.png emd_13119.png | 57.9 KB | ||
| マスクデータ |  emd_13119_msk_1.map emd_13119_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13119_half_map_1.map.gz emd_13119_half_map_1.map.gz emd_13119_half_map_2.map.gz emd_13119_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13119 http://ftp.pdbj.org/pub/emdb/structures/EMD-13119 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13119 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13119 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13119.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13119.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
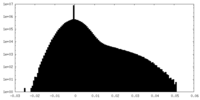
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13119_msk_1.map emd_13119_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
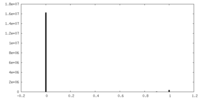
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13119_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
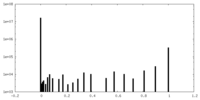
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13119_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
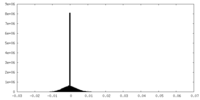
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : transcriptional repressor BusR bound to target DNA
| 全体 | 名称: transcriptional repressor BusR bound to target DNA |
|---|---|
| 要素 |
|
-超分子 #1: transcriptional repressor BusR bound to target DNA
| 超分子 | 名称: transcriptional repressor BusR bound to target DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Streptococcus agalactiae (バクテリア) Streptococcus agalactiae (バクテリア) |
| 分子量 | 理論値: 122 KDa |
-分子 #1: GntR family transcriptional regulator
| 分子 | 名称: GntR family transcriptional regulator / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Streptococcus agalactiae (バクテリア) Streptococcus agalactiae (バクテリア) |
| 分子量 | 理論値: 23.88016 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: GPMVSEQSEI VTSKYQKIAV AVAQRIANGD YEVGEKLKSR TTIASTFNVS PETARKGLNI LADLQILTLK HGSGAIILSK EKAIEFLNQ YETSHSVAIL KGKIRDNIKA QQQEMEELAT LVDDFLLQTR AVSKQYPLAP YEIIVSEDSE HLGKSIGELN V WHQTGATI ...文字列: GPMVSEQSEI VTSKYQKIAV AVAQRIANGD YEVGEKLKSR TTIASTFNVS PETARKGLNI LADLQILTLK HGSGAIILSK EKAIEFLNQ YETSHSVAIL KGKIRDNIKA QQQEMEELAT LVDDFLLQTR AVSKQYPLAP YEIIVSEDSE HLGKSIGELN V WHQTGATI VAIEHEGKFI VSPGPFSVIE QGDHIFFVGD EDVYARMKTY FNLRMGL |
-分子 #2: pBusA_rev
| 分子 | 名称: pBusA_rev / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Streptococcus agalactiae (バクテリア) Streptococcus agalactiae (バクテリア) |
| 分子量 | 理論値: 46.903086 KDa |
| 配列 | 文字列: (DA)(DT)(DA)(DG)(DG)(DA)(DT)(DC)(DC)(DG) (DT)(DG)(DT)(DC)(DT)(DT)(DA)(DT)(DC)(DT) (DA)(DT)(DA)(DA)(DA)(DG)(DT)(DG)(DA) (DC)(DG)(DT)(DT)(DG)(DA)(DG)(DT)(DT)(DA) (DT) (DC)(DG)(DT)(DA)(DA) ...文字列: (DA)(DT)(DA)(DG)(DG)(DA)(DT)(DC)(DC)(DG) (DT)(DG)(DT)(DC)(DT)(DT)(DA)(DT)(DC)(DT) (DA)(DT)(DA)(DA)(DA)(DG)(DT)(DG)(DA) (DC)(DG)(DT)(DT)(DG)(DA)(DG)(DT)(DT)(DA) (DT) (DC)(DG)(DT)(DA)(DA)(DA)(DA)(DG) (DG)(DG)(DT)(DA)(DG)(DT)(DC)(DA)(DC)(DT) (DT)(DT) (DT)(DT)(DT)(DA)(DC)(DT)(DC) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DT)(DA)(DA) (DA)(DC)(DT) (DG)(DC)(DC)(DG)(DA)(DG) (DA)(DC)(DA)(DG)(DA)(DC)(DC)(DA)(DT)(DA) (DA)(DC)(DT)(DA) (DT)(DA)(DA)(DT)(DA) (DT)(DC)(DA)(DT)(DG)(DT)(DA)(DT)(DG)(DG) (DT)(DC)(DT)(DC)(DT) (DT)(DT)(DA)(DT) (DG)(DT)(DC)(DA)(DA)(DC)(DA)(DC)(DC)(DT) (DT)(DG)(DC)(DT)(DT)(DA) (DG)(DA)(DG) (DA)(DA)(DG)(DC)(DT)(DT)(DT)(DA)(DT) |
-分子 #3: pBusA_for
| 分子 | 名称: pBusA_for / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Streptococcus agalactiae (バクテリア) Streptococcus agalactiae (バクテリア) |
| 分子量 | 理論値: 46.903062 KDa |
| 配列 | 文字列: (DA)(DT)(DA)(DA)(DA)(DG)(DC)(DT)(DT)(DC) (DT)(DC)(DT)(DA)(DA)(DG)(DC)(DA)(DA)(DG) (DG)(DT)(DG)(DT)(DT)(DG)(DA)(DC)(DA) (DT)(DA)(DA)(DA)(DG)(DA)(DG)(DA)(DC)(DC) (DA) (DT)(DA)(DC)(DA)(DT) ...文字列: (DA)(DT)(DA)(DA)(DA)(DG)(DC)(DT)(DT)(DC) (DT)(DC)(DT)(DA)(DA)(DG)(DC)(DA)(DA)(DG) (DG)(DT)(DG)(DT)(DT)(DG)(DA)(DC)(DA) (DT)(DA)(DA)(DA)(DG)(DA)(DG)(DA)(DC)(DC) (DA) (DT)(DA)(DC)(DA)(DT)(DG)(DA)(DT) (DA)(DT)(DT)(DA)(DT)(DA)(DG)(DT)(DT)(DA) (DT)(DG) (DG)(DT)(DC)(DT)(DG)(DT)(DC) (DT)(DC)(DG)(DG)(DC)(DA)(DG)(DT)(DT)(DT) (DA)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DG) (DA)(DG)(DT)(DA)(DA)(DA)(DA)(DA)(DA)(DG) (DT)(DG)(DA)(DC) (DT)(DA)(DC)(DC)(DC) (DT)(DT)(DT)(DT)(DA)(DC)(DG)(DA)(DT)(DA) (DA)(DC)(DT)(DC)(DA) (DA)(DC)(DG)(DT) (DC)(DA)(DC)(DT)(DT)(DT)(DA)(DT)(DA)(DG) (DA)(DT)(DA)(DA)(DG)(DA) (DC)(DA)(DC) (DG)(DG)(DA)(DT)(DC)(DC)(DT)(DA)(DT) |
-分子 #4: (2R,3R,3aS,5R,7aR,9R,10R,10aS,12R,14aR)-2,9-bis(6-amino-9H-purin-...
| 分子 | 名称: (2R,3R,3aS,5R,7aR,9R,10R,10aS,12R,14aR)-2,9-bis(6-amino-9H-purin-9-yl)octahydro-2H,7H-difuro[3,2-d:3',2'-j][1,3,7,9,2,8 ]tetraoxadiphosphacyclododecine-3,5,10,12-tetrol 5,12-dioxide タイプ: ligand / ID: 4 / コピー数: 2 / 式: 2BA |
|---|---|
| 分子量 | 理論値: 658.412 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.5 構成要素:
詳細: degassed, filtered | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K 詳細: 0.05% beta-octyl glycoside added prior to plunge freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 詳細 | RCK_C domains were build by rigid body fit of the model 7B5U. The flexible linker between RCK_C domain and coiled-coil domain were build de novo. | ||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 177 | ||||||
| 得られたモデル |  PDB-7oz3: |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X