+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10472 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Euglena gracilis mitochondrial ATP synthase, OSCP/F1/cring rotational state 2 | |||||||||
 マップデータ マップデータ | local resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  mitochondria (ミトコンドリア) / mitochondria (ミトコンドリア) /  ATP synthase (ATP合成酵素) / ATP synthase (ATP合成酵素) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 生物種 |   Euglena gracilis (ミドリムシ) Euglena gracilis (ミドリムシ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.14 Å クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Muhleip A / Amunts A | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Structure of a mitochondrial ATP synthase with bound native cardiolipin. 著者: Alexander Mühleip / Sarah E McComas / Alexey Amunts /  要旨: The mitochondrial ATP synthase fuels eukaryotic cells with chemical energy. Here we report the cryo-EM structure of a divergent ATP synthase dimer from mitochondria of , a member of the phylum ...The mitochondrial ATP synthase fuels eukaryotic cells with chemical energy. Here we report the cryo-EM structure of a divergent ATP synthase dimer from mitochondria of , a member of the phylum Euglenozoa that also includes human parasites. It features 29 different subunits, 8 of which are newly identified. The membrane region was determined to 2.8 Å resolution, enabling the identification of 37 associated lipids, including 25 cardiolipins, which provides insight into protein-lipid interactions and their functional roles. The rotor-stator interface comprises four membrane-embedded horizontal helices, including a distinct subunit . The dimer interface is formed entirely by phylum-specific components, and a peripherally associated subcomplex contributes to the membrane curvature. The central and peripheral stalks directly interact with each other. Last, the ATPase inhibitory factor 1 (IF) binds in a mode that is different from human, but conserved in Trypanosomatids. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10472.map.gz emd_10472.map.gz | 185.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10472-v30.xml emd-10472-v30.xml emd-10472.xml emd-10472.xml | 26.3 KB 26.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10472.png emd_10472.png | 64.6 KB | ||
| マスクデータ |  emd_10472_msk_1.map emd_10472_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10472.cif.gz emd-10472.cif.gz | 7.7 KB | ||
| その他 |  emd_10472_half_map_1.map.gz emd_10472_half_map_1.map.gz emd_10472_half_map_2.map.gz emd_10472_half_map_2.map.gz | 259.9 MB 259.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10472 http://ftp.pdbj.org/pub/emdb/structures/EMD-10472 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10472 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10472 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10472.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10472.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution filtered map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10472_msk_1.map emd_10472_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
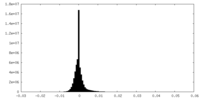
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10472_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
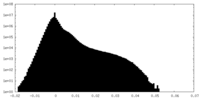
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10472_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Euglena gracilis mitochondrial ATP synthase dimer
+超分子 #1: Euglena gracilis mitochondrial ATP synthase dimer
+分子 #1: oligomycin sensitivity conferring protein (OSCP)
+分子 #2: subunit d
+分子 #3: subunit c
+分子 #4: subunit gamma
+分子 #5: subunit delta
+分子 #6: subunit epsilon
+分子 #7: subunit c
+分子 #8: subunit alpha
+分子 #9: subunit beta
+分子 #10: p18
+分子 #11: inhibitor of F1 (IF1)
+分子 #12: ADENOSINE-5'-TRIPHOSPHATE
+分子 #13: MAGNESIUM ION
+分子 #14: ADENOSINE-5'-DIPHOSPHATE
+分子 #15: FRAGMENT OF TRITON X-100
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 seconds blot. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 実像数: 9045 / 平均露光時間: 10.0 sec. / 平均電子線量: 36.3 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 555269 |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 3次元分類 | ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.14 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 122085 |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X