+検索条件
-Structure paper
| タイトル | Structural basis of hydroxycarboxylic acid receptor signaling mechanisms through ligand binding. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 14, Issue 1, Page 5899, Year 2023 |
| 掲載日 | 2023年9月22日 |
 著者 著者 | Shota Suzuki / Kotaro Tanaka / Kouki Nishikawa / Hiroshi Suzuki / Atsunori Oshima / Yoshinori Fujiyoshi /  |
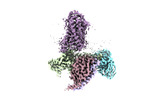
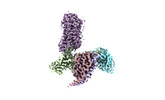
| PubMed 要旨 | Hydroxycarboxylic acid receptors (HCA) are expressed in various tissues and immune cells. HCA2 and its agonist are thus important targets for treating inflammatory and metabolic disorders. Only ...Hydroxycarboxylic acid receptors (HCA) are expressed in various tissues and immune cells. HCA2 and its agonist are thus important targets for treating inflammatory and metabolic disorders. Only limited information is available, however, on the active-state binding of HCAs with agonists. Here, we present cryo-EM structures of human HCA2-Gi and HCA3-Gi signaling complexes binding with multiple compounds bound. Agonists were revealed to form a salt bridge with arginine, which is conserved in the HCA family, to activate these receptors. Extracellular regions of the receptors form a lid-like structure that covers the ligand-binding pocket. Although transmembrane (TM) 6 in HCAs undergoes dynamic conformational changes, ligands do not directly interact with amino acids in TM6, suggesting that indirect signaling induces a slight shift in TM6 to activate Gi proteins. Structural analyses of agonist-bound HCA2 and HCA3 together with mutagenesis and molecular dynamics simulation provide molecular insights into HCA ligand recognition and activation mechanisms. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:37736747 / PubMed:37736747 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.85 - 3.21 Å |
| 構造データ | EMDB-35442, PDB-8ihb: EMDB-35443, PDB-8ihf: EMDB-35444, PDB-8ihh: EMDB-35445, PDB-8ihi: EMDB-35446, PDB-8ihj: EMDB-35447, PDB-8ihk: |
| 化合物 |  ChemComp-NAG:  ChemComp-OKL:  ChemComp-FI7: 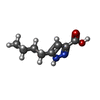 ChemComp-P8A:  ChemComp-P9X: |
| 由来 |
|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  GPCR (Gタンパク質共役受容体) GPCR (Gタンパク質共役受容体) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について