+検索条件
-Structure paper
| タイトル | X-Ray Crystallography and Electron Microscopy of Cross- and Multi-Module Nonribosomal Peptide Synthetase Proteins Reveal a Flexible Architecture. |
|---|---|
| ジャーナル・号・ページ | Structure, Vol. 25, Issue 5, Page 783-793.e4, Year 2017 |
| 掲載日 | 2017年5月2日 |
 著者 著者 | Michael J Tarry / Asfarul S Haque / Khanh Huy Bui / T Martin Schmeing /  |
| PubMed 要旨 | Nonribosomal peptide synthetases (NRPS) are macromolecular machines that produce peptides with diverse activities. Structural information exists for domains, didomains, and even modules, but little ...Nonribosomal peptide synthetases (NRPS) are macromolecular machines that produce peptides with diverse activities. Structural information exists for domains, didomains, and even modules, but little is known about higher-order organization. We performed a multi-technique study on constructs from the dimodular NRPS DhbF. We determined a crystal structure of a cross-module construct including the adenylation (A) and peptidyl carrier protein (PCP) domains from module 1 and the condensation domain from module 2, complexed with an adenosine-vinylsulfonamide inhibitor and an MbtH-like protein (MLP). The action of the inhibitor and the role of the MLP were investigated using adenylation reactions and isothermal titration calorimetry. In the structure, the PCP and A domains adopt a novel conformation, and noncovalent, cross-module interactions are limited. We calculated envelopes of dimodular DhbF using negative-stain electron microscopy. The data show large conformational variability between modules. Together, our results suggest that NRPSs lack a uniform, rigid supermodular architecture. |
 リンク リンク |  Structure / Structure /  PubMed:28434915 PubMed:28434915 |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 3.075 - 25.8 Å |
| 構造データ |  EMDB-8510:  PDB-5u89: |
| 化合物 | 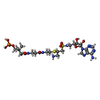 ChemComp-MJ8: |
| 由来 |
|
 キーワード キーワード | HYDROLASE/INHIBITOR /  nonribosomal peptide synthetase (非リボソームペプチド) / MbtH-like protein / nonribosomal peptide synthetase (非リボソームペプチド) / MbtH-like protein /  mechanism-based inhibitor / megaenzyme / HYDROLASE-INHIBITOR complex mechanism-based inhibitor / megaenzyme / HYDROLASE-INHIBITOR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 Anoxybacillus kamchatkensis G10 (バクテリア)
Anoxybacillus kamchatkensis G10 (バクテリア)