+検索条件
-Structure paper
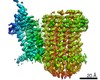
| タイトル | Structure of mycobacterial ATP synthase bound to the tuberculosis drug bedaquiline. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 589, Issue 7840, Page 143-147, Year 2021 |
| 掲載日 | 2020年12月9日 |
 著者 著者 | Hui Guo / Gautier M Courbon / Stephanie A Bueler / Juntao Mai / Jun Liu / John L Rubinstein /  |
| PubMed 要旨 | Tuberculosis-the world's leading cause of death by infectious disease-is increasingly resistant to current first-line antibiotics. The bacterium Mycobacterium tuberculosis (which causes tuberculosis) ...Tuberculosis-the world's leading cause of death by infectious disease-is increasingly resistant to current first-line antibiotics. The bacterium Mycobacterium tuberculosis (which causes tuberculosis) can survive low-energy conditions, allowing infections to remain dormant and decreasing their susceptibility to many antibiotics. Bedaquiline was developed in 2005 from a lead compound identified in a phenotypic screen against Mycobacterium smegmatis. This drug can sterilize even latent M. tuberculosis infections and has become a cornerstone of treatment for multidrug-resistant and extensively drug-resistant tuberculosis. Bedaquiline targets the mycobacterial ATP synthase, which is an essential enzyme in the obligate aerobic Mycobacterium genus, but how it binds the intact enzyme is unknown. Here we determined cryo-electron microscopy structures of M. smegmatis ATP synthase alone and in complex with bedaquiline. The drug-free structure suggests that hook-like extensions from the α-subunits prevent the enzyme from running in reverse, inhibiting ATP hydrolysis and preserving energy in hypoxic conditions. Bedaquiline binding induces large conformational changes in the ATP synthase, creating tight binding pockets at the interface of subunits a and c that explain the potency of this drug as an antibiotic for tuberculosis. |
 リンク リンク |  Nature / Nature /  PubMed:33299175 PubMed:33299175 |
| 手法 | EM (単粒子) |
| 解像度 | 3.2 - 3.7 Å |
| 構造データ | EMDB-22311, PDB-7jg5: EMDB-22312: Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 2 EMDB-22313: Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 3 EMDB-22314: Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase rotational state 1 EMDB-22315: Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase rotational state 2 EMDB-22316, PDB-7jga:  EMDB-22317:  EMDB-22318:  EMDB-22319: EMDB-22320, PDB-7jgb: EMDB-22321, PDB-7jgc:  EMDB-22322: |
| 化合物 |  ChemComp-ATP:  ChemComp-MG:  ChemComp-PO4:  ChemComp-ADP: 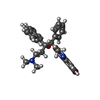 ChemComp-BQ1: |
| 由来 |
|
 キーワード キーワード |  TRANSLOCASE (輸送酵素) / TRANSLOCASE (輸送酵素) /  Bedaquiline (ベダキリン) / Bedaquiline (ベダキリン) /  Sirturo (ベダキリン) / Sirturo (ベダキリン) /  TMC207 (ベダキリン) / T207910 / TMC207 (ベダキリン) / T207910 /  ATP synthase (ATP合成酵素) / ATP synthase (ATP合成酵素) /  mycobacteria (マイコバクテリウム属) / mycobacteria (マイコバクテリウム属) /  tuberculosis (結核) / tuberculosis (結核) /  HYDROLASE (加水分解酵素) HYDROLASE (加水分解酵素) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について