+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7jg5 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
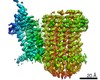
| タイトル | Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 1 | |||||||||
 要素 要素 | (ATP synthase ... ATP合成酵素) x 9 ATP合成酵素) x 9 | |||||||||
 キーワード キーワード |  TRANSLOCASE (輸送酵素) / TRANSLOCASE (輸送酵素) /  Bedaquiline (ベダキリン) / Bedaquiline (ベダキリン) /  Sirturo (ベダキリン) / Sirturo (ベダキリン) /  TMC207 (ベダキリン) / T207910 / TMC207 (ベダキリン) / T207910 /  ATP synthase (ATP合成酵素) / ATP synthase (ATP合成酵素) /  mycobacteria (マイコバクテリウム属) / mycobacteria (マイコバクテリウム属) /  tuberculosis (結核) tuberculosis (結核) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton motive force-driven plasma membrane ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton-transporting ATP synthase complex, catalytic core F(1) /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / membrane => GO:0016020 / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / membrane => GO:0016020 /  hydrolase activity / hydrolase activity /  lipid binding / lipid binding /  ATP hydrolysis activity ...proton motive force-driven plasma membrane ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton-transporting ATP synthase complex, catalytic core F(1) / ATP hydrolysis activity ...proton motive force-driven plasma membrane ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton-transporting ATP synthase complex, catalytic core F(1) /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / membrane => GO:0016020 / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / membrane => GO:0016020 /  hydrolase activity / hydrolase activity /  lipid binding / lipid binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Guo, H. / Courbon, G.M. / Rubinstein, J.L. | |||||||||
| 資金援助 |  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure of mycobacterial ATP synthase bound to the tuberculosis drug bedaquiline. 著者: Hui Guo / Gautier M Courbon / Stephanie A Bueler / Juntao Mai / Jun Liu / John L Rubinstein /  要旨: Tuberculosis-the world's leading cause of death by infectious disease-is increasingly resistant to current first-line antibiotics. The bacterium Mycobacterium tuberculosis (which causes tuberculosis) ...Tuberculosis-the world's leading cause of death by infectious disease-is increasingly resistant to current first-line antibiotics. The bacterium Mycobacterium tuberculosis (which causes tuberculosis) can survive low-energy conditions, allowing infections to remain dormant and decreasing their susceptibility to many antibiotics. Bedaquiline was developed in 2005 from a lead compound identified in a phenotypic screen against Mycobacterium smegmatis. This drug can sterilize even latent M. tuberculosis infections and has become a cornerstone of treatment for multidrug-resistant and extensively drug-resistant tuberculosis. Bedaquiline targets the mycobacterial ATP synthase, which is an essential enzyme in the obligate aerobic Mycobacterium genus, but how it binds the intact enzyme is unknown. Here we determined cryo-electron microscopy structures of M. smegmatis ATP synthase alone and in complex with bedaquiline. The drug-free structure suggests that hook-like extensions from the α-subunits prevent the enzyme from running in reverse, inhibiting ATP hydrolysis and preserving energy in hypoxic conditions. Bedaquiline binding induces large conformational changes in the ATP synthase, creating tight binding pockets at the interface of subunits a and c that explain the potency of this drug as an antibiotic for tuberculosis. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Structure of mycobacterial ATP synthase with the TB drug bedaquiline 著者: Guo, H. / Courbon, G.M. / Bueler, S.A. / Mai, J. / Liu, J. / Rubinstein, J.L. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7jg5.cif.gz 7jg5.cif.gz | 819.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7jg5.ent.gz pdb7jg5.ent.gz | 665.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7jg5.json.gz 7jg5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jg/7jg5 https://data.pdbj.org/pub/pdb/validation_reports/jg/7jg5 ftp://data.pdbj.org/pub/pdb/validation_reports/jg/7jg5 ftp://data.pdbj.org/pub/pdb/validation_reports/jg/7jg5 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase ... , 9種, 20分子 ABCDEFGHabd123456789
| #1: タンパク質 |  ATP合成酵素 / ATP synthase F1 sector subunit alpha / F-ATPase subunit alpha ATP合成酵素 / ATP synthase F1 sector subunit alpha / F-ATPase subunit alpha分子量: 58951.461 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IV93, UniProt: A0R202*PLUS,  ATP合成酵素 ATP合成酵素#2: タンパク質 | |  ATP合成酵素 / ATP synthase F1 sector subunit alpha / F-ATPase subunit alpha ATP合成酵素 / ATP synthase F1 sector subunit alpha / F-ATPase subunit alpha分子量: 58509.234 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IV93, UniProt: A0R202*PLUS,  ATP合成酵素 ATP合成酵素#3: タンパク質 |  ATP合成酵素 / ATP synthase F1 sector subunit beta / F-ATPase subunit beta ATP合成酵素 / ATP synthase F1 sector subunit beta / F-ATPase subunit beta分子量: 51670.453 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IU77, UniProt: A0R200*PLUS,  ATP合成酵素 ATP合成酵素#4: タンパク質 | | 分子量: 33439.836 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IUE3, UniProt: A0R201*PLUS #5: タンパク質 | | 分子量: 13277.741 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IU73, UniProt: A0R1Z9*PLUS #6: タンパク質 | |  ATP合成酵素 / ATP synthase F0 sector subunit a / F-ATPase subunit 6 ATP合成酵素 / ATP synthase F0 sector subunit a / F-ATPase subunit 6分子量: 27568.482 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0R206 #7: タンパク質 | |  ATP合成酵素 / ATP synthase F(0) sector subunit b / ATPase subunit I / F-type ATPase subunit b / F-ATPase subunit b ATP合成酵素 / ATP synthase F(0) sector subunit b / ATPase subunit I / F-type ATPase subunit b / F-ATPase subunit b分子量: 17636.701 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0A0D6IV98, UniProt: A0R204*PLUS #8: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 47504.723 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: A0R203 #9: タンパク質 |  / ATP synthase F(0) sector subunit c / F-type ATPase subunit c / F-ATPase subunit c / Lipid-binding protein / ATP synthase F(0) sector subunit c / F-type ATPase subunit c / F-ATPase subunit c / Lipid-binding protein分子量: 8597.982 Da / 分子数: 9 / 由来タイプ: 天然 由来: (天然)   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア)参照: UniProt: Q5TIX5, UniProt: A0R205*PLUS |
|---|
-非ポリマー , 4種, 10分子 






| #10: 化合物 |  アデノシン三リン酸 アデノシン三リン酸#11: 化合物 | ChemComp-MG / #12: 化合物 |  リン酸塩 リン酸塩#13: 化合物 | ChemComp-ADP / |  アデノシン二リン酸 アデノシン二リン酸 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: ATP synthase from Mycobacterium smegmatis ATP合成酵素 ATP合成酵素タイプ: COMPLEX / Entity ID: #1-#9 / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.55 MDa / 実験値: NO |
| 由来(天然) | 生物種:   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 緩衝液 | pH: 7.4 |
| 試料 | 濃度: 6 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | グリッドの材料: COPPER/RHODIUM / グリッドのタイプ: Homemade |
急速凍結 | 装置: FEI VITROBOT MARK III / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 75000 X / Calibrated defocus min: 800 nm / 最大 デフォーカス(補正後): 2300 nm / Cs Bright-field microscopy / 倍率(公称値): 75000 X / Calibrated defocus min: 800 nm / 最大 デフォーカス(補正後): 2300 nm / Cs : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: ZEMLIN TABLEAU : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: ZEMLIN TABLEAU |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 45 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 7691 |
| 画像スキャン | 横: 4096 / 縦: 4096 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1435679 | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 152372 / アルゴリズム: BACK PROJECTION / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: RECIPROCAL |
 ムービー
ムービー コントローラー
コントローラー































 PDBj
PDBj






