-検索条件
-検索結果
検索 (著者・登録者: kopylov & m)の結果59件中、1から50件目までを表示しています

EMDB-42371: 
Mouse apoferritin imaged with a square aperture

EMDB-42372: 
Mouse apoferritin imaged with a round aperture

EMDB-42373: 
Mouse apoferritin imaged with a square aperture with P2 projection lens rotation, reconstructed from 25,000 particles

EMDB-42374: 
Mouse apoferritin imaged with a round aperture without P2 projection lens rotation, reconstructed from 25,000 particles
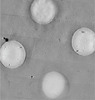
EMDB-42851: 
5x5 tiled montage tomogram of a holey carbon grid with apoferritin imaged with a square electron beam

EMDB-42879: 
3x3 tiled montage tomogram of a yeast lamella imaged with a square electron beam

EMDB-43013: 
Myxococcus xanthus HEnc-K417N(A) protein shell with icosahedral T=1 symmetry

EMDB-43016: 
Myxococcus xanthus HEnc-K417N(A) protein shell with tetrahedral symmetry (12 pentamers, 4 hexamers)

EMDB-43037: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 3 hexamers)

EMDB-43038: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D6 symmetry (12 pentamers, 8 hexamers)

EMDB-43039: 
Myxococcus xanthus HEnc-K417N(A) protein shell with C2 symmetry (12 pentamers, 9 hexamers)

EMDB-43040: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 11 hexamers)

EMDB-43041: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D2 symmetry (12 pentamers, 12 hexamers)

EMDB-43042: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D2 symmetry (12 pentamers, 14 hexamers)

EMDB-43043: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D5 symmetry (12 pentamers, 15 hexamers)

EMDB-43113: 
Myxococcus xanthus EncA WT protein shell with icosahedral symmetry T=3

EMDB-43036: 
Myxococcus xanthus HEnc-K417N(A) protein shell with icosahedral T=3 symmetry

EMDB-29027: 
CryoEM structure of bacteriophage Q-beta coat protein dimer with AYGG linker

PDB-8feh: 
CryoEM structure of bacteriophage Q-beta coat protein dimer with AYGG linker

EMDB-41919: 
Structure of E.coli beta-Galactosidase from a multi-species dataset

EMDB-41923: 
Structure of human Apoferritin from a multi-species dataset

EMDB-41924: 
Structure of Tobacco Mosaic Virus from a multi-species dataset

EMDB-41917: 
CryoEM structure of PP7 virus-like particle from a multi-species dataset

EMDB-28022: 
Adeno-associated virus type 2 VLP displaying M2 peptide
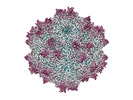
EMDB-28040: 
Adeno-associated virus type 2 VLP displaying Linker peptide

EMDB-29028: 
CryoEM structure of Conalbumin from chicken egg white (sigma-Cas 1391-06-6)

PDB-8fei: 
CryoEM structure of Conalbumin from chicken egg white (sigma-Cas 1391-06-6)
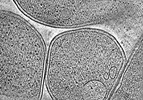
EMDB-26396: 
CryoET of E. coli prepared with the Waffle Method

EMDB-24181: 
The structure of bovine thyroglobulin with iodinated tyrosines

PDB-7n4y: 
The structure of bovine thyroglobulin with iodinated tyrosines

EMDB-21108: 
Structure of DNA Polymerase Zeta (Apo)

EMDB-21115: 
Structure of DNA Polymerase Zeta/DNA/dNTP Ternary Complex

PDB-6v8p: 
Structure of DNA Polymerase Zeta (Apo)

PDB-6v93: 
Structure of DNA Polymerase Zeta/DNA/dNTP Ternary Complex

EMDB-22078: 
Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis

PDB-6x6p: 
Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis

EMDB-21479: 
Class 2 from 3D classification of Bacillus subtilus delta HPF ribosomes

EMDB-21480: 
Class 3 from 3D classification of Bacillus subtilus delta HPF ribosomes

EMDB-21467: 
Consensus map of a delta hpf 70S ribosome from Bacillus subtilis subsp. subtilis str. 168

EMDB-21478: 
Class1 from classification of Bacillus subtilus delta HPF ribosomes

EMDB-20521: 
Horse spleen apoferritin light chain

PDB-6pxm: 
Horse spleen apoferritin light chain

EMDB-0344: 
CryoEM structure of Leviviridae PP7 WT coat protein dimer capsid (PP7PP7-WT)

EMDB-0351: 
CryoEM structure of Leviviridae PP7 WT coat protein dimer capsid (PP7PP7-WT)

EMDB-0352: 
CryoEM structure of Leviviridae PP7 WT coat protein dimer capsid (PP7PP7-WT)

EMDB-0353: 
CryoEM structure of Leviviridae PP7 coat protein dimer capsid with a loop insertion (PP7-aloop-PP7)

EMDB-0354: 
CryoEM structure of Leviviridae PP7 coat protein dimer capsid with a loop insertion and a C-terminal extension (PP7-aloop-PP7-150loop)

PDB-6n4v: 
CryoEM structure of Leviviridae PP7 WT coat protein dimer capsid (PP7PP7-WT)

EMDB-7541: 
Rabbit muscle aldolase at 2.4A resolution (17dec27a 205k particles, all images)

EMDB-7550: 
Rabbit muscle aldolase at 2.4A resolution (17dec27a 205k particles, all images)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
