-検索条件
-検索結果
検索 (著者・登録者: king & ja)の結果181件中、1から50件目までを表示しています

EMDB-42481: 
I53_dn5 nanoparticle displaying the trimeric HA heads with heptad domain, TH-1heptad-I53_dn5

EMDB-42482: 
I53_dn5 nanoparticle displaying the trimeric HA heads with heptad domain, TH-1heptad-I53_dn5 (local refinement of TH-1heptad)

EMDB-42483: 
I53_dn5 nanoparticle displaying the trimeric HA heads with heptad domain, TH-2heptad-I53_dn5

EMDB-42485: 
I53_dn5 nanoparticle displaying the trimeric HA heads with heptad domain, TH-6heptad-I53_dn5

EMDB-42486: 
I53_dn5 nanoparticle displaying the trimeric HA heads with heptad domain, TH-6heptad-I53_dn5 (local refinement of TH-6heptad)

EMDB-18191: 
X. laevis CMG dimer bound to dimeric DONSON - without ATPase

EMDB-18192: 
X. laevis CMG dimer bound to dimeric DONSON - MCM ATPase

EMDB-18195: 
Single CMG purified from replicating Xenopus egg extracts

PDB-8q6o: 
X. laevis CMG dimer bound to dimeric DONSON - without ATPase

PDB-8q6p: 
X. laevis CMG dimer bound to dimeric DONSON - MCM ATPase

EMDB-29857: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

PDB-8g8w: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

EMDB-15616: 
Four subunit cytochrome b-c1 complex from Rhodobacter sphaeroides in native nanodiscs - consensus refinement in the b-b conformation

EMDB-15617: 
Four subunit cytochrome b-c1 complex from Rhodobacter sphaeroides in native nanodiscs - focussed refinement in the b-c conformation

PDB-8asi: 
Four subunit cytochrome b-c1 complex from Rhodobacter sphaeroides in native nanodiscs - consensus refinement in the b-b conformation

PDB-8asj: 
Four subunit cytochrome b-c1 complex from Rhodobacter sphaeroides in native nanodiscs - focussed refinement in the b-c conformation

EMDB-16370: 
Structure of CUL2-KLHDC2 E3 ligase autoinhibited by C-degron mimicry

EMDB-16450: 
Production of antigenically stable enterovirus A71 virus-like particles in Pichia pastoris as a vaccine candidate.

PDB-8c6d: 
Production of antigenically stable enterovirus A71 virus-like particles in Pichia pastoris as a vaccine candidate.

EMDB-25575: 
D8-C4 computationally-designed Rotor

EMDB-25576: 
D3-C5 computationally-designed rotor
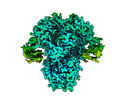
EMDB-26727: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation
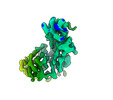
EMDB-26729: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)
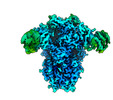
EMDB-26730: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation
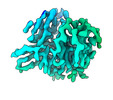
EMDB-26731: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

PDB-7us6: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation

PDB-7us9: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)

PDB-7usa: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation

PDB-7usb: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

EMDB-14106: 
Growing microtubule plus-end in presence of Tip1, Tea2 and Mal3

EMDB-14107: 
Growing microtubules in presence of Tip1, Tea2 and Mal3

EMDB-14108: 
Microtubule plus-end in presence of Mal3

EMDB-14109: 
Growing microtubule minus-end in presence of Mal3

EMDB-14110: 
Growing microtubule plus-end in absence of additional proteins

EMDB-14111: 
Growing microtubule minus-end in absence of additional proteins

EMDB-14112: 
Tip1, Tea2 and Mal3 in presence of PEG without tubulin or microtubules

EMDB-14182: 
Tip1, Tea2 and Mal3 in presence of PEG, microtubules and tubulin

EMDB-13930: 
3D reconstruction of the membrane domains of the sialic acid TRAP transporter HiSiaQM from Haemophilus influenzae in lipid nanodiscs bound to a high affinity megabody

PDB-7qe5: 
Structure of the membrane domains of the sialic acid TRAP transporter HiSiaQM from Haemophilus influenzae

EMDB-33069: 
Cryo-EM structure of neuropeptide Y Y1 receptor in complex with NPY and Gi

EMDB-33070: 
Cryo-EM structure of neuropeptide Y Y2 receptor in complex with NPY and Gi

EMDB-33071: 
Cryo-EM structure of neuropeptide Y Y4 receptor in complex with PP and Gi

PDB-7x9a: 
Cryo-EM structure of neuropeptide Y Y1 receptor in complex with NPY and Gi

PDB-7x9b: 
Cryo-EM structure of neuropeptide Y Y2 receptor in complex with NPY and Gi

PDB-7x9c: 
Cryo-EM structure of neuropeptide Y Y4 receptor in complex with PP and Gi

EMDB-25578: 
D3-C5 computationally-designed rotor (axel only)

EMDB-25579: 
D3-C3 computationally-designed rotor

EMDB-25580: 
C3-C3 computationally-designed rotor

EMDB-25921: 
CryoET of presequence protease single particle

EMDB-13453: 
Cryo-EM structure of the agonist setmelanotide bound to the active melanocortin-4 receptor (MC4R) in complex with the heterotrimeric Gs protein at 2.6 A resolution.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
