-検索条件
-検索結果
検索 (著者・登録者: inoue & s)の結果204件中、1から50件目までを表示しています

EMDB-36467: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (form A)

EMDB-36468: 
Cryo-EM structures of the head region of full-length ERGIC-53 with MCFD2 (form B)

EMDB-36469: 
Cryo-EM structures of the head region of full-length ERGIC-53 with MCFD2 (Substate A)

EMDB-36470: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate B)

EMDB-36471: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate C)

EMDB-36472: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate D)

EMDB-36479: 
Cryo-EM structure of full-length ERGIC-53 with MCFD2
EMDB-36482: 
cryoEM structure of ERGIC-53 deltaH34 mutant with MCFD2

PDB-8jp4: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (form A)

PDB-8jp5: 
Cryo-EM structures of the head region of full-length ERGIC-53 with MCFD2 (form B)

PDB-8jp6: 
Cryo-EM structures of the head region of full-length ERGIC-53 with MCFD2 (Substate A)

PDB-8jp7: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate B)

PDB-8jp8: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate C)

PDB-8jp9: 
Cryo-EM structure of the head region of full-length ERGIC-53 with MCFD2 (Substate D)

PDB-8jpg: 
Cryo-EM structure of full-length ERGIC-53 with MCFD2
EMDB-37881: 
GPR3/Gs complex

PDB-8ww2: 
GPR3/Gs complex

EMDB-35234: 
Cryo-EM structure of Acipimox bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-35235: 
Cryo-EM structure of GSK256073 bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

PDB-8i7v: 
Cryo-EM structure of Acipimox bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

PDB-8i7w: 
Cryo-EM structure of GSK256073 bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-36900: 
Cryo-EM structure of niacin bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-36901: 
Cryo-EM structure of Acipimox bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-36902: 
Cryo-EM structure of GSK256073 bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-34437: 
Cryo-EM structure of niacin bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-36048: 
Cryo-EM structure of hZnT7-Fab complex in zinc-unbound state, determined in outward-facing conformation

EMDB-36049: 
Cryo-EM structure of hZnT7-Fab complex in zinc-bound state, determined in outward-facing conformation

EMDB-36050: 
Cryo-EM structure of hZnT7-Fab complex in zinc-unbound state, determined in heterogeneous conformations- one subunit in an inward-facing and the other in an outward-facing conformation

EMDB-36051: 
Cryo-EM structure of hZnT7-Fab complex in zinc state 2, determined in heterogeneous conformations- one subunit in an inward-facing zinc-bound and the other in an outward-facing zinc-bound conformation

EMDB-36052: 
Cryo-EM structure of hZnT7DeltaHis-loop-Fab complex in zinc-unbound state, determined in outward-facing conformation

EMDB-36053: 
Cryo-EM structure of hZnT7DeltaHis-loop-Fab complex in zinc-bound state, determined in outward-facing conformation

EMDB-36055: 
Cryo-EM structure of hZnT7-Fab complex in zinc state 1, determined in heterogeneous conformations- one subunit in an inward-facing zinc-bound and the other in an outward-facing zinc-unbound conformation

PDB-8j7t: 
Cryo-EM structure of hZnT7-Fab complex in zinc-unbound state, determined in outward-facing conformation

PDB-8j7u: 
Cryo-EM structure of hZnT7-Fab complex in zinc-bound state, determined in outward-facing conformation

PDB-8j7v: 
Cryo-EM structure of hZnT7-Fab complex in zinc-unbound state, determined in heterogeneous conformations- one subunit in an inward-facing and the other in an outward-facing conformation

PDB-8j7w: 
Cryo-EM structure of hZnT7-Fab complex in zinc state 2, determined in heterogeneous conformations- one subunit in an inward-facing zinc-bound and the other in an outward-facing zinc-bound conformation

PDB-8j7x: 
Cryo-EM structure of hZnT7DeltaHis-loop-Fab complex in zinc-unbound state, determined in outward-facing conformation

PDB-8j7y: 
Cryo-EM structure of hZnT7DeltaHis-loop-Fab complex in zinc-bound state, determined in outward-facing conformation

PDB-8j80: 
Cryo-EM structure of hZnT7-Fab complex in zinc state 1, determined in heterogeneous conformations- one subunit in an inward-facing zinc-bound and the other in an outward-facing zinc-unbound conformation

EMDB-34530: 
Membrane protein A

EMDB-34531: 
Membrane protein B

EMDB-35713: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

PDB-8h86: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 in lipid nanodisc

PDB-8h87: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR2 in lipid nanodisc

PDB-8iu0: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc
EMDB-33972: 
Cryo-EM structure of the SARS-CoV-2 spike protein (3-up RBD) bound to neutralizing antibody CSW1-1805
EMDB-33973: 
Cryo-EM structure of the SARS-CoV-2 spike protein (1-up RBD) bound to neutralizing antibody CSW2-1353
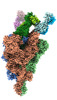
EMDB-33974: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing antibody CSW2-1353
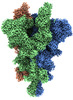
EMDB-33975: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) with C480A mutation

EMDB-34429: 
Cryo-EM structure of KpFtsZ-monobody double helical tube
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
