+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21481 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human diacylglycerol O-acyltransferase 1 complexed with acyl-CoA substrate | |||||||||||||||
 マップデータ マップデータ | CryoEM structure of human diacylglycerol O-acyltransferase 1 complexed with acyl-CoA substrate | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Triglyceride Biosynthesis /  Lipid Storage / Lipid Storage /  Lipid Metabolism (脂質代謝) / Lipid Metabolism (脂質代謝) /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 retinol O-fatty-acyltransferase / retinol O-fatty-acyltransferase /  2-acylglycerol O-acyltransferase activity / 2-acylglycerol O-acyltransferase activity /  retinol O-fatty-acyltransferase activity / monoacylglycerol biosynthetic process / Triglyceride biosynthesis / diacylglycerol metabolic process / Acyl chain remodeling of DAG and TAG / triglyceride biosynthetic process / long-chain fatty-acyl-CoA metabolic process / retinol O-fatty-acyltransferase activity / monoacylglycerol biosynthetic process / Triglyceride biosynthesis / diacylglycerol metabolic process / Acyl chain remodeling of DAG and TAG / triglyceride biosynthetic process / long-chain fatty-acyl-CoA metabolic process /  diacylglycerol O-acyltransferase ... diacylglycerol O-acyltransferase ... retinol O-fatty-acyltransferase / retinol O-fatty-acyltransferase /  2-acylglycerol O-acyltransferase activity / 2-acylglycerol O-acyltransferase activity /  retinol O-fatty-acyltransferase activity / monoacylglycerol biosynthetic process / Triglyceride biosynthesis / diacylglycerol metabolic process / Acyl chain remodeling of DAG and TAG / triglyceride biosynthetic process / long-chain fatty-acyl-CoA metabolic process / retinol O-fatty-acyltransferase activity / monoacylglycerol biosynthetic process / Triglyceride biosynthesis / diacylglycerol metabolic process / Acyl chain remodeling of DAG and TAG / triglyceride biosynthetic process / long-chain fatty-acyl-CoA metabolic process /  diacylglycerol O-acyltransferase / diacylglycerol O-acyltransferase /  diacylglycerol O-acyltransferase activity / very-low-density lipoprotein particle assembly / diacylglycerol O-acyltransferase activity / very-low-density lipoprotein particle assembly /  lipid storage / triglyceride metabolic process / lipid storage / triglyceride metabolic process /  acyltransferase activity / fatty acid homeostasis / specific granule membrane / Neutrophil degranulation / endoplasmic reticulum membrane / acyltransferase activity / fatty acid homeostasis / specific granule membrane / Neutrophil degranulation / endoplasmic reticulum membrane /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Sui X / Wang K / Gluchowski N / Liao M / Walther CT | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure and catalytic mechanism of a human triacylglycerol-synthesis enzyme. 著者: Xuewu Sui / Kun Wang / Nina L Gluchowski / Shane D Elliott / Maofu Liao / Tobias C Walther / Robert V Farese /  要旨: Triacylglycerols store metabolic energy in organisms and have industrial uses as foods and fuels. Excessive accumulation of triacylglycerols in humans causes obesity and is associated with metabolic ...Triacylglycerols store metabolic energy in organisms and have industrial uses as foods and fuels. Excessive accumulation of triacylglycerols in humans causes obesity and is associated with metabolic diseases. Triacylglycerol synthesis is catalysed by acyl-CoA diacylglycerol acyltransferase (DGAT) enzymes, the structures and catalytic mechanisms of which remain unknown. Here we determined the structure of dimeric human DGAT1, a member of the membrane-bound O-acyltransferase (MBOAT) family, by cryo-electron microscopy at approximately 3.0 Å resolution. DGAT1 forms a homodimer through N-terminal segments and a hydrophobic interface, with putative active sites within the membrane region. A structure obtained with oleoyl-CoA substrate resolved at approximately 3.2 Å shows that the CoA moiety binds DGAT1 on the cytosolic side and the acyl group lies deep within a hydrophobic channel, positioning the acyl-CoA thioester bond near an invariant catalytic histidine residue. The reaction centre is located inside a large cavity, which opens laterally to the membrane bilayer, providing lipid access to the active site. A lipid-like density-possibly representing an acyl-acceptor molecule-is located within the reaction centre, orthogonal to acyl-CoA. Insights provided by the DGAT1 structures, together with mutagenesis and functional studies, provide the basis for a model of the catalysis of triacylglycerol synthesis by DGAT. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21481.map.gz emd_21481.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21481-v30.xml emd-21481-v30.xml emd-21481.xml emd-21481.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21481.png emd_21481.png | 57.8 KB | ||
| Filedesc metadata |  emd-21481.cif.gz emd-21481.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21481 http://ftp.pdbj.org/pub/emdb/structures/EMD-21481 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21481 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21481 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21481.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21481.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of human diacylglycerol O-acyltransferase 1 complexed with acyl-CoA substrate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : human diacylglycerol O-acyltransferase 1
| 全体 | 名称: human diacylglycerol O-acyltransferase 1 Diglyceride acyltransferase Diglyceride acyltransferase |
|---|---|
| 要素 |
|
-超分子 #1: human diacylglycerol O-acyltransferase 1
| 超分子 | 名称: human diacylglycerol O-acyltransferase 1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: human diacylglycerol O-acyltransferase 1 complexed with oleoyl-CoA substrate |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Diacylglycerol O-acyltransferase 1
| 分子 | 名称: Diacylglycerol O-acyltransferase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  diacylglycerol O-acyltransferase diacylglycerol O-acyltransferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 55.339133 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGDRGSSRRR RTGSRPSSHG GGGPAAAEEE VRDAAAGPDV GAAGDAPAPA PNKDGDAGVG SGHWELRCHR LQDSLFSSDS GFSNYRGIL NWCVVMLILS NARLFLENLI KYGILVDPIQ VVSLFLKDPY SWPAPCLVIA ANVFAVAAFQ VEKRLAVGAL T EQAGLLLH ...文字列: MGDRGSSRRR RTGSRPSSHG GGGPAAAEEE VRDAAAGPDV GAAGDAPAPA PNKDGDAGVG SGHWELRCHR LQDSLFSSDS GFSNYRGIL NWCVVMLILS NARLFLENLI KYGILVDPIQ VVSLFLKDPY SWPAPCLVIA ANVFAVAAFQ VEKRLAVGAL T EQAGLLLH VANLATILCF PAAVVLLVES ITPVGSLLAL MAHTILFLKL FSYRDVNSWC RRARAKAASA GKKASSAAAP HT VSYPDNL TYRDLYYFLF APTLCYELNF PRSPRIRKRF LLRRILEMLF FTQLQVGLIQ QWMVPTIQNS MKPFKDMDYS RII ERLLKL AVPNHLIWLI FFYWLFHSCL NAVAELMQFG DREFYRDWWN SESVTYFWQN WNIPVHKWCI RHFYKPMLRR GSSK WMART GVFLASAFFH EYLVSVPLRM FRLWAFTGMM AQIPLAWFVG RFFQGNYGNA AVWLSLIIGQ PIAVLMYVHD YYVLN YEAP AAEA UniProtKB:  Diacylglycerol O-acyltransferase 1 Diacylglycerol O-acyltransferase 1 |
-分子 #2: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 2 / コピー数: 4 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 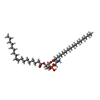 ChemComp-6OU: |
-分子 #3: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydrox...
| 分子 | 名称: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydroxy-3-(phosphonooxy)tetrahydrofuran-2-yl]-3,5,9-trihydroxy-8,8-dimethyl-3,5-dioxido-10,14-dioxo-2,4,6-trioxa-11,15-diaza- ...名称: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydroxy-3-(phosphonooxy)tetrahydrofuran-2-yl]-3,5,9-trihydroxy-8,8-dimethyl-3,5-dioxido-10,14-dioxo-2,4,6-trioxa-11,15-diaza-3lambda~5~,5lambda~5~-diphosphaheptadecan-17-yl} (9Z)-octadec-9-enethioate (non-preferred name) タイプ: ligand / ID: 3 / コピー数: 2 / 式: 3VV |
|---|---|
| 分子量 | 理論値: 1.03198 KDa |
| Chemical component information |  ChemComp-3VV: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: GATAN CRYOPLUNGE 3 |
| 詳細 | Protein sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 平均電子線量: 43.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 28165 |
 ムービー
ムービー コントローラー
コントローラー
















