[wwPDB] PDBアーカイブの2019年1月1日付けのスナップショットを公開しました
2019年1月1日現在の、PDBアーカイブ (wwPDB:ftp://ftp.wwpdb.org、 PDBj:ftp://ftp.pdbj.org)のスナップショットを、スナップショットサイト (wwPDB:ftp://snapshots.wwpdb.org、PDBj:ftp://snapshots.pdbj.org)に追加しました。 2005年1月以来、毎年、スナップショットをアーカイブし、PDBアーカイブに関する研究に役立つデータセットを提供し続けています。
ディレクトリ 20190101は、 147,610件の実験的に決定された座標ファイルと、関連する実験データを含んでいます。 座標及び関連データのファイルは、PDBx/mmCIFフォーマット、PDBフォーマット、XMLフォーマットファイルで、それぞれご利用頂けます。 各ファイルの日付とタイムスタンプは、そのファイルが最後に更新された日付を表しています。 スナップショットのサイズは、1,529GBあります。
[ wwPDB ニュース ]
1/30(木)、蛋白研セミナー:PDBj・BINDSユニット連携講習会を開催します
蛋白研セミナー:PDBj・BINDSユニット連携講習会「創薬・生命科学における構造データの利用法」のご案内
- 開催趣旨
-
タンパク質や化合物の構造データには、創薬や生物学の基礎となる、化合物とタンパク質の相互作用の解析に有効な知見が含まれています。
今回は、BINDSの最適化ユニット(PDBj)、インシリコユニット、ケミカルシーズ・リード探索ユニットが連携して、
こうした構造データを活用した解析法・化合物スクリーニング法について計算機講習・講演を行います。
計算機演習としては、PDBに登録されている化合物-蛋白質の複合体立体構造の活用法(川端)、蛋白質の立体構造比較法(Rozewicki・Standley)、
創薬ターゲットのデータベース(土方・白井)についてPCを用いた実習を行います。また、より実践的な化合物探索について、
大阪大学での化合物ライブラリのスクリーニング支援活動(布村・藤井)、並びに計算機による化合物スクリーニング技術(広川)についての紹介を行います。
※本セミナーは、2020年1月29日のケンブリッジ構造データベース(CSD)利用セミナーと協同で 蛋白研セミナーとして開催いたします。両日参加ではなく1日だけの参加でもかまいません。
- 主催
- 蛋白質研究所、創薬等先端技術支援基盤プラットフォーム(BINDS)
- 日時
- 2020年1月30日(木)10:30—18:00
- 場所
- 大阪大学 蛋白質研究所 本館1階講堂
- 定員
- 40名程度
- 計算機
- 原則として各自でノートPCを持参していただくようお願いいたします。 無線LANが使用できるPCであればOSはWindowsでもMacでもかまいません。 また、阪大で保有するWindows PCの貸し出しも行いますので、希望される方は、参加申し込みの際にお知らせください。 なお、貸出用PCの数には限りがあります。希望者が多い場合は先着順とさせていただきます。
- 使用言語(language)
- 日本語。ただし、Rozewicki先生, Standley先生は英語で講演します。
- 申し込み
- 参加申し込みフォームよりお申込みください。
- 申込締切日時
- 2020年1月29日(水) 17:00
- お問い合わせ
- PDBjのお問い合わせページ をご利用ください。
<プログラム>
2020年1月30日(木)- 10:30-10:35
- 「本講習会の趣旨について」栗栖源嗣 (大阪大学 蛋白質研究所)
- 10:35 – 12:00
- 「HOMCOSによるPDB内の低分子化合物の検索とモデリング」川端 猛 (大阪大学 蛋白質研究所) [講義資料]
- 12:00 – 13:00
- 休憩(昼食)
- 13:00-14:00
- 「大阪大学創薬サイエンス研究支援拠点におけるBINDS支援」
布村 一人(大阪大学・薬・附属化合物ライブラリー・スクリーニングセンター)
[講義資料(パスワード付き)]
藤井 晋太郎(大阪大学・薬・附属創薬センター 構造展開ユニット) [講義資料(パスワード付き)] - 14:05 – 15:05
- 「インシリコスクリーニングによる医薬品候補化合物の探索」
広川 貴次 (産総研・創薬分子プロファイリング研究センター) [講義資料(パスワード付き)] - 15:25 – 16:25
- 「Application of structural alignment in immunology」
John Rozewicki, [講義資料] Daron Standley (大阪大学 微生物研究所) [講義資料] - 16:30 – 17:30
- 「Drug Target Excavator (DTX)による創薬ターゲット探索 」土方敦司・白井 剛(長浜バイオ大学) [講義資料]
生命医薬情報学連合大会(IIBMP2018)にて、PDBjランチョンセミナーを開催します
荘銀タクト鶴岡 にて開催される生命医薬情報学連合大会(IIBMP2018)にてPDBjランチョンセミナーを開催します。
- 日時
- 2018年9月20日(木)12:30-13:30
- 場所
- 荘銀タクト鶴岡 会議室 (B会場) [会場]
- 講演者
- 「PDBj とwwPDB の最近の活動について」栗栖 源嗣(大阪大学蛋白質研究所)
- “Introduction to PDBj services for searching and exploring the PDB” BEKKER, Gert-Jan (大阪大学蛋白質研究所)
要旨および ランチョンセミナープログラムもご覧ください。
1/6(月)・7(火)に、CiCLE: 電子顕微鏡 試料評価講習会を開催します
CiCLE: 電子顕微鏡 試料評価講習会のご案内
開催趣旨:近年の低温電子顕微鏡の単粒子解析の進展は著しく、X線結晶解析に迫る分解能の生体高分子像が得られるようになってきています。 高解像度の解析には、試料の調整が非常に重要です。そこで、電顕の初心者の方を対象に、基本的な試料調整・評価の講習会を企画しました。 低温電子顕微鏡の準備段階に必要な、負染色法によるグリッドの作成と小型の透過型電子顕微鏡での試料評価の実習を行います。
- 日程
- 2020年1月6日(月)、7日(火)※どちらか1日を選択してください
- 実施場所
- 大阪大学 蛋白質研究所
- 主催
- 医療研究開発革新基盤創成事業(CiCLE) 「タンパク質構造解析のハイスループット化へ向けた装置開発」
- 講義・講習の担当
- Christoph Gerle(大阪大学・蛋白質研究所)、栗栖 源嗣(大阪大学・蛋白質研究所)
- 定員
-
1/6, 1/7のそれぞれ5人程度まで。計10名程度。
※受講希望者が定員を超えた場合は、受講希望理由・申し込み順などを参考にして、こちらでセレクションさせていただきます。
実際に電顕の使用を考えている方を優先いたします。 - 事前準備等
- 講習に必要な物品・実験装置、講義資料等はこちらで全て準備します。
- 集合時間・場所
- 朝9:00に、蛋白研・本館1階講堂に集合してください。
- 使用言語(language)
- 日本語と英語
- 参加費
- 無料
- 申し込み
- 参加申し込みフォームよりお申込みください。
- 申し込み締め切り日時
- 2019年12月20日(金) 17:00
- お問い合わせ
- PDBjのお問い合わせページ をご利用ください。
<プログラム>
2020年1月6日 (月)もしくは 1月7日(火)* 1/6と1/7とも同じ内容で講習を行います。参加者は各日5名までに限定いたします。
-
午前:講義 (本館1階講堂)
- 9:15-9:30:
- 講習会の趣旨の説明(栗栖)
- 9:30 - 10:50:
- 負染色法の特徴と概要(Gerle)
- 11:00- 12:00:
- 実験の留意点と試料評価の判断基準(Gerle)
-
午後:実習 (Gerle、三角)
- 13:30 - 15:00:
- グリッド作成 [本館3階]
- 15:10 - 17:00:
- 透過型電子顕微鏡H-7650を用いた負染色法による試料撮影と評価 [構造解析棟1階]
12/3(火)~6(金)に福岡で開催される第42回 日本分子生物学会年会に出展します
PDBjでは、2019年12月3日(火)~6(金)に福岡国際会議場・マリンメッセ福岡で開催される第42回 日本分子生物学会年会に出展し活動の紹介を行います。
【ブース出展】
- タイトル
- 特別企画「使ってみようバイオデータベース-広がるデータ、つながる世界」(BioDBコーナー)
- 日時
- 2019年12月3日(火)~5(木)09:30~17:002019年12月6日(金)09:30~12:45
- 場所
- 出展場所
-
BioDBコーナー12
- 資料
【フォーラム】
- タイトル
- フォーラム(チュートリアル企画)生命科学のデータベース活用法2019
- 日時
- 2019年12月3日(火)18:30~20:00(PDBjからの講演は18:36~18:48)
- 場所
- 資料
11/19(火)、日本結晶学会 令和元年(2019年)度年会にて、ランチョンセミナーを開催します
<PDBj ランチョンセミナー>
日本結晶学会 令和元年(2019年)度年会および会員総会 にて、PDBjはランチョンセミナーを開催します。
| 日時 | 2019年11月19日(火)12:00-13:00 |
|---|---|
| 会場(部屋名) | 金沢市文化ホール A会場:会議棟3階 大会議室 |
| 参加費 | 無料(別途年会参加費は必要) |
<演者・演題>
-
栗栖 源嗣(大阪大学 蛋白質研究所)
"PDBx/mmCIFフォーマットによるPDB登録とデータ検証ポリシーについて" -
Gert-Jan Bekker (大阪大学 蛋白質研究所)
"Editing PDBx/mmCIF files via a web-based CIF editor"
講演要旨:こちら
※ランチョンセミナー当日朝(学会受付開始後)整理券を配布します。
整理券をお持ちの方は開始時間までに会場前でお待ち下さい。
数に限りがあります。(先着80名様 但し、これを超えてもセミナーをご聴講いただくことは可能です。)
11/30(土)ららぽーとEXPOCITYにて開催される「2019大阪大学共創DAY@EXPOCITY」にブースを出展します
- 日時
- 2019年11月30日(土)11:00~17:00
- 場所
- 出展場所
-
1階「光の広場」
- 出展タイトル
-
タンパク質のかたちをみよう
- 内容
-
分子の立体視、ペーパーモデルなどを使って目に見えない小さなタンパク質の「かたち」にふれてみましょう。※当イベントで紹介する内容はPDBj一般向けサイト(PDBj入門 )でも提供しています。
[wwPDB] OneDepにEMマップの検証機能が追加されました
OneDepシステムで提供されるwwPDBの検証レポートに、電子顕微鏡法(EM)のマップの検証機能が追加され、データに間違いがある場合に、利用者が問題を確認しやすくなりました。
新しいOneDepシステムの検証レポートには、これまでEMDBページで使用されてきた様々なEMデータ検証方法を統合し、広範囲なEMマップの検証処理を組み込みました。 このEMの検証レポートは、当初はOneDepシステムへの登録者へのみ提供しますが、将来はPDBとEMDBアーカイブ全体のエントリーに対して提供する予定です。
PDB構造モデルとEMDBマップの重なり具合の解析が検証処理に含まれ、アミノ酸レベルでの残基プロパティプロット図や、マップとモデル全体を視覚的に重ね合わせた図で示されます(以下の画像を参照してください)。また、ハーフマップ又はFSCデータが登録された場合には、登録者による分解能と検証ソフトが提供する分解能を比較するために、レポートにFSC曲線も表示されます。
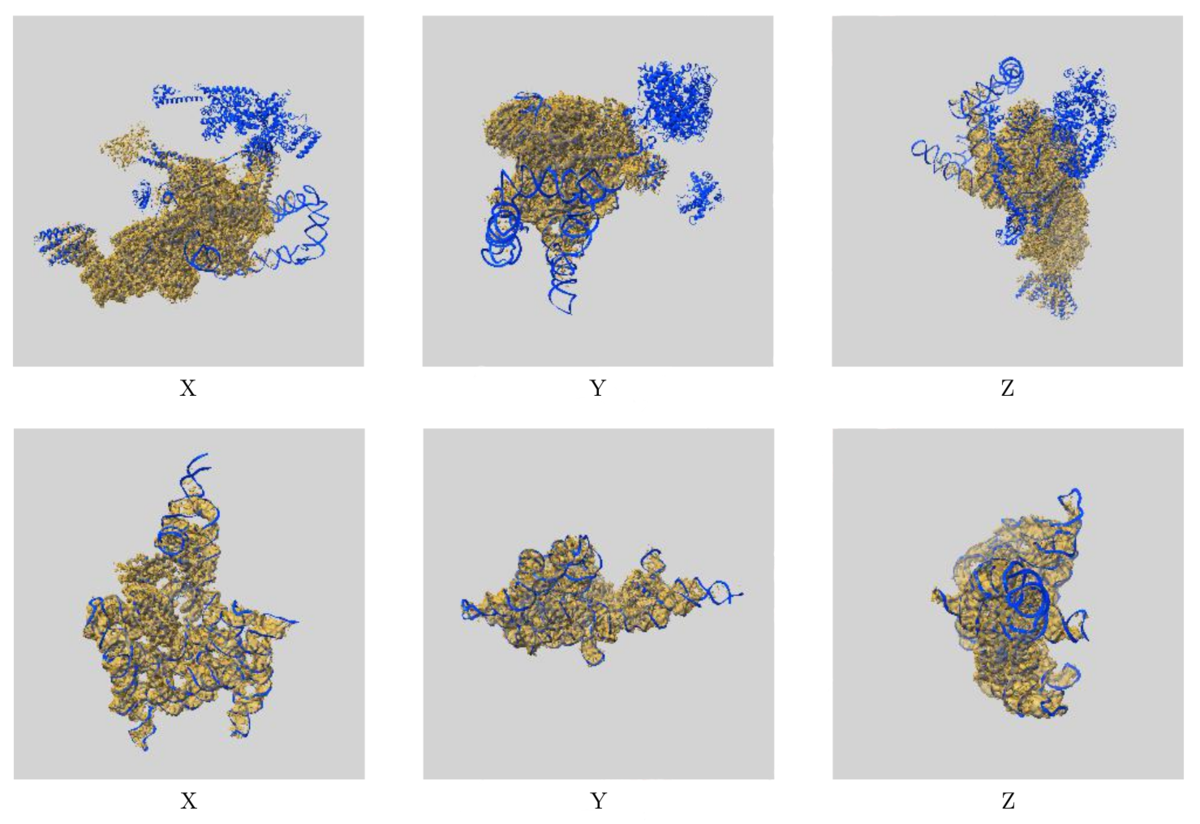 画像は、新しい検証レポートの一部ですが、EMマップと構造モデルの重なりを、視覚的に表しています。上側の図では、マップ(EMD-0360)とモデル(6N7P)の3つの直交方向の重ね合わせを表していますが、登録者による表面レベルではEMマップでカバーされないモデル領域が存在することがわかります。一方下側の図は、登録者による表面レベルで、モデル構造(6ME0)の大部分が、EMマップ(EMD-9105)とよく一致していることを示しています。
画像は、新しい検証レポートの一部ですが、EMマップと構造モデルの重なりを、視覚的に表しています。上側の図では、マップ(EMD-0360)とモデル(6N7P)の3つの直交方向の重ね合わせを表していますが、登録者による表面レベルではEMマップでカバーされないモデル領域が存在することがわかります。一方下側の図は、登録者による表面レベルで、モデル構造(6ME0)の大部分が、EMマップ(EMD-9105)とよく一致していることを示しています。
レポートには、直交投影像、中央断面図、マスクの視覚化などの画像が表示され、マップの詳細な検証結果と矛盾点を確認できます。 また、EMマップボリュームの統計分析には、マップの密度分布のグラフ、表面レベルによるボリューム評価、回転平均したパワースペクトルが含まれ、EMボリュームをさらに詳細に分析します。
新しく加わった検証情報によって、登録者とユーザの双方が、EMデータにエラーがある可能性を判断でき、PDBとEMDBの両方においてデータの制限が明確になります。
EMエントリーの検証レポートについて更に詳細な情報は、wwPDBのヘルプページをご覧ください。
検証レポートについてご質問などございましたら、validation@mail.wwpdb.orgまで、ご連絡ください。
[ wwPDB News ]
トーゴーの日シンポジウム2019
第9回 トーゴーの日シンポジウム2019にてポスター発表とワークショップを行いました。
- 場所
- 日本科学未来館(東京都江東区、アクセス)
- 日時
- 2019年10月5日(土)
- 参加費
- 無料
- ポスター
-
- 【32】蛋白質構造データバンク(PDBj)の高度化と統合的運用
-
タンパク質、核酸、糖鎖などの生体高分子の立体構造情報は Protein Data Bank(PDB)として集積され、2019 年 3 月には 15 万件を超える構造データを公開している。PDBj は、国際的組織である wwPDB の一員として、共通した品質管理によるデータ登録作業とダウンロード・サイトの運営および独自のサービス・ツールや二次 DB の提供、セマンティック化を実施している。また構造データの検証レポートについてもXML/RDF化し、PDBのメタデータだけではなく検証レポートの評価指標についても検索しやすくしている。本発表ではそれらを紹介する。
- 【33】PDB検証レポートの高度化とNMR統合ファイル検証機能の開発
-
PDBでは決定された生体高分子の立体構造の品質を評価する文書として、データの検証レポートを登録時に発行している。さらなる構造品質の向上のため、X線回折、核磁気共鳴、電子顕微鏡の各構造決定法分野の専門委員会(Validation Task Force)の提言を基に高度化した検証レポートを開発し、PDBデータベースの登録・アノテーション・検証を行うパイプライン・システム(OneDep)において提供を開始した。また、NMR実験データベース(BioMagResBank)を運営するPDBj-BMRBグループは、米ウィスコンシン大学との国際協力の下、NMR実験データベースの収集、登録、データ管理、評価と公開を行っている。2019年7月時点で13,000件を超えるNMR実験データに加え、SPARQLエンドポイントを公開し、生体系NMRコミュニティーに貢献してきた。NMRデータである化学シフト、距離制限情報およびピークリストを用いてPDB検証レポートの生成をおこなう。このデータから構成されるNEF/NMR-STAR V3.2のNMR統合ファイルの検証機能を開発しOneDepへの実装を行った。本発表ではこれらについての紹介を行う。
- ワークショップ:つないで使って役に立てようProtein Data Bank
-
蛋白質構造データバンク(Protein Data Bank:PDB)は、蛋白質、核酸、糖鎖など生体高分子の立体構造情報を集めたデータベースである。放射光施設の高度化、超高磁場NMR装置の普及や革新的な電子顕微鏡装置の開発により、PDBの登録件数は増加の一途をたどっている。2019年7月末時点でのPDB登録件数は15万件を優に超えている。PDBj(PDB Japan)は、国際組織であるworldwide PDB(wwPDB)の一員として、共通した品質管理によりアジア地区からのデータ登録を行ってきた。また、全PDBデータに関するダウンロード・サイトの運営や、横断的な検索サービス・ツール類を独自に開発するとともに、ライフサイエンス・データベースの統合的な利用を可能にするため、wwPDBでPDB関連データのRDF化を実施している。 PDB利用研究のうち、機能分子である蛋白質の理解を立体構造に基づいて加速させようとする研究の割合が、年々大きくなっている。本ワークショップでは、有用物質生産を目指して行われているPDBの活用例をご講演いただく。代謝工学をベースにしたPDBの活用例を大阪大学の清水先生に、分子工学をベースにした活用例を産総研の亀田先生にお願いしている。併せて、関連するPDBjの検索サービスのデモンストレーションを行い、エンドユーザーだけでなくDB開発者からもご意見をいただきたい。デモンストレーションでは、簡易検索、詳細検索、SQL検索やSPARQL検索などの実演を行う。
- PDBj Mine(PDB検索サービス)の紹介:工藤 高裕・横地 政志(大阪大学蛋白質研究所)
[wwPDB] PDBエントリーのDOI参照の仕組みを改良しました
wwPDBは、公開済みのPDBエントリーに紐づくデジタルオブジェクト識別子(DOIs, digital object identifiers)の仕組みを改良しました。 公開されたPDBエントリー各々に対して、wwPDBサイトの新しいランディングページを公開しました。 このページ上では、対応するPDBエントリーについての基本的な情報を表示し、座標や実験データ、検証レポートをwwPDBのFTPサイトからダウンロードするためのリンクを提供しています。 さらに重要なことに、PDB構造について更に発展した情報を参照できるよう、wwPDBパートナー各ウェブサイトへのリンクを張っています。 例えば、最近のエントリーのランディングページ(https://doi.org/10.2210/pdb6qw9/pdb)をご覧ください。 従来の仕組みでは、DOIsはPDBフォーマットファイルに紐づいていたことに比べて、大きな進歩となっています。
PDBエントリーの構造が発表されるオンライン版論文から、各エントリーごとに発行されるDOIへリンクを張ることで、科学雑誌が、これらランディングページとDOIを活用することをお勧めします。 wwPDBはまた、この機会を利用して、各PDB DOIに紐づくメタデータを更新し、DOI提供者(CrossRef)が提供するAPIを使って、直接その情報が得られるようにしました。
また、なじみがある4文字のPDBアクセションコードは近いうちに発行が尽きるので、徐々に導入されている新しい8文字形式のPDBアクセションコードもご活用ください。 新しいコードは、接頭語「PDB_」から始まり、8文字の英数字が続きますが、最後の4文字はなじみがある4文字のPDBアクセションコードと同一です。
[ wwPDB News ]
9/24(火)、日本生物物理学会年会にてPDBjランチョンセミナーを開催します
2019/9/24(火)、第57回日本生物物理学会年会にて下記の通りランチョンセミナーを開催します。
- 日時
- 2019/9/24(火)11:30 ~ 12:20
- 場所
- 宮崎県・シーガイアコンベンションセンター 会場:G会場(アイボリー)/ Room G (IVORY) アクセス
- 参加費
- 無料(別途年会参加費は必要)
- 参加方法
-
事前オンライン予約期間(8月27日10時 ~ 8月29日16時まで)に、年会のウェブサイトからお申込みください(抽選制)。
※当日の朝08:10頃~10:00まで「セミナー整理券」配布デスクを設置し、抽選にもれた方のうち先着10人の方へ昼食を年会事務局スタッフにて配布します。また、昼食なしでのご参加をご希望の方も、当日会場で入場の可否をお問い合わせください。 - 講演言語
- 英語
- 演題・演者
-
-
「PDBjとwwPDBの今後の活動方針について」(Recent activities of PDBj and wwPDB)
栗栖 源嗣(大阪大学蛋白質研究所) -
「構造データを編集するための新ツールについて」(New tools for editing and annotating structural data)
Gert-Jan Bekker(大阪大学蛋白質研究所)
-
「PDBjとwwPDBの今後の活動方針について」(Recent activities of PDBj and wwPDB)
- その他資料
9/10(火)、第8回生命医薬情報学連合大会(IIBMP2019)にてランチョンセミナーを開催します
日本バイオインフォマティクス学会 2019年年会・第8回生命医薬情報学連合大会(IIBMP2019)にて、下記の通りランチョンセミナーを開催します。
- 日時
- 2019/9/10(火)12:00 ~ 13:00
- 会場
- 東京工業大学 大岡山キャンパス くらまえホール アクセス
- 参加費
- 無料。ただし、別に学会への参加登録(有料)が必要です。
- 参加方法
- 事前の整理券配布などはありません。直接セミナー会場にお越しください。
- 講演言語
- 日本語
- 演題・演者
-
-
「PDBjでの分子検索・モデリングサービス」
川端 猛(大阪大学蛋白質研究所) -
「PDB検証レポートを利用したPDBj検索サービスの紹介」
横地 政志(大阪大学蛋白質研究所)
-
「PDBjでの分子検索・モデリングサービス」
- その他資料
8/18(日)第29回 青少年のための科学の祭典(サイエンスフェスタ)大阪大会 にブースを出展します
第29回 青少年のための科学の祭典(サイエンスフェスタ)大阪大会にブースを出展します。 PDBjの出展は8/18(日)のみです。
予定している主な出展内容は以下の通りです。
- タンパク質についての基礎的な解説
- タンパク質の形を立体視で体感してみる
- タンパク質の形を解明する方法についての解説
- 場所
- ハービスホール(大阪市北区、公式サイト、Google Map)
- 出展日時
- 2019年8月18日(日)10:00-17:00
- 参加費
- 無料
[wwPDB] OneDepでPDB IDを変更せずに、公開済みのPDBエントリーの座標を改善できます
PDBバージョニングシステムにより、公開済みPDBエントリーに対して、同じPDBアクセションコードを保持したまま座標の更新ができるようになりました。
登録者は既存のエントリーについて座標を差し替えできます。 当面はこの機能は、2014年に導入されたOneDepシステムから登録されたPDBエントリーに限定されます。 将来的には、レガシーシステム(ADITやAutodep等)で登録されたエントリーについても拡張する予定です。時期については今後発表する予定です。
登録者はまず、該当のエントリーについて、OneDepのコミュニケーションパネルから、変更したい旨を連絡する必要があります。 新しい座標の登録後、wwPDBのバイオキュレータが再処理を行い、新しいバージョンが公開されます。 PDBエントリーのバージョニングは、座標等の変更に限定され、登録済みの実験データへの変更は行えません。 座標の変更は、年間に1エントリー当たり1回まで、一人の研究責任者(PI)に対して3件のエントリーまで、という制限があります。
各エントリーの最新のバージョンは、メインPDBアーカイブのFTPサイト(ftp.pdbj.org [PDBj]/ ftp.wwpdb.org [wwPDB])で公開されます。 全てのメジャーバージョンのPDB構造ファイルは、バージョン化アーカイブ(ftp-versioned.pdbj.org [PDBj]/ ftp-versioned.wwpdb.org [wwPDB])に保管されます。 詳細は、wwPDBウェブサイトをご覧ください。 バージョン化アーカイブの構造は、将来のPDBアクセションコードの拡張を想定したものとなっています。 そのためPDBエントリー1abcのファイルは、pdb_00001abcというフォルダ内に配置されています。
バージョニング管理の中でエントリーに行われる変更は、「メジャー」又は「マイナー」のどちらかに分類されます。 原子座標やポリマー配列、化合物等の化学的な表現に更新があると、メジャーバージョンが一つ増えることになります。 他の変更は「マイナー」バージョンの更新になります。 バージョン化の中で行われた変更はすべて、PDBx/mmCIFファイルのauditカテゴリに記録されます。
PDBバージョニングについて何かご質問がございましたら、wwPDB(deposit-help@mail.wwpdb.org)まで、英語でお問い合わせください。
[ wwPDB News ]
[wwPDB] 結晶構造のPDBへの登録において、本日からPDBx/mmCIFフォーマットファイルが必須になりました
本日2019年7月1日より、結晶構造解析法によるPDBへの登録において、PDBx/mmCIFフォーマットファイルの登録が必須になりました。
X線、中性子、ファイバー、電子線回折法を含む、高分子の結晶構造解析によるPDB構造の登録に対して、OneDepでは、PDBx/mmCIFフォーマットのみが受け付けられます。 旧来のPDBフォーマットと比べて、PDBx/mmCIFフォーマットの登録では、登録過程が効率化され、またPDBx/mmCIFでサポートされる、実験的メタデータを広範囲に収集できるため、構造の検証を強化することにつながります。 10万個以上の原子や、2文字以上の鎖名を持つPDBエントリーは、既に、旧来のPDBフォーマットではサポートされません。 更に、2021年までには、PDBの化合物IDは4文字以上への拡張が必要になると予想され、その結果、PDBのコアアーカイブからPDBフォーマットファイルを廃止することにつながります。
RefmacやPhenix.refineやBusterなどのプログラムを利用すると、PDBx/mmCIFフォーマットファイルを出力できます。構造決定及び精密化のために他のソフトウェアを利用するユーザのために、wwPDBは、PDBフォーマットからPDBx/mmCIFフォーマットへ変換するためのスタンドアロンやウェブベースのツール(MAXITやpdb_extract)を提供しています。登録のためのPDBx/mmCIFフォーマットファイルの準備・出力に関する詳細情報は、wwPDBサイトからご覧ください。
PDBx/mmCIFワーキンググループは、PDBx/mmCIFデータモデルに取り組んできました。 PDBx/mmCIFはまた、Jmol/JSMol, Chimera, OpenRasMol, CCP4MG, COOT, PyMOL, VMD, Molmil, LiteMol, Mol*, NGL などの可視化アプリケーションによってサポートされています。 加えて、Protein Model PortalやSASBDBのような他のデータリソースも、PDBx/mmCIFフレイムワークをデータの表現のために採用し、拡張しました。
ご質問などございましたら、wwPDB(deposit-help@mail.wwpdb.org)まで、英語でお問い合わせください。
[ wwPDB News ]
6/26(水)蛋白質科学会年会@神戸にてPDBjランチョンセミナーを開催します
2019/6/26(水)、第19回日本蛋白質科学会年会にて下記の通りランチョンセミナーを開催します。
- 日時
- 2019/6/26(水)12:55-13:45
- 場所
- 神戸国際会議場(兵庫県神戸市中央区港島中町6丁目9−1)アクセス・Google Map E会場(5F 501)
- 参加費
- 無料(別途年会参加費は必要)
- 参加方法
- セミナーへのご参加自体については、学会に参加されていれば特に追加の手続きはありません。 開始時間前になりましたら直接会場にお越しください。 入場は先着順です。満席となりました場合はご容赦ください。 昼食は当日8:30より総合受付で配布する整理券をあらかじめお受け取りになってお持ちください。 整理券と引き換えに昼食をお渡しします。
- 発表言語
- 日本語
- 演題・演者・内容
-
(1)
演題:「PDBjとwwPDBの今後の活動方針について」(Activity plan of PDBj and wwPDB)
演者:栗栖 源嗣(大阪大学蛋白質研究所)
PDBj(https://pdbj.org/)は,worldwide PDB(wwPDB、https://wwpdb.org/)のメンバーとしてアジア・中東地区で決定された蛋白質等の生体高分子の構造を登録し、国際的に協調して座標と実験データを全世界へ無償で公開しています。 今回のセミナーでは、1) wwPDBが管理するコアアーカイブ(PDB、BMRB、EMDB)の今後の運営体制(RCSB-PDB、PDBe、PDBj、BMRB、EMDB)とデータ検証について、 2) PDBx/mmCIFフォーマットを登録時の必須フォーマットとする方針変更、3)OneDep登録サーバー利用時の注意点について紹介します。さらに2018年wwPDB運営諮問委員会で議論されたORCID iDに関する今後の活用方針など最近の活動を紹介します。
-
(2)
演題:「wwPDB検証レポートの利用法とPDB/BMRB登録システムの紹介」(Introduction of PDB/BMRB deposition system along with usage of wwPDB validation reports)
演者:横地 政志(大阪大学蛋白質研究所)
PDBアーカイブの検証レポート(wwPDB validation reports)は、X線回折、核磁気共鳴、Cryo電子顕微鏡による構造決定法の各専門委員会(Validation Task Force)の意見に基づき、広く受け入れられている評価基準を使って、PDB登録された立体構造モデルの品質を評価する文書です。wwPDBは、新しい立体構造を扱う論文の投稿・査読過程において、検証レポートの利用を推奨しています。 PDBjでは、検証レポートに含まれる評価指標の検索を容易にするため、機械処理しやすい検証レポートの代替フォーマットによるアーカイブ(PDBx/mmCIFと高い互換性のあるXML版とRDF版)を開発しました。生成したアーカイブを利用して、検証レポートのリレーショナルデータベースおよびSPARQLエンドポイントを公開しています。 PDB登録システム(OneDep)と検証レポートは深く結びついており、検証項目の拡充が予定されています。また、従来のADIT-NMRを代替する新BMRB登録システム(BMRBDep)の開発も進んでいます。検証レポートの利用法を中心にして、PDB及びBMRBの登録システムの進展について紹介します。
[wwPDB] wwPDBの検証レポートを改良し、リガンドの検証結果と電子密度マップを可視化しました
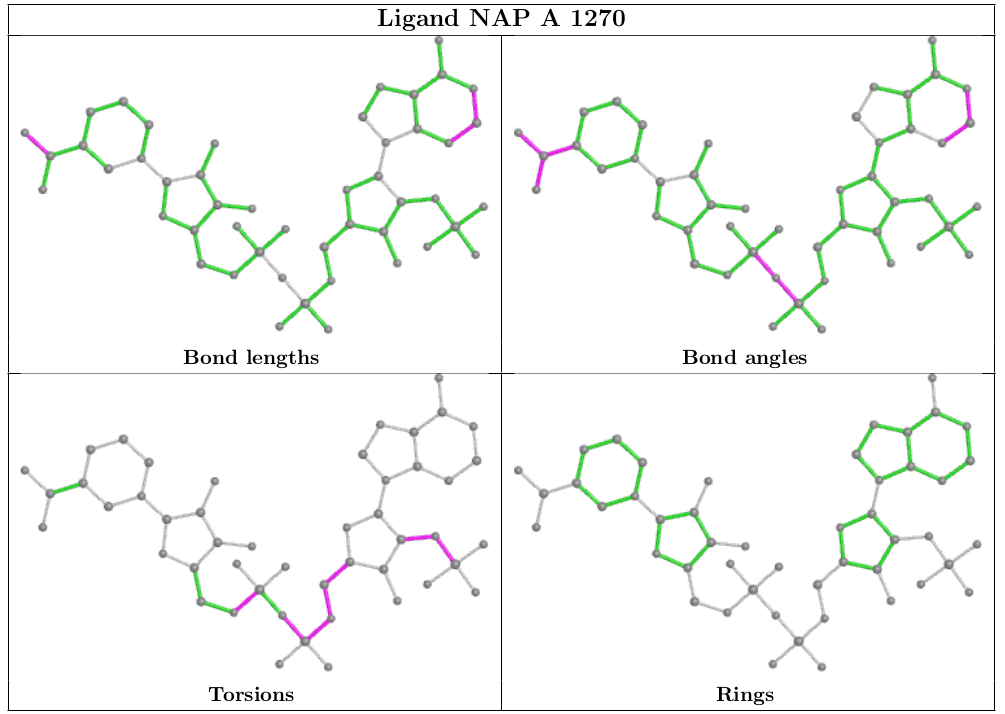
wwPDBが最近行った検証レポートの改良により、リガンドの検証情報が非常に明確になりました。
改良した検証レポートには、リガンドの幾何学的な検証を強調表示した2次元平面図と、結晶解析構造の場合には、電子密度の3次元表示図が含まれます。
また、検証レポートの解析に使用された電子密度マップの係数を登録者に提供します。
リガンドの検証
wwPDBは、Global Phasing社と協力して、wwPDB/CCDC/D3Rリガンド検証ワークショップによって提案されたように、 buster-report のリガンド可視化情報をwwPDBの検証レポートに組み込みました。 このリガンド可視化情報は、登録者が「Ligand of Interest(注目するリガンド)」として選択したリガンドか、分子量が250ダルトンより大きく、かつ構造化学的に標準値から外れた値を持つリガンドに対して表示されます。
以下のリガンドNAPは、構造化学とX線データとの一致度の双方の視点において、よくある例として意図的に選択して例示しています。
CCDC Mogulによる構造化学的な評価は、以下に示すようにリガンドの2次元平面図上の色で強調表示されます。
リガンドの幾何学的な検証情報が追加されたことに加え、X線結晶構造のPDBエントリーに対しては、 リガンドとその周辺の電子密度マップを表す画像が、wwPDBの検証レポートに追加表示されています。
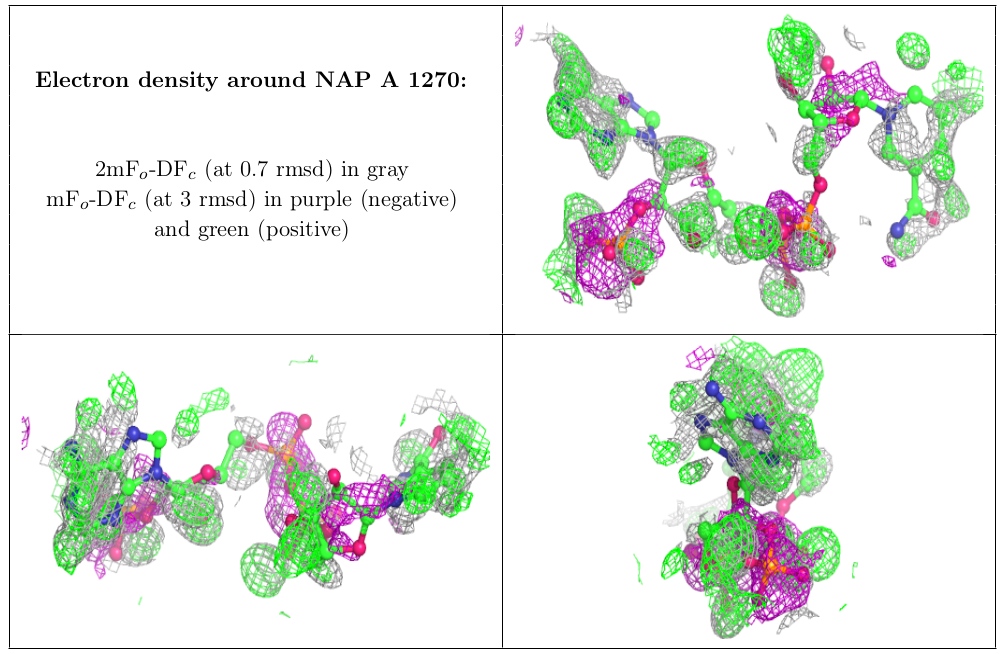
電子密度マップ係数ファイル
今回の更新により、wwPDBは検証レポートに加えて、検証の過程で作成される電子密度マップの係数(2mFo-DFc と mFo-DFc)を登録者に提供します。 wwPDBの検証レポートのために生成された電子密度マップの係数は、新しいエントリーが公開される際にPDBアーカイブに追加され、 既存のエントリーに対しては、全エントリーの検証レポートを再作成する際に、PDBアーカイブに追加する予定です。
この検証パイプラインへの変更によって、 登録者が提供された検証情報を解釈しやすくなることを願っています。 ご質問などございましたら、wwPDB(deposit-help@mail.wwpdb.org)まで、英語でお問い合わせください。
[ wwPDB News ]
[wwPDB] Michael G. Rossmann先生を追悼して
この度、Michael G. Rossmann先生ご逝去の知らせを聞いて、wwPDB一同悲しみに包まれるとともに、Rossmann先生のご家族ならびにご友人の方々に心よりお悔やみ申し上げます。 Rossmann先生は、構造生物学の分野で大きな貢献をなさり、また、wwPDBとその活動を支援してくださいました。Rossmann先生への感謝とともにPDBアーカイブへのRossmann先生の貢献について共有したいと思います。
1977年から2019年の間で、Rossmann先生はPDBエントリーを300件以上登録されているという、構造生物学において類い稀な経歴の持ち主です。Rossmann先生はまた、分子置換法開発の基盤を確立しておられますが、この手法はX線結晶学における位相決定の幅を広げ、これまでに約10万件のPDBエントリーの構造決定に使用されてきました。Rossmann先生の重要な発見の一つは、ジヌクレオチド補因子へ結合する酵素に共通してみられる構造モチーフ、有名なロスマンフォールドの発見です。その重要性は、ロスマンフォールドモチーフを含むPDBエントリーが約2万件存在するという事実(それは、PDBアーカイブでの彼の永遠の財産でもあります)によって強調されます。
Rossmann先生はまた、ウイルス学の分野で非常に大きな貢献をなさり、彼が解いたウイルス構造は数百にものぼります。この中には、一般的な風邪としても知られる、ヒトライノウイルスの最初の構造が含まれています。Rossmann先生の研究によって、ウイルスの構造と機能についての理解が深まり、ウイルス感染を治療し予防するための新しい治療学の開発へとつながっています。
Rossmann先生はまた、2009年から2012年までの4年間、wwPDB諮問委員会のメンバーであり、RCSB PDBとEMDataBankの諮問委員会にも参加していました。この間、Rossmann先生はwwPDBへ多くの助言と指導を与えてくださり、この4年間と将来のアーカイブ活動を形作ることにつながっています。RCSB PDBの元代表であるHelen Berman氏は、「1970年代初頭のProtein Data Bank開始以来、Rossmann先生は、PDBの積極的な支持者でした」と強調します。Protein Data Bank Japan (PDBj) の元代表である中村春木氏は、「wwPDB 諮問委員会ではwwPDBの活動に対するRossmann先生の思慮深い発言・忠告にいつも感銘を受けておりました。」「彼は、実験の生データを蓄積することの重要性を理解しており、データ科学の振興に何年も前から言及されていました。」と語ります。
Protein Data Bank in Europe (PDBe)の元代表であるGerard Kleywegt氏は、Rossmann先生を「人生と科学に対し非常に強い熱意をもった人で、誰とでも仲良くできる人でした」と振り返ります。Kleywegt氏はまた、ここ数か月でさえも「彼は自分でデータ収集実験をしていたんです」と強調し、Rossmann先生が助成金不採用の知らせに落胆し、「科学的に優れた申請なのに(おそらく80歳という高齢を理由に)助成金不採択となったことは、理解できなかったんでしょう」と振り返ります。
Berman氏が「Rossmann先生は、構造生物学への貢献において本当にかけがえのない人で、彼のなさったこと全てに永遠に感謝したい」と述べているように、我々wwPDBは、wwPDBと構造生物学研究全体へのRossmann先生の支援に深く感謝いたします。
[ wwPDB News ]
6/20(木)・21(金)に、CiCLE:クライオ電顕単粒子解析 データ処理講習会を開催します(5/24(金)締め切り)
CiCLE: クライオ電顕単粒子解析 データ処理講習会
開催趣旨:近年のクライオ電子顕微鏡の単粒子解析の進展は著しく、X線結晶解析に迫る分解能の生体高分子像が得られるようになってきています。 本講習会では、電顕の初心者の方を対象に、電顕画像取得後のデータ処理のノウハウを、実際に計算機を使った講習会形式で体験していただきます。 日本電子のクライオ電子顕微鏡CRYO ARMで撮影された画像を題材として、3次元画像再構成、原子モデルの作成の講義・講習を行います。
- 主催
- 医療研究開発革新基盤創成事業(CiCLE) 「タンパク質構造解析のハイスループット化へ向けた装置開発」
- 日時
- 2019年6月20日(木)、21日(金)9:00~17:30
- 場所
- 大阪大学 蛋白質研究所 本館7階セミナー室・構造解析研究棟3階ワークステーション室
- 参加費
- 無料
- 申し込み
- 参加申し込みフォームよりお申込みください。
- 申し込み締め切り日時
-
2019年5月24日(金) 17:00 ※参加申し込み者多数のため、締め切りを早めました。
2019年6月13日(木)17:00 ※参加希望者多数の場合、早めに締め切る場合もございます。 - 定員
-
約10名
※受講希望者が定員を超えた場合は、受講希望理由・申し込み順などを参考にして、 こちらでセレクションさせていただきます。実際に電顕の使用を予定している方、 電顕のデータ処理の研究を考えている方を優先いたします。 - 講義・講習の担当
- 川端 猛(大阪大学・蛋白質研究所)、加藤 貴之(大阪大学大学院・生命機能研究科)、 田中秀明(大阪大学・蛋白質研究所)、栗栖 源嗣(大阪大学・蛋白質研究所)、難波 啓一(大阪大学大学院・生命機能研究科)
- 講習の内容
- 日本電子 CRYO ARMで撮影された2Dの顕微鏡画像群を用いて、単粒子解析の3次元画像再構成と原子モデリングの講習を行います。3次元再構成は、Relion を用いて、チュートリアルに基づいて行います。原子モデリングは、UCSF Chimera、gmfit、Phenix、COOTを用いて行います。
- 実行計算機環境について
- 演習に必要な計算機はこちらで全て準備します。ご自分のPCを持参していただく必要はありません。 3次元再構成は、一人1台のWindowsノートPCから、GPUマシンに遠隔ログインして実行します。 原子モデリングは、二人一組で1台のワークステーションを使用して行う予定です。 GPUマシン、ワークステーションのOSはLinuxです。
- お問い合わせ
- PDBjのお問い合わせページ をご利用ください。
<プログラム>
2019年6月20日 (木)
-
講義 (本館7階セミナー室)
- 9:15-9:30:
- 講習会の趣旨の説明(栗栖、難波)
- 9:30 - 10:50:
- 単粒子解析の理論の概要(加藤)
- 11:00- 12:00:
- Relionの背景にあるベイズ理論(川端)
-
実習 (本館7階セミナー室)
- 13:30 - 17:00:
- Relionを用いた単粒子解析 I(加藤、川端)
(動画補正、CTF推定、粒子ピッキング、2D分類、初期3Dマップ)
2019年6月21日 (金)
-
実習 (本館7階セミナー室)
- 9:30 - 10:30 :
- Relionを用いた単粒子解析 II(加藤、川端)
(初期3Dマップの作製、3D分類による精密化)
-
講義(本館7階セミナー室)
- 11:00 - 12:00:
- 剛体フィッティングによるモデリング法(川端)
-
講義(本館7階セミナー室)
- 13:30 - 14:30 :
- デノボモデリングと精密化計算(田中)
-
実習(構造解析研究棟 3階ワークステーション室)
- 15:00 - 16:00 :
- UCSF Chimeraとgmfitを用いた剛体フィッティング(川端)
- 16:00 - 17:30 :
- COOTとPhenixによるデノボモデリングと精密化計算(田中)
[wwPDB] PDBx/mmCIFフォーマットファイル必須化についての論文が、Acta Cryst Dから出版されました
結晶構造をPDBへ登録する際にPDBx/mmCIFフォーマットファイルが必須になることをActa Crystallographica Section D (Acta Cryst. D)に発表しました。 著者には、wwPDBの各サイトのメンバーに加えて、主要な結晶構造精密化ソフトウェアの代表も含まれています。
Acta Cryst. Dから出版された論文“Announcing mandatory submission of PDBx/mmCIF format files for crystallographic depositions to the Protein Data Bank (PDB)” では、2019年7月1日以降、X線、中性子、ファイバー、電子線回折法を含む、結晶構造解析法(MX)によって解かれた構造をPDBへ登録する際に、OneDepで受け付けるモデル構造ファイルのフォーマットは、PDBx/mmCIFフォーマットのみとなることを強調しています。
Refmac(CCP4)やPhenix.refineやBusterなどのプログラムを利用すると、PDBx/mmCIFフォーマットファイルを出力できます。 各ソフトウェアの代表との共著とすることで、PDBx/mmCIFデータモデルに対する結晶学ソフトウェアのコミュニティの関わりを強調しています。 旧来のPDBフォーマットと比べて、PDBx/mmCIFフォーマットの登録では、登録過程が効率化され、またPDBx/mmCIFでサポートされる実験的メタデータを広範囲に収集できるため、構造の検証を強化することにつながります。
登録のために必要なPDBx/mmCIFフォーマットファイルの出力や準備についての情報は、wwPDBウェブサイトをご覧ください。
[ wwPDB ニュース ]
[wwPDB] 公募: 中国蛋白質構造データバンク(PDBc)の代表
蛋白質構造データバンクの中国における新たなアーカイブ拠点「中国蛋白質構造データバンク」(Protein Data Bank China: PDBc)を設けるにあたり、当拠点の統括責任者を公募します。 この職位は、中国の国立蛋白質科学研究センター上海(NCPSS)、上海科技大学 iHuman研究所、同大学 免疫化学研究所(SIAIS)の兼任となります。採用者は、国際蛋白質構造データバンク(wwPDB)の中心メンバー(米国のRCSB PDB、BMRB、欧州のPDBe、日本のPDBj)と協力し、PDBcがwwPDBのメンバーとなる要件を満たすよう活動することを求められます。
[ wwPDB ニュース ]
[wwPDB] PDBのエントリー数が15万件を突破しました
今週の公開で、PDBアーカイブのエントリ数は15万件に達し、総数は150,145 となりました。
実験で決定したタンパク質や核酸の膨大な構造データを、世界中の構造生物学者が登録してくださったおかげで、1971年に創設されたPDBアーカイブは大きな節目を迎えることができました。このアーカイブは、世界中の研究者が無償で利用できます。
wwPDBを構成する4つのデータセンターでは、生体高分子の三次元構造のオンラインアクセスを提供しています。これらPDBのサービスは臨床医学、農業、基礎生物学において、病気や健康に関わるタンパク質から生物エネルギー変換反応に至るまでさまざまな事象を研究者が理解することにつながっています。 また、PDBアーカイブは各エントリーに関連する190万以上のファイルを収容しており、データ量は512ギガバイトを超える容量となっています。
PDBアーカイブは、世界結晶年である2014年に10万件に到達しました。この記録が打ち立てられて以来、PDBの登録数は急増しデータの複雑さも急速に増加しています。 データ急増への対応は、各実験手法に対応したPDBデータの登録、検証、編集を行う世界共通のシステム「OneDep」を立ち上げたことが支えになっています。 「OneDep」システムとその基本フォーマット「PDBx/mmCIF」があれば、構造生物学の発展によってもたらされた課題に対処するため、今後、適切にPDBアーカイブを修正していくことができます。 これまでに41,000以上の構造が「OneDep」を使って登録、編集、検証され、PDBアーカイブで公開しています。またアーカイブの整合性を保つため、さらに多くのエントリーをOneDepで更新しました。
今週の公開で、262個の新しい構造がアーカイブに追加されます。これらの構造は、他の構造ともに、基礎生物学、創薬、生物エネルギーの研究や教育に不可欠なものとなります。 発足当初からアーカイブのサイズは、10-15年ごとにおおよそ10倍に増加しています。PDBは、1982年に100件、1993年には1000件に、2000年には10,000件に到達しました。 そして今や15万件目の構造が利用できるようになりました。現在アーカイブにある構造の半分以上が、この10年間で公開されたものです。
我々研究者は、次なる15万件の構造とそれらの新しいデータがもたらす膨大かつ貴重な知識に対し大きな期待を寄せています。 しかし、PDBに登録される構造の件数が増加し、大きさや複雑さが増していることに加え、ハイブリッド構造決定法が登場したことで、それに対応した構造データの記述や管理が新しい課題となっています。 wwPDBではこれからも研究者コミュニティーと協力しながら、これらの課題に取り組み、品質、完全性、整合性において可能な限り高い水準を維持していきます。
PDBアーカイブとwwPDBの組織、それに開発経緯と今後の方針については、PDBアーカイブに関して述べた新たな参考文献に書かれています。主な文献を以下に挙げますのでご参考ください。
- Protein Data Bank: the single global archive for 3D macromolecular structure data (Nucleic Acids Res., 2019)
- Protein Data Bank (PDB): The Single Global Macromolecular Structure Archive (Methods in Molecular Biology, 2017)
- How community has shaped the Protein Data Bank (Structure, 2013)
- Creating a Community Resource for Protein Science (Protein Science, 2012)

[ wwPDB ニュース ]
[wwPDB] 結晶構造解析法によるPDBへの登録において、PDBx/mmCIFフォーマットファイルが必須になります
2019年7月1日より、結晶構造解析法によるPDBへの登録において、PDBx/mmCIFフォーマットファイルの登録が必須になります。
2019年7月1日からは、X線、中性子、ファイバー、電子線回折法を含む、高分子の結晶構造解析によるPDB構造の登録に対して、OneDepでは、PDBx/mmCIFフォーマットのみが受け付けられるようになります。 旧来のPDBフォーマットと比べて、PDBx/mmCIFフォーマットの登録では、登録過程が効率化され、またPDBx/mmCIFでサポートされる、実験的メタデータを広範囲に収集できるため、構造の検証を強化することにつながります。10万個以上の原子や、2文字以上の鎖名を持つPDBエントリーは、既に、旧来のPDBフォーマットではサポートされません。更に、2021年までには、PDBの化合物IDは、4文字以上への拡張が必要になると予想され、その結果、PDBのコアアーカイブからPDBフォーマットファイルを廃止することにつながります。
RefmacやPhenix.refineやBusterなどのプログラムを利用すると、PDBx/mmCIFフォーマットファイルを出力できます。 構造決定及び精密化のために他のソフトウェアを利用するユーザのために、wwPDBは、PDBフォーマットからPDBx/mmCIFフォーマットへ変換するためのスタンドアロンやウェブベースのツール(MAXITやpdb_extract)を提供しています。 登録のためのPDBx/mmCIFフォーマットファイルの準備・出力に関する詳細情報は、wwPDBサイトからご覧ください。
PDBx/mmCIFワーキンググループは、PDBx/mmCIFデータモデルに取り組んできました。PDBx/mmCIFはまた、Jmol/JSMol, Molmil, Chimera, OpenRasMol, CCP4MG, COOT, PyMOL, VMDを含む可視化アプリケーションによってサポートされています。加えて、Protein Model PortalやSASBDBのような他のデータリソースも、PDBx/mmCIFフレイムワークをデータの表現のために採用し、拡張しました。
[ wwPDB ニュース ]
[wwPDB] 研究公正局(ORI)による判断に従い、PDBエントリーが廃止され、論文も取り消されました
2018年に、米国保健福祉省(U.S. Department of Health and Human Services)の研究公正局(Office of Research Integrity, ORI)は、 H.M. Krishna Murthy事件の最終的な調査で、研究不正行為があったと発表しました。 Murthy氏は、10件の研究論文と、それらに対応する12件のPDB構造において、偽造及び/又は捏造した研究を報告したことが判明しました 。 研究公正局がこの問題で証拠を集めて評価している間に、 Murthy氏の5件のPDB構造は、4件の研究論文の撤回に対応するwwPDBの方針に従って、すでに廃止(obsolete)されました。 2018年4月23日に受け取ったORIからの正式な要請に応じて、Murthy氏の残りの7つのPDB構造は、wwPDBの方針に従って再び廃止されました。 ORIの調査は、研究上の不正行為で非難された個人に対し、適正な手続きを行うことを目的としており、厳格な機密保持が維持されています。 研究不正行為の最終的な調査結果だけが公表されています。
2009年12月に、アラバマ大学バーミンガム校(UAB)は、 当時、研究責任者(PI)とUAB雇用者という立場で発表した、H.M. Krishna Murthy氏のPDBエントリー12件と 10件の関連出版物を取り下げる予定であることを発表しました。 UABの要請に応じてwwPDBがレビューを行い、wwPDBの方針に従って、構造のうちの5つは、関連する文献の撤回時に廃止されました。 UABの要請に応じてwwPDBがレビューを行い、wwPDBの方針に従って、残りの7件の構造は、ORIからの要請を受けた際に廃止されました。
それ以来、7つの構造に関連した5つの論文が、学術誌から取り消されました。 この事件の詳細なPDBの経緯は、wwPDBサイトをご覧ください。
- RETRACTED: Crystal structure of a complement control protein that regulates both pathways of complement activation and binds heparan sulfate proteoglycans (2001) Cell 104, 301-311 doi: 10.1016/S0092-8674(01)00214-8
- Retraction of "Structures of Apolipoprotein A-II and a Lipid-Surrogate Complex Provide Insights into Apolipoprotein-Lipid Interactions" (2018) Biochemistry 57, 6044 doi: 10.1021/acs.biochem.8b00956
- Retraction for Ganesh et al., Structure of vaccinia complement protein in complex with heparin and potential implications for complement regulation (2018) Proc Natl Acad Sci USA 115, E6965 doi: 10.1073/pnas.1806429115
- Retraction of "Structural Basis for Antagonism by Suramin of Heparin Binding to Vaccinia Complement Protein" (2018) Biochemistry 57, 6043 doi: 10.1021/acs.biochem.8b00955
- Retraction for Ajees et al., Crystal structure of human apolipoprotein A-I: Insights into its protective effect against cardiovascular diseases (2018) Proc Natl Acad Sci USA 115, E6966 doi: 10.1073/pnas.1806430115
構造の検証と、アーカイブデータのリソースとしてのPDBの役割
PDBは、構造モデルとその関連実験データを保存し、アノテーションを行い、配布するアーカイブリソースです。 wwPDBは、X線(2008年)、NMR(2009年)及び、Cryo-EM(2010年)(EMDataBankと協力)に対して、 検証に関する専門委員会(Validation Task Forces)を招集し、 構造が登録された際に、構造(モデル構造、実験データ、モデルとデータの適合)を検証する際に使用する最も適切な評価基準について、助言を行っています。 検証に関する専門家委員会の提言は、PDB構造の登録、アノテーション及び検証を行う、wwPDBのOneDepシステムの一部に取り入れられてきました。
wwPDBの検証手続きの結果は、検証レポートに取り込まれます。 このレポートは、登録者に提供され、対応する論文が投稿される際に登録者から学術誌にも提出されます。 このような報告書が利用できることで、構造的データの信頼性の評価や、学術誌の編集者と査読者によるその解釈が、同じように非常に容易になっています。 wwPDBは、生体高分子の構造データを扱う学術誌に、投稿論文と一緒にwwPDBの検証レポートの提出を求めるよう、要請しています。 wwPDBパートナーの継続的な使命は、国際的な構造生物学コミュニティの支援を受けて、構造アーカイブを完全に守り、品質を向上させることです。
以下の文献もご覧ください。
- Case Summary: Murthy, Krishna H.M. (2018) US Department of Health and Human Services, The Office of Research Integrity
- Findings of Research Misconduct Notice 2018-07782 (2018) Federal Register 83: 16370
- Berman, H. M., G. J. Kleywegt, H. Nakamura, J. L. Markley and S. K. Burley (2010). "Safeguarding the integrity of protein archive." Nature 463(7280): 425 doi: 10.1038/463425c.
- Baker, E. N., Z. Dauter, H. Einspahr and M. S. Weiss (2010). "In defence of our science--validation now!" Acta Crystallogr D Biol Crystallogr 66(Pt 2): 115 doi: 10.1107/S1744309110001326
[ wwPDB ニュース ]
2019/1/28(月)All-in-one 合同講習会 2018/統合データベース講習会:AJACS 番町2 にてPDBjからも講演します
2019/1/28(月)、東京都千代田区で開催される「All-in-one 合同講習会 2018/統合データベース講習会:AJACS 番町2」にて、 PDBjからも講演を行います。詳細は以下の通りです。
- 日時
- 2019/1/28(月)10:15-16:00
- 場所
- 科学技術振興機構東京本部別館1階ホール(東京都千代田区五番町7 Kʼs五番町)アクセス・Google Map
- 参加費
- 無料
- 締め切り
- 2019/1/25(金)12:00
- 申し込み
- 申し込みフォームからお願いします。
- 演題・演者・時間・内容
-
「タンパク質構造データバンク・PDB の使い方」鈴木 博文(早稲田大学先進理工学部):13:50-14:50
立体構造解析や構造インフォマティクスについての専門外の方から初学者を対象とし、PDB のデータの解説や PDBj が提供するツールの紹介をします。
※他の講演も含む全体の情報については公式サイトをご覧ください。
- その他
- 当講演は講義形式の講習です。PCは使用しません。








