+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6x6p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
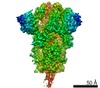
| タイトル | Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis | |||||||||
 要素 要素 | Spike glycoprotein スパイクタンパク質 スパイクタンパク質 | |||||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  coronavirus (オルトコロナウイルス亜科) / coronavirus (オルトコロナウイルス亜科) /  SARS-CoV-2 (SARSコロナウイルス2) / SARS-CoV-2 (SARSコロナウイルス2) /  SARS-CoV (SARSコロナウイルス) / SARS-CoV (SARSコロナウイルス) /  spike glycoprotein (スパイクタンパク質) / spike glycoprotein (スパイクタンパク質) /  fusion protein (融合タンパク質) / trimer fusion protein (融合タンパク質) / trimer | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / entry receptor-mediated virion attachment to host cell / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.22 Å クライオ電子顕微鏡法 / 解像度: 3.22 Å | |||||||||
 データ登録者 データ登録者 | Herrera, N.G. / Morano, N.C. / Celikgil, A. / Georgiev, G.I. / Malonis, R. / Lee, J.H. / Tong, K. / Vergnolle, O. / Massimi, A. / Yen, L.Y. ...Herrera, N.G. / Morano, N.C. / Celikgil, A. / Georgiev, G.I. / Malonis, R. / Lee, J.H. / Tong, K. / Vergnolle, O. / Massimi, A. / Yen, L.Y. / Noble, A.J. / Kopylov, M. / Bonanno, J.B. / Garrett-Thompson, S.C. / Hayes, D.B. / Brenowitz, M. / Garforth, S.J. / Eng, E.T. / Lai, J.R. / Almo, S.C. | |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2020 ジャーナル: bioRxiv / 年: 2020タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis. 著者: Natalia G Herrera / Nicholas C Morano / Alev Celikgil / George I Georgiev / Ryan J Malonis / James H Lee / Karen Tong / Olivia Vergnolle / Aldo B Massimi / Laura Y Yen / Alex J Noble / ...著者: Natalia G Herrera / Nicholas C Morano / Alev Celikgil / George I Georgiev / Ryan J Malonis / James H Lee / Karen Tong / Olivia Vergnolle / Aldo B Massimi / Laura Y Yen / Alex J Noble / Mykhailo Kopylov / Jeffrey B Bonanno / Sarah C Garrett-Thomson / David B Hayes / Robert H Bortz / Ariel S Wirchnianski / Catalina Florez / Ethan Laudermilch / Denise Haslwanter / J Maximilian Fels / M Eugenia Dieterle / Rohit K Jangra / Jason Barnhill / Amanda Mengotto / Duncan Kimmel / Johanna P Daily / Liise-Anne Pirofski / Kartik Chandran / Michael Brenowitz / Scott J Garforth / Edward T Eng / Jonathan R Lai / Steven C Almo /  要旨: Coronavirus disease 2019 ( ) is a global health crisis caused by the novel severe acute respiratory syndrome coronavirus 2 ( ), and there is a critical need to produce large quantities of high- ...Coronavirus disease 2019 ( ) is a global health crisis caused by the novel severe acute respiratory syndrome coronavirus 2 ( ), and there is a critical need to produce large quantities of high-quality SARS-CoV-2 Spike ( ) protein for use in both clinical and basic science settings. To address this need, we have evaluated the expression and purification of two previously reported S protein constructs in Expi293F and ExpiCHO-S cells, two different cell lines selected for increased expression of secreted glycoproteins. We show that ExpiCHO-S cells produce enhanced yields of both SARS-CoV-2 S proteins. Biochemical, biophysical, and structural ( ) characterization of the SARS-CoV-2 S proteins produced in both cell lines demonstrate that the reported purification strategy yields high quality S protein (non-aggregated, uniform material with appropriate biochemical and biophysical properties). Importantly, we show that multiple preparations of these two recombinant S proteins from either cell line exhibit identical behavior in two different serology assays. We also evaluate the specificity of S protein-mediated host cell binding by examining interactions with proposed binding partners in the human secretome. In addition, the antigenicity of these proteins is demonstrated by standard ELISAs, and in a flexible protein microarray format. Collectively, we establish an array of metrics for ensuring the production of high-quality S protein to support clinical, biological, biochemical, structural and mechanistic studies to combat the global pandemic caused by SARS-CoV-2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6x6p.cif.gz 6x6p.cif.gz | 575.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6x6p.ent.gz pdb6x6p.ent.gz | 460.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6x6p.json.gz 6x6p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x6/6x6p https://data.pdbj.org/pub/pdb/validation_reports/x6/6x6p ftp://data.pdbj.org/pub/pdb/validation_reports/x6/6x6p ftp://data.pdbj.org/pub/pdb/validation_reports/x6/6x6p | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  22078MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10433 (タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis EMPIAR-10433 (タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic AnalysisData size: 484.3 Data #1: Unaligned multi frame micrographs of SARS CoV-2 SPIKE protein produced in CHO cells [micrographs - multiframe] Data #2: Processed data - aligned frame sum with dose-weighting [micrographs - single frame]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper:
|
- 要素
要素
| #1: タンパク質 |  スパイクタンパク質 / S glycoprotein / E2 / Peplomer protein / SARS-CoV-2 spike glycoprotein スパイクタンパク質 / S glycoprotein / E2 / Peplomer protein / SARS-CoV-2 spike glycoprotein分子量: 140824.406 Da / 分子数: 3 / 変異: R682G R683S R685S K986P V987P / 由来タイプ: 組換発現 由来: (組換発現)   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)遺伝子: S, 2 発現宿主:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ)参照: UniProt: P0DTC2 #2: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose  オリゴ糖 / 分子量: 424.401 Da / 分子数: 12 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 424.401 Da / 分子数: 12 / 由来タイプ: 組換発現#3: 糖 | ChemComp-NAG /  N-アセチルグルコサミン N-アセチルグルコサミン研究の焦点であるリガンドがあるか | N | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Severe acute respiratory syndrome coronavirus 2 SARSコロナウイルス2 SARSコロナウイルス2タイプ: VIRUS / Entity ID: #1 / 由来: RECOMBINANT | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | |||||||||||||||
| 由来(組換発現) | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) | |||||||||||||||
| ウイルスについての詳細 | 中空か: YES / エンベロープを持つか: YES / 単離: OTHER / タイプ: VIRION | |||||||||||||||
| 緩衝液 | pH: 8 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | |||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: UltrAuFoil | |||||||||||||||
急速凍結 | 装置: LEICA EM GP / 凍結剤: ETHANE / 湿度: 100 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 800 nm / C2レンズ絞り径: 100 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 800 nm / C2レンズ絞り径: 100 µm |
| 撮影 | 平均露光時間: 2.5 sec. / 電子線照射量: 66.5 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 1131 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 75582 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C3 (3回回転対称 : C3 (3回回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.22 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 54395 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 120 / プロトコル: RIGID BODY FIT / 空間: REAL / Target criteria: CC | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 6VXX PDB chain-ID: A / Pdb chain residue range: 27-1147 | ||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 120.7 Å2 | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






