+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6w4h | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | 1.80 Angstrom Resolution Crystal Structure of NSP16 - NSP10 Complex from SARS-CoV-2 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  Structural Genomics (構造ゲノミクス) / Center for Structural Genomics of Infectious Diseases / CSGID / nsp16 / nsp10 / Structural Genomics (構造ゲノミクス) / Center for Structural Genomics of Infectious Diseases / CSGID / nsp16 / nsp10 /  complex complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein guanylyltransferase activity / RNA endonuclease activity, producing 3'-phosphomonoesters /  mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs ...protein guanylyltransferase activity / RNA endonuclease activity, producing 3'-phosphomonoesters / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs ...protein guanylyltransferase activity / RNA endonuclease activity, producing 3'-phosphomonoesters /  mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex /  snRNP Assembly / TRAF3-dependent IRF activation pathway / Replication of the SARS-CoV-2 genome / snRNP Assembly / TRAF3-dependent IRF activation pathway / Replication of the SARS-CoV-2 genome /  加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ /  : / double membrane vesicle viral factory outer membrane / : / double membrane vesicle viral factory outer membrane /  3C様プロテアーゼ / host cell endosome / 3'-5'-RNA exonuclease activity / host cell endoplasmic reticulum-Golgi intermediate compartment / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated degradation of host mRNA / 3C様プロテアーゼ / host cell endosome / 3'-5'-RNA exonuclease activity / host cell endoplasmic reticulum-Golgi intermediate compartment / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated degradation of host mRNA /  mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / SARS-CoV-2 modulates host translation machinery / mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / SARS-CoV-2 modulates host translation machinery /  omega peptidase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / host cell Golgi apparatus / symbiont-mediated perturbation of host ubiquitin-like protein modification / omega peptidase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / host cell Golgi apparatus / symbiont-mediated perturbation of host ubiquitin-like protein modification /  mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity /  ヘリカーゼ / ヘリカーゼ /  ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /  single-stranded RNA binding / host cell endoplasmic reticulum membrane / host cell perinuclear region of cytoplasm / single-stranded RNA binding / host cell endoplasmic reticulum membrane / host cell perinuclear region of cytoplasm /  viral protein processing / viral protein processing /  lyase activity / lyase activity /  ヘリカーゼ / induction by virus of host autophagy / ヘリカーゼ / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / copper ion binding / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / copper ion binding / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / RNA-dependent RNA polymerase activity / DNA-templated transcription /  lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses / lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  ATP binding / ATP binding /  生体膜 生体膜類似検索 - 分子機能 | ||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Minasov, G. / Shuvalova, L. / Rosas-Lemus, M. / Kiryukhina, O. / Wiersum, G. / Godzik, A. / Jaroszewski, L. / Stogios, P.J. / Skarina, T. / Satchell, K.J.F. / Center for Structural Genomics of Infectious Diseases (CSGID) | ||||||
 引用 引用 |  ジャーナル: Sci.Signal. / 年: 2020 ジャーナル: Sci.Signal. / 年: 2020タイトル: High-resolution structures of the SARS-CoV-2 2'- O -methyltransferase reveal strategies for structure-based inhibitor design. 著者: Rosas-Lemus, M. / Minasov, G. / Shuvalova, L. / Inniss, N.L. / Kiryukhina, O. / Brunzelle, J. / Satchell, K.J.F. #1: ジャーナル: Biorxiv / 年: 2020 タイトル: The crystal structure of nsp10-nsp16 heterodimer from SARS-CoV-2 in complex with S-adenosylmethionine. 著者: Rosas-Lemus, M. / Minasov, G. / Shuvalova, L. / Inniss, N.L. / Kiryukhina, O. / Wiersum, G. / Kim, Y. / Jedrzejczak, R. / Maltseva, N.I. / Endres, M. / Jaroszewski, L. / Godzik, A. / ...著者: Rosas-Lemus, M. / Minasov, G. / Shuvalova, L. / Inniss, N.L. / Kiryukhina, O. / Wiersum, G. / Kim, Y. / Jedrzejczak, R. / Maltseva, N.I. / Endres, M. / Jaroszewski, L. / Godzik, A. / Joachimiak, A. / Satchell, K.J.F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6w4h.cif.gz 6w4h.cif.gz | 205.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6w4h.ent.gz pdb6w4h.ent.gz | 161.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6w4h.json.gz 6w4h.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/w4/6w4h https://data.pdbj.org/pub/pdb/validation_reports/w4/6w4h ftp://data.pdbj.org/pub/pdb/validation_reports/w4/6w4h ftp://data.pdbj.org/pub/pdb/validation_reports/w4/6w4h | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  6w75C  6wjtC  6wkqC  6wq3C  6wrzC  6wvnC  3r24S S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 実験データセット #1 | データ参照:  10.18430/m36w4h / データの種類: diffraction image data 10.18430/m36w4h / データの種類: diffraction image data |
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 33627.504 Da / 分子数: 1 / 断片: UNP residues 6799-7096 / 由来タイプ: 組換発現 由来: (組換発現)   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)遺伝子: rep, 1a-1b / プラスミド: pMCSG53 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): magic Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): magic参照: UniProt: P0DTD1,  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの |
|---|---|
| #2: タンパク質 | 分子量: 15075.190 Da / 分子数: 1 / 断片: UNP residues 4254-4392 / 由来タイプ: 組換発現 由来: (組換発現)   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)遺伝子: rep, 1a-1b / プラスミド: pMCSG53 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): magic / 参照: UniProt: P0DTD1 Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): magic / 参照: UniProt: P0DTD1 |
-糖 , 1種, 2分子 
| #6: 糖 |  フルクトース フルクトース |
|---|
-非ポリマー , 5種, 448分子 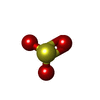








| #3: 化合物 | ChemComp-SO3 /  Sulfite Sulfite | ||||
|---|---|---|---|---|---|
| #4: 化合物 | ChemComp-SAM /  S-アデノシルメチオニン S-アデノシルメチオニン | ||||
| #5: 化合物 |  酢酸塩 酢酸塩#7: 化合物 | #8: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.33 Å3/Da / 溶媒含有率: 71.6 % |
|---|---|
結晶化 | 温度: 292 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 5.3 mg/mL 1:1 nsp16/nsp10 in 0.15 M sodium chloride, 0.01 M Tris, pH 7.5, 2 mM SAM, 1 mM TCEP, 5% glycerol against ComPAS screen A7 (0.2 M calcium acetate, 0.1 M HEPES, pH 7.5, 18% w/v PEG ...詳細: 5.3 mg/mL 1:1 nsp16/nsp10 in 0.15 M sodium chloride, 0.01 M Tris, pH 7.5, 2 mM SAM, 1 mM TCEP, 5% glycerol against ComPAS screen A7 (0.2 M calcium acetate, 0.1 M HEPES, pH 7.5, 18% w/v PEG 8000), cryoprotectant: 1:1 screen + 50% sucrose |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 21-ID-F / 波長: 0.97872 Å / ビームライン: 21-ID-F / 波長: 0.97872 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2020年3月7日 / 詳細: C(111) |
| 放射 | モノクロメーター: diamond(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97872 Å / 相対比: 1 : 0.97872 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→30 Å / Num. obs: 77886 / % possible obs: 100 % / Observed criterion σ(I): -3 / 冗長度: 7.3 % / Biso Wilson estimate: 25.4 Å2 / Rmerge(I) obs: 0.06 / Rpim(I) all: 0.024 / Rrim(I) all: 0.065 / Rsym value: 0.06 / Χ2: 1.007 / Net I/σ(I): 29.3 |
| 反射 シェル | 解像度: 1.8→1.83 Å / 冗長度: 6.8 % / Rmerge(I) obs: 0.765 / Mean I/σ(I) obs: 2.6 / Num. unique obs: 3873 / CC1/2: 0.762 / CC star: 0.93 / Rpim(I) all: 0.313 / Rrim(I) all: 0.828 / Rsym value: 0.765 / Χ2: 0.992 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB entry 3R24 解像度: 1.8→29.76 Å / Cor.coef. Fo:Fc: 0.972 / Cor.coef. Fo:Fc free: 0.966 / SU B: 2.962 / SU ML: 0.047 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.073 / ESU R Free: 0.07 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : WITH TLS ADDED
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 115.28 Å2 / Biso mean: 31.531 Å2 / Biso min: 15.7 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.8→29.76 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.8→1.847 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj















