+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6qxf | ||||||
|---|---|---|---|---|---|---|---|
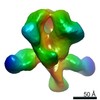
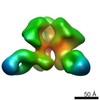
| タイトル | Cas1-Cas2-Csn2-DNA complex from the Type II-A CRISPR-Cas system | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN (DNA結合タンパク質) /  CRISPR (CRISPR) / Cas / CRISPR (CRISPR) / Cas /  DNA binding (デオキシリボ核酸) / DNA binding (デオキシリボ核酸) /  Nuclease (ヌクレアーゼ) / Nuclease (ヌクレアーゼ) /  Adaptation (適応 (生物学)) / Spacer acquisition / Adaptation (適応 (生物学)) / Spacer acquisition /  Protein complex (タンパク質複合体) / Protein complex (タンパク質複合体) /  Protein-DNA complex (デオキシリボ核酸) / Protein-DNA complex (デオキシリボ核酸) /  Calcium (カルシウム) / Metal-binding / Calcium (カルシウム) / Metal-binding /  Antiviral (抗ウイルス薬) Antiviral (抗ウイルス薬) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / RNA endonuclease activity / DNA endonuclease activity / defense response to virus /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  DNA binding / DNA binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)  Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||
 データ登録者 データ登録者 | Wilkinson, M. / Drabavicius, G. / Silanskas, A. / Gasiunas, G. / Siksnys, V. / Wigley, D.B. | ||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2019 ジャーナル: Mol Cell / 年: 2019タイトル: Structure of the DNA-Bound Spacer Capture Complex of a Type II CRISPR-Cas System. 著者: Martin Wilkinson / Gediminas Drabavicius / Arunas Silanskas / Giedrius Gasiunas / Virginijus Siksnys / Dale B Wigley /  要旨: CRISPR and associated Cas proteins function as an adaptive immune system in prokaryotes to combat bacteriophage infection. During the immunization step, new spacers are acquired by the CRISPR ...CRISPR and associated Cas proteins function as an adaptive immune system in prokaryotes to combat bacteriophage infection. During the immunization step, new spacers are acquired by the CRISPR machinery, but the molecular mechanism of spacer capture remains enigmatic. We show that the Cas9, Cas1, Cas2, and Csn2 proteins of a Streptococcus thermophilus type II-A CRISPR-Cas system form a complex and provide cryoelectron microscopy (cryo-EM) structures of three different assemblies. The predominant form, with the stoichiometry Cas1-Cas2-Csn2, referred to as monomer, contains ∼30 bp duplex DNA bound along a central channel. A minor species, termed a dimer, comprises two monomers that sandwich a further eight Cas1 and four Cas2 subunits and contains two DNA ∼30-bp duplexes within the channel. A filamentous form also comprises Cas1-Cas2-Csn2 units (typically 2-6) but with a different Cas1-Cas2 interface between them and a continuous DNA duplex running along a central channel. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6qxf.cif.gz 6qxf.cif.gz | 839.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6qxf.ent.gz pdb6qxf.ent.gz | 709.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6qxf.json.gz 6qxf.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qx/6qxf https://data.pdbj.org/pub/pdb/validation_reports/qx/6qxf ftp://data.pdbj.org/pub/pdb/validation_reports/qx/6qxf ftp://data.pdbj.org/pub/pdb/validation_reports/qx/6qxf | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-CRISPR-associated ... , 3種, 20分子 ABCDEFGHIJLMOPRSKNQT
| #1: タンパク質 | 分子量: 25533.473 Da / 分子数: 8 / 由来タイプ: 組換発現 由来: (組換発現)   Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)遺伝子: csn2 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: G3ECR4 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: G3ECR4#2: タンパク質 | 分子量: 35363.461 Da / 分子数: 8 / Mutation: C-terminal Strep tag / 由来タイプ: 組換発現 由来: (組換発現)   Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)遺伝子: cas1 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: G3ECR2,  加水分解酵素; エステル加水分解酵素 加水分解酵素; エステル加水分解酵素#3: タンパク質 | 分子量: 13431.560 Da / 分子数: 4 / 由来タイプ: 組換発現 由来: (組換発現)   Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)遺伝子: cas2 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: G3ECR3,  加水分解酵素; エステル加水分解酵素 加水分解酵素; エステル加水分解酵素 |
|---|
-DNA鎖 , 2種, 2分子 XY
| #4: DNA鎖 | 分子量: 7559.863 Da / 分子数: 1 / 由来タイプ: 天然 詳細: Ordered portion representing the mixed DNA fragments bound to the complex after sonicating cells and treating with DNaseI. 由来: (天然)   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
|---|---|
| #5: DNA鎖 | 分子量: 7785.214 Da / 分子数: 1 / 由来タイプ: 天然 詳細: Ordered portion representing the mixed DNA fragments bound to the complex after sonicating cells and treating with DNaseI. 由来: (天然)   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
-非ポリマー , 1種, 8分子 
| #6: 化合物 | ChemComp-CA / |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.53 MDa / 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) | ||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 / 詳細: Buffer solution was filtered | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 0.05 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: Sample was DNaseI treated followed by gel filtration. Peak fractions were concentrated prior to making grids. | ||||||||||||||||||||||||
| 試料支持 | 詳細: Graphene oxide sheets were applied to the grid prior to sample application グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: C-flat-2/1 | ||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 90 % / 凍結前の試料温度: 277 K / 詳細: 15 s wait time, 1 s blot time. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 75000 X / 倍率(補正後): 129032 X / 最大 デフォーカス(公称値): 2800 nm / 最小 デフォーカス(公称値): 1300 nm / Cs Bright-field microscopy / 倍率(公称値): 75000 X / 倍率(補正後): 129032 X / 最大 デフォーカス(公称値): 2800 nm / 最小 デフォーカス(公称値): 1300 nm / Cs : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 1 sec. / 電子線照射量: 92.8 e/Å2 / 検出モード: INTEGRATING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 4908 / 詳細: Images were collected in movie mode with 39 frames |
| 画像スキャン | サンプリングサイズ: 14 µm |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 853391 詳細: Reproductions of an initial ab initio generated 3D reconstruction were used as templates for auto picking | ||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 306470 / クラス平均像の数: 5 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL Target criteria: Correlation coefficient and visual inspection 詳細: Rounds of manual fitting in Coot, jelly-body refinement in REFMAC and real-space refinement in PHENIX | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj









































