登録情報 データベース : PDB / ID : 6nnaタイトル Human Fatty Acid Synthase Psi/KR Tri-Domain with NADPH and Compound 22 Fatty acid synthase,Fatty acid synthase キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 2.26 Å データ登録者 Toms, A.V. / Martin, M.W. ジャーナル : Bioorg. Med. Chem. Lett. / 年 : 2019タイトル : Discovery and optimization of novel piperazines as potent inhibitors of fatty acid synthase (FASN).著者: Martin, M.W. / Lancia Jr., D.R. / Li, H. / Schiller, S.E.R. / Toms, A.V. / Wang, Z. / Bair, K.W. / Castro, J. / Fessler, S. / Gotur, D. / Hubbs, S.E. / Kauffman, G.S. / Kershaw, M. / Luke, G. ... 著者 : Martin, M.W. / Lancia Jr., D.R. / Li, H. / Schiller, S.E.R. / Toms, A.V. / Wang, Z. / Bair, K.W. / Castro, J. / Fessler, S. / Gotur, D. / Hubbs, S.E. / Kauffman, G.S. / Kershaw, M. / Luke, G.P. / McKinnon, C. / Yao, L. / Lu, W. / Millan, D.S. 履歴 登録 2019年1月14日 登録サイト / 処理サイト 改定 1.0 2019年2月20日 Provider / タイプ 改定 1.1 2019年3月13日 Group / Database referencesカテゴリ / citation_author / pdbx_database_procItem _citation.journal_abbrev / _citation.pdbx_database_id_PubMed ... _citation.journal_abbrev / _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.identifier_ORCID / _citation_author.name 改定 1.2 2019年3月20日 Group / Database references / カテゴリ Item / _citation.page_first / _citation.page_last改定 1.3 2023年10月11日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) /
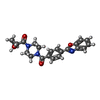
TRANSFERASE (転移酵素) /  FATTY ACID SYNTHASE (脂肪酸合成酵素) / HUMAN FAS / KETO-REDUCTASE
FATTY ACID SYNTHASE (脂肪酸合成酵素) / HUMAN FAS / KETO-REDUCTASE 機能・相同性情報
機能・相同性情報 グリコーゲン / establishment of endothelial intestinal barrier / [acyl-carrier-protein] S-acetyltransferase / [acyl-carrier-protein] S-acetyltransferase activity / Fatty acyl-CoA biosynthesis / oleoyl-[acyl-carrier-protein] hydrolase / (3R)-3-hydroxypalmitoyl-[acyl-carrier-protein] dehydratase activity / modulation by host of viral process / ChREBP activates metabolic gene expression / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity /
グリコーゲン / establishment of endothelial intestinal barrier / [acyl-carrier-protein] S-acetyltransferase / [acyl-carrier-protein] S-acetyltransferase activity / Fatty acyl-CoA biosynthesis / oleoyl-[acyl-carrier-protein] hydrolase / (3R)-3-hydroxypalmitoyl-[acyl-carrier-protein] dehydratase activity / modulation by host of viral process / ChREBP activates metabolic gene expression / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity /  fatty acid synthase activity / 3-hydroxyacyl-[acyl-carrier-protein] dehydratase / (3R)-3-hydroxymyristoyl-[acyl-carrier-protein] dehydratase activity / beta-ketoacyl-[acyl-carrier-protein] synthase I / NR1H2 & NR1H3 regulate gene expression linked to lipogenesis /
fatty acid synthase activity / 3-hydroxyacyl-[acyl-carrier-protein] dehydratase / (3R)-3-hydroxymyristoyl-[acyl-carrier-protein] dehydratase activity / beta-ketoacyl-[acyl-carrier-protein] synthase I / NR1H2 & NR1H3 regulate gene expression linked to lipogenesis /  mammary gland development / 3-oxoacyl-[acyl-carrier-protein] reductase / 3-oxoacyl-[acyl-carrier-protein] reductase (NADPH) activity /
mammary gland development / 3-oxoacyl-[acyl-carrier-protein] reductase / 3-oxoacyl-[acyl-carrier-protein] reductase (NADPH) activity /  phosphopantetheine binding / monocyte differentiation / 3-oxoacyl-[acyl-carrier-protein] synthase activity / cellular response to interleukin-4 / fatty acid metabolic process / Activation of gene expression by SREBF (SREBP) / osteoblast differentiation / fatty acid biosynthetic process /
phosphopantetheine binding / monocyte differentiation / 3-oxoacyl-[acyl-carrier-protein] synthase activity / cellular response to interleukin-4 / fatty acid metabolic process / Activation of gene expression by SREBF (SREBP) / osteoblast differentiation / fatty acid biosynthetic process /  メラノソーム /
メラノソーム /  cadherin binding /
cadherin binding /  炎症 /
炎症 /  ゴルジ体 /
ゴルジ体 /  RNA binding / extracellular exosome /
RNA binding / extracellular exosome /  生体膜 /
生体膜 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.26 Å
分子置換 / 解像度: 2.26 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg. Med. Chem. Lett. / 年: 2019
ジャーナル: Bioorg. Med. Chem. Lett. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6nna.cif.gz
6nna.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6nna.ent.gz
pdb6nna.ent.gz PDB形式
PDB形式 6nna.json.gz
6nna.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nn/6nna
https://data.pdbj.org/pub/pdb/validation_reports/nn/6nna ftp://data.pdbj.org/pub/pdb/validation_reports/nn/6nna
ftp://data.pdbj.org/pub/pdb/validation_reports/nn/6nna
 リンク
リンク 集合体
集合体


 要素
要素
 Homo sapiens (ヒト) / 遺伝子: FASN, FAS
Homo sapiens (ヒト) / 遺伝子: FASN, FAS
 Spodoptera frugiperda (ツマジロクサヨトウ)
Spodoptera frugiperda (ツマジロクサヨトウ) ニコチンアミドアデニンジヌクレオチドリン酸
ニコチンアミドアデニンジヌクレオチドリン酸 エチレングリコール
エチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL18U1 / 波長: 0.97853 Å
/ ビームライン: BL18U1 / 波長: 0.97853 Å : 0.97853 Å / 相対比: 1
: 0.97853 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj