+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6kig | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of cyanobacterial photosystem I-IsiA supercomplex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  PHOTOSYNTHESIS (光合成) / PHOTOSYNTHESIS (光合成) /  Photosystem / Antenna / Chlorophyll-binding protein / Photosystem / Antenna / Chlorophyll-binding protein /  Membrane protein (膜タンパク質) / iron stress-induced protein A Membrane protein (膜タンパク質) / iron stress-induced protein A | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / photosynthetic electron transport chain / 光化学系I / photosynthetic electron transport in photosystem I / photosynthetic electron transport chain /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding / 光合成 / 4 iron, 4 sulfur cluster binding /  electron transfer activity ... electron transfer activity ... photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / photosynthetic electron transport chain / 光化学系I / photosynthetic electron transport in photosystem I / photosynthetic electron transport chain /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding / 光合成 / 4 iron, 4 sulfur cluster binding /  electron transfer activity / magnesium ion binding / electron transfer activity / magnesium ion binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Synechococcus elongatus (バクテリア) Synechococcus elongatus (バクテリア) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Cao, P. / Cao, D.F. / Si, L. / Su, X.D. / Chang, W.R. / Liu, Z.F. / Zhang, X.Z. / Li, M. | ||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2020 ジャーナル: Nat Plants / 年: 2020タイトル: Structural basis for energy and electron transfer of the photosystem I-IsiA-flavodoxin supercomplex. 著者: Peng Cao / Duanfang Cao / Long Si / Xiaodong Su / Lijin Tian / Wenrui Chang / Zhenfeng Liu / Xinzheng Zhang / Mei Li /  要旨: Under iron-deficiency stress, which occurs frequently in natural aquatic environments, cyanobacteria reduce the amount of iron-enriched proteins, including photosystem I (PSI) and ferredoxin (Fd), ...Under iron-deficiency stress, which occurs frequently in natural aquatic environments, cyanobacteria reduce the amount of iron-enriched proteins, including photosystem I (PSI) and ferredoxin (Fd), and upregulate the expression of iron-stress-induced proteins A and B (IsiA and flavodoxin (Fld)). Multiple IsiAs function as the peripheral antennae that encircle the PSI core, whereas Fld replaces Fd as the electron receptor of PSI. Here, we report the structures of the PSI-IsiA-Fld and PSI-IsiA supercomplexes from Synechococcus sp. PCC 7942, revealing features that are different from the previously reported PSI structures, and a sophisticated pigment network that involves previously unobserved pigment molecules. Spectroscopic results demonstrated that IsiAs are efficient light harvesters for PSI. Three Flds bind symmetrically to the trimeric PSI core-we reveal the detailed interaction and the electron transport path between PSI and Fld. Our results provide a structural basis for understanding the mechanisms of light harvesting, energy transfer and electron transport of cyanobacterial PSI under stressed conditions. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6kig.cif.gz 6kig.cif.gz | 3.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6kig.ent.gz pdb6kig.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  6kig.json.gz 6kig.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ki/6kig https://data.pdbj.org/pub/pdb/validation_reports/ki/6kig ftp://data.pdbj.org/pub/pdb/validation_reports/ki/6kig ftp://data.pdbj.org/pub/pdb/validation_reports/ki/6kig | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Photosystem I ... , 10種, 30分子 AGeBHfCNgDOhEQiFRjISkJTlKUmLVn
| #1: タンパク質 |  光化学系I / PsaA 光化学系I / PsaA分子量: 83994.336 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31LJ0,  光化学系I 光化学系I#2: タンパク質 |  光化学系I / PsaB 光化学系I / PsaB分子量: 81554.742 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31LJ1,  光化学系I 光化学系I#3: タンパク質 |  光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC 光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC分子量: 8807.235 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31QV2,  光化学系I 光化学系I#4: タンパク質 |  光化学系I / PsaD 光化学系I / PsaD分子量: 15536.583 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31PI7 #5: タンパク質 |  光化学系I 光化学系I分子量: 8147.134 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31NL7 #6: タンパク質 |  光化学系I 光化学系I分子量: 17125.742 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31NT9 #7: タンパク質・ペプチド |  光化学系I / PsaI / Photosystem I subunit VIII 光化学系I / PsaI / Photosystem I subunit VIII分子量: 4002.733 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: P95823 #8: タンパク質・ペプチド |  光化学系I / PsaJ 光化学系I / PsaJ分子量: 4713.585 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31NU0 #9: タンパク質 |  光化学系I / Photosystem I subunit X 光化学系I / Photosystem I subunit X分子量: 8397.120 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: Q31PR9 #10: タンパク質 |  光化学系I / PSI subunit V / PSI-L 光化学系I / PSI subunit V / PSI-L分子量: 17302.688 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: P95822 |
|---|
-タンパク質・ペプチド / タンパク質 / 糖 , 3種, 39分子 MWo123456YZabcdqrstuv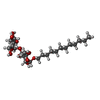

| #11: タンパク質・ペプチド | 分子量: 3239.867 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照:  光化学系I 光化学系I#12: タンパク質 | 分子量: 37004.453 Da / 分子数: 18 / 由来タイプ: 天然 由来: (天然)   Synechococcus elongatus (strain PCC 7942) (バクテリア) Synechococcus elongatus (strain PCC 7942) (バクテリア)株: PCC 7942 / 参照: UniProt: P15347 #18: 糖 | ChemComp-LMU / |
|---|
-非ポリマー , 8種, 882分子 














| #13: 化合物 | ChemComp-CLA /  クロロフィルa クロロフィルa#14: 化合物 | ChemComp-PQN /  フィロキノン フィロキノン#15: 化合物 | ChemComp-SF4 /  鉄・硫黄クラスター 鉄・硫黄クラスター#16: 化合物 | ChemComp-BCR /  Β-カロテン Β-カロテン#17: 化合物 | ChemComp-LHG /  Phosphatidylglycerol Phosphatidylglycerol#19: 化合物 | ChemComp-LMG / #20: 化合物 | ChemComp-SQD / #21: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Photosystem I-IsiA supercomplex 光化学系I / タイプ: COMPLEX / Entity ID: #1-#12 / 由来: NATURAL 光化学系I / タイプ: COMPLEX / Entity ID: #1-#12 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Synechococcus elongatus PCC 7942 (バクテリア) Synechococcus elongatus PCC 7942 (バクテリア)株: PCC 7942 |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | 電子線照射量: 60 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||
| 対称性 | 点対称性 : C3 (3回回転対称 : C3 (3回回転対称 ) ) | |||||||||
3次元再構成 | 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 63332 / 対称性のタイプ: POINT | |||||||||
| 原子モデル構築 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

















