+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6hqb | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Monomeric cyanobacterial photosystem I | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  PHOTOSYNTHESIS (光合成) / PHOTOSYNTHESIS (光合成) /  Crystal structure (結晶構造) / Crystal structure (結晶構造) /  Cyanobacteria (藍藻) / Membrane complexes / Cyanobacteria (藍藻) / Membrane complexes /  Photosystem I (光化学系I) / Photosystem I (光化学系I) /  Synechocystis Synechocystis | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 plasma membrane-derived photosystem I / plasma membrane-derived photosystem I /  photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding / 光合成 / 4 iron, 4 sulfur cluster binding /  electron transfer activity ... electron transfer activity ... plasma membrane-derived photosystem I / plasma membrane-derived photosystem I /  photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding / 光合成 / 4 iron, 4 sulfur cluster binding /  electron transfer activity / magnesium ion binding / electron transfer activity / magnesium ion binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 4 Å 分子置換 / 解像度: 4 Å | ||||||
 データ登録者 データ登録者 | Netzer-El, S.Y. / Nelson, N. / Caspy, I. | ||||||
| 資金援助 |  ベルギー, 1件 ベルギー, 1件
| ||||||
 引用 引用 |  ジャーナル: Front Plant Sci / 年: 2018 ジャーナル: Front Plant Sci / 年: 2018タイトル: Crystal Structure of Photosystem I Monomer From Synechocystis PCC 6803. 著者: Netzer-El, S.Y. / Caspy, I. / Nelson, N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6hqb.cif.gz 6hqb.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6hqb.ent.gz pdb6hqb.ent.gz | 1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6hqb.json.gz 6hqb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hq/6hqb https://data.pdbj.org/pub/pdb/validation_reports/hq/6hqb ftp://data.pdbj.org/pub/pdb/validation_reports/hq/6hqb ftp://data.pdbj.org/pub/pdb/validation_reports/hq/6hqb | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Photosystem I P700 chlorophyll a apoprotein ... , 2種, 2分子 AB
| #1: タンパク質 |  光化学系I / PsaA 光化学系I / PsaA分子量: 83036.398 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P29254,  光化学系I 光化学系I |
|---|---|
| #2: タンパク質 |  光化学系I / PsaB 光化学系I / PsaB分子量: 81369.531 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P29255,  光化学系I 光化学系I |
-Photosystem I reaction center subunit ... , 8種, 8分子 DEFIJKLM
| #4: タンパク質 |  光化学系I / Photosystem I 16 kDa polypeptide / PSI-D 光化学系I / Photosystem I 16 kDa polypeptide / PSI-D分子量: 15663.749 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P19569 |
|---|---|
| #5: タンパク質 |  光化学系I / Photosystem I 8.1 kDa protein / p30 protein 光化学系I / Photosystem I 8.1 kDa protein / p30 protein分子量: 7680.478 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P12975 |
| #6: タンパク質 |  光化学系I / PSI-F 光化学系I / PSI-F分子量: 15791.086 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P29256 |
| #7: タンパク質・ペプチド |  光化学系I 光化学系I分子量: 4414.148 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: Q55330 |
| #8: タンパク質・ペプチド |  光化学系I 光化学系I分子量: 4535.415 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: Q55329 |
| #9: タンパク質 |  光化学系I / Photosystem I subunit X 2 光化学系I / Photosystem I subunit X 2分子量: 7339.811 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P74564 |
| #10: タンパク質 |  光化学系I / PSI subunit V / PSI-L 光化学系I / PSI subunit V / PSI-L分子量: 14523.503 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P37277 |
| #11: タンパク質・ペプチド |  光化学系I / PSI-M 光化学系I / PSI-M分子量: 3382.063 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P72986 |
-タンパク質 / 糖 , 2種, 2分子 C

| #24: 糖 | ChemComp-LMT / |
|---|---|
| #3: タンパク質 |  光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC 光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC分子量: 8706.064 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: PCC 6803 / Kazusa / 参照: UniProt: P32422,  光化学系I 光化学系I |
-非ポリマー , 13種, 129分子 










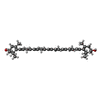













| #12: 化合物 | ChemComp-CLA /  クロロフィルa クロロフィルa#13: 化合物 |  フィロキノン フィロキノン#14: 化合物 |  鉄・硫黄クラスター 鉄・硫黄クラスター#15: 化合物 | ChemComp-BCR /  Β-カロテン Β-カロテン#16: 化合物 | ChemComp-LHG /  Phosphatidylglycerol Phosphatidylglycerol#17: 化合物 | ChemComp-45D / |  カンタキサンチン カンタキサンチン#18: 化合物 | ChemComp-CL / |  塩化物 塩化物#19: 化合物 |  Echinenone Echinenone#20: 化合物 | #21: 化合物 | #22: 化合物 | #23: 化合物 | #25: 化合物 | ChemComp-EQ3 / ( |  3'-Hydroxyechinenone 3'-Hydroxyechinenone |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.35 Å3/Da / 溶媒含有率: 69.9 % |
|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 詳細: KCl, MES-NaOH, PEG 3350, sulfoquinovosyldiacylglycerol or Jeffamine M-600 |
-データ収集
| 回折 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| |||||||||||||||
| 検出器 |
| |||||||||||||||
| 放射 |
| |||||||||||||||
| 放射波長 |
| |||||||||||||||
| 反射 | 解像度: 4→50 Å / Num. obs: 34852 / % possible obs: 99.7 % / 冗長度: 24 % / Rpim(I) all: 0.092 / Net I/σ(I): 4.9 | |||||||||||||||
| 反射 シェル | 解像度: 4→50 Å / Rpim(I) all: 0.437 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 4→49.289 Å / SU ML: 0.5 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 34.87 / 立体化学のターゲット値: ML 分子置換 / 解像度: 4→49.289 Å / SU ML: 0.5 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 34.87 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 4→49.289 Å
| ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -40.0247 Å / Origin y: -23.0823 Å / Origin z: -34.8269 Å
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj

























