| #3: 化合物 | ChemComp-J7T / molybdate cluster
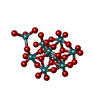  分子量 分子量: 1359.441 Da / 分子数: 1 / 由来タイプ: 合成 / 式: Mo 9O 31 |
|---|
| #4: 化合物 | ChemComp-J7N / 2,2,4-tris(oxidanyl)-1,3-dioxa-2$l^{4},4$l^{3}-dimolybdacyclobutane
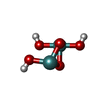  分子量 分子量: 274.901 Da / 分子数: 1 / 由来タイプ: 合成 / 式: H 3Mo 2O 5 |
|---|
| #5: 化合物 | ChemComp-J8E / oxidanyl-[[2,2,4,4,4-pentakis($l^{1}-oxidanyl)-1-(oxidanylmolybdenio)-1$l^{3},3-dioxa-2$l^{5},4$l^{5}-dimolybdacyclobut-2-yl]oxy]molybdenum
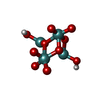  分子量 分子量: 545.770 Da / 分子数: 1 / 由来タイプ: 合成 / 式: H 2Mo 4O 10 |
|---|
| #6: 化合物 | ChemComp-FUQ / Mo5 Cluster
  分子量 分子量: 899.844 Da / 分子数: 1 / 由来タイプ: 合成 / 式: H 20Mo 5O 25 |
|---|
| #7: 化合物 | ChemComp-J7Q / tetrakis($l^{1}-oxidanyl)-[[2,2,2,4,4,4,4,6,6,6,8-undecakis($l^{1}-oxidanyl)-8-(oxidanylmolybdeniooxy)-6-[tris($l^{1}-oxidanyl)molybdeniooxy]-1,3,5,7-tetraoxa-2$l^{6},4$l^{6},6$l^{6},8$l^{4}-tetramolybdacyclooct-2-yl]oxy]molybdenum
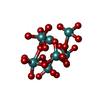  分子量 分子量: 1088.572 Da / 分子数: 1 / 由来タイプ: 合成 / 式: HMo 7O 26 |
|---|
| #8: 化合物 | ChemComp-ATP / ADENOSINE-5'-TRIPHOSPHATE / ATP /  アデノシン三リン酸 アデノシン三リン酸
  分子量 分子量: 507.181 Da / 分子数: 1 / 由来タイプ: 合成 / 式: C 10H 16N 5O 13P 3 / コメント: ATP, エネルギー貯蔵分子 *YM |
|---|
| #9: 化合物 | ChemComp-MG / MAGNESIUM ION / マグネシウムジカチオン
  分子量 分子量: 24.305 Da / 分子数: 1 / 由来タイプ: 合成 / 式: Mg |
|---|
| #10: 化合物 | ChemComp-J8B / Molybdate cluster
  分子量 分子量: 1183.504 Da / 分子数: 1 / 由来タイプ: 合成 / 式: Mo 8O 26 |
|---|
| #11: 化合物 | ChemComp-J85 / [[[bis(oxidanylmolybdenio)-$l^{3}-oxidanyl]-$l^{1}-oxidanyl-oxidanylidene-molybdenio]-(oxidanylmolybdenio)-$l^{3}-oxidanyl]-tetrakis($l^{1}-oxidanyl)molybdenum
  分子量 分子量: 658.717 Da / 分子数: 1 / 由来タイプ: 合成 / 式: H 3Mo 5O 11 |
|---|
| #12: 化合物 | ChemComp-MOO / MOLYBDATE ION / MOLYBDATE /  モリブデン酸塩 モリブデン酸塩
  分子量 分子量: 159.938 Da / 分子数: 1 / 由来タイプ: 合成 / 式: MoO 4 |
|---|
| #13: 水 | ChemComp-HOH / water /  水 水
 分子量 分子量: 18.015 Da / 分子数: 199 / 由来タイプ: 天然 / 式: H 2O |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Azotobacter vinelandii (窒素固定)
Azotobacter vinelandii (窒素固定) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J. Inorg. Biochem. / 年: 2018
ジャーナル: J. Inorg. Biochem. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6h8b.cif.gz
6h8b.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6h8b.ent.gz
pdb6h8b.ent.gz PDB形式
PDB形式 6h8b.json.gz
6h8b.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/h8/6h8b
https://data.pdbj.org/pub/pdb/validation_reports/h8/6h8b ftp://data.pdbj.org/pub/pdb/validation_reports/h8/6h8b
ftp://data.pdbj.org/pub/pdb/validation_reports/h8/6h8b リンク
リンク 集合体
集合体

 要素
要素
 Azotobacter vinelandii (窒素固定) / 参照: UniProt: P84253
Azotobacter vinelandii (窒素固定) / 参照: UniProt: P84253
 Azotobacter vinelandii (窒素固定) / 参照: UniProt: P84308
Azotobacter vinelandii (窒素固定) / 参照: UniProt: P84308
















 アデノシン三リン酸
アデノシン三リン酸 モリブデン酸塩
モリブデン酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X10SA / 波長: 1 Å
/ ビームライン: X10SA / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj









