+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6gyo | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human HCN4 hyperpolarization-activated cyclic nucleotide-gated ion channel in complex with cAMP | |||||||||
 要素 要素 | Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 4 | |||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  ION CHANNEL (イオンチャネル) / PACEMAKER CURRENT ION CHANNEL (イオンチャネル) / PACEMAKER CURRENT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development /  HCN channels / HCN channels /  HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential ...voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development / HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential ...voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development /  HCN channels / HCN channels /  HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential / sodium ion import across plasma membrane / HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential / sodium ion import across plasma membrane /  voltage-gated sodium channel activity / voltage-gated sodium channel activity /  循環器 / 循環器 /  regulation of membrane depolarization / potassium ion import across plasma membrane / regulation of heart rate by cardiac conduction / regulation of membrane depolarization / potassium ion import across plasma membrane / regulation of heart rate by cardiac conduction /  voltage-gated potassium channel activity / monoatomic cation transport / sodium ion transmembrane transport / regulation of cardiac muscle contraction / voltage-gated potassium channel activity / monoatomic cation transport / sodium ion transmembrane transport / regulation of cardiac muscle contraction /  cAMP binding / cellular response to cAMP / potassium ion transmembrane transport / cAMP binding / cellular response to cAMP / potassium ion transmembrane transport /  regulation of membrane potential / regulation of membrane potential /  regulation of heart rate / regulation of heart rate /  muscle contraction / muscle contraction /  神経繊維 / 神経繊維 /  樹状突起 / perinuclear region of cytoplasm / identical protein binding / 樹状突起 / perinuclear region of cytoplasm / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Shintre, C.A. / Pike, A.C.W. / Tessitore, A. / Young, M. / Bushell, S.R. / Strain-Damerell, C. / Mukhopadhyay, S. / Burgess-Brown, N.A. / Huiskonen, J.T. / Arrowsmith, C.H. ...Shintre, C.A. / Pike, A.C.W. / Tessitore, A. / Young, M. / Bushell, S.R. / Strain-Damerell, C. / Mukhopadhyay, S. / Burgess-Brown, N.A. / Huiskonen, J.T. / Arrowsmith, C.H. / Edwards, A.M. / Bountra, C. / Carpenter, E.P. / Structural Genomics Consortium (SGC) | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of human HCN4 hyperpolarization-activated cyclic nucleotide-gated ion channel 著者: Shintre, C.A. / Pike, A.C.W. / Tessitore, A. / Young, M. / Bushell, S.R. / Strain-Damerell, C. / Mukhopadhyay, S. / Burgess-Brown, N.A. / Huiskonen, J.T. / Arrowsmith, C.H. / Edwards, A.M. / ...著者: Shintre, C.A. / Pike, A.C.W. / Tessitore, A. / Young, M. / Bushell, S.R. / Strain-Damerell, C. / Mukhopadhyay, S. / Burgess-Brown, N.A. / Huiskonen, J.T. / Arrowsmith, C.H. / Edwards, A.M. / Bountra, C. / Carpenter, E.P. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6gyo.cif.gz 6gyo.cif.gz | 386.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6gyo.ent.gz pdb6gyo.ent.gz | 328 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6gyo.json.gz 6gyo.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gy/6gyo https://data.pdbj.org/pub/pdb/validation_reports/gy/6gyo ftp://data.pdbj.org/pub/pdb/validation_reports/gy/6gyo ftp://data.pdbj.org/pub/pdb/validation_reports/gy/6gyo | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 60836.754 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: HCN4 / プラスミド: pFB-LIC-Bse Homo sapiens (ヒト) / 遺伝子: HCN4 / プラスミド: pFB-LIC-Bse発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q9Y3Q4 |
|---|
-非ポリマー , 5種, 48分子 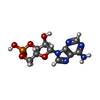








| #2: 化合物 | ChemComp-CMP /  環状アデノシン一リン酸 環状アデノシン一リン酸#3: 化合物 | ChemComp-PC1 /  ホスファチジルコリン ホスファチジルコリン#4: 化合物 | ChemComp-PIE / #5: 化合物 | ChemComp-3PE /  ホスファチジルエタノールアミン ホスファチジルエタノールアミン#6: 化合物 | ChemComp-UNX / |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Potassium/Sodium hyperpolarization-activated cyclic nucleotide-gated ion channel 4 タイプ: ORGANELLE OR CELLULAR COMPONENT / Entity ID: #1 / 由来: RECOMBINANT | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) | ||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||
| 試料 | 濃度: 5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 278 K / 詳細: blotted for 5.5s before plunge |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS / 詳細: EBIC TITAN KRIOS M07 |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 37313 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1300 nm Bright-field microscopy / 倍率(公称値): 37313 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1300 nm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 16 sec. / 電子線照射量: 48 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2338 |
| 画像スキャン | 動画フレーム数/画像: 48 / 利用したフレーム数/画像: 1-48 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.13_2998: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
| 画像処理 | 詳細: Images were motioncorrected and dose-weighted with MOTIONCOR2 | ||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 234908 詳細: Autopicking with templates derived from manually picked particles | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C4 (4回回転対称 : C4 (4回回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 55829 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: REAL 詳細: Initial model was from ligand-free HCN4. Model refined against the cryosparc b-factor sharpened map using default restraints |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj









