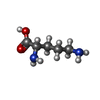
登録構造単位
A: L-amino acid oxidase/monooxygenase
B: L-amino acid oxidase/monooxygenase
C: L-amino acid oxidase/monooxygenase
D: L-amino acid oxidase/monooxygenase
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 262,267 16 ポリマ- 258,067 4 非ポリマー 4,199 12 水 14,268 792
1
A: L-amino acid oxidase/monooxygenase
C: L-amino acid oxidase/monooxygenase
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 131,133 8 ポリマ- 129,034 2 非ポリマー 2,100 6 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
Buried area 7570 Å2 ΔGint -27.5 kcal/mol Surface area 37100 Å2 手法
2
B: L-amino acid oxidase/monooxygenase
D: L-amino acid oxidase/monooxygenase
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 131,133 8 ポリマ- 129,034 2 非ポリマー 2,100 6 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
Buried area 7590 Å2 ΔGint -28 kcal/mol Surface area 37000 Å2 手法
単位格子 Length a, b, c (Å) 94.600, 132.900, 101.900 Angle α, β, γ (deg.) 90.000, 111.500, 90.000 Int Tables number 4 Space group name H-M P121 1
非結晶学的対称性 (NCS) NCSドメイン NCSドメイン領域 Dom-ID Component-ID Ens-ID Refine code Auth asym-ID Auth seq-ID 1 1 1 2 A1 - 600 2 1 1 2 B1 - 600 3 1 1 2 C1 - 600 4 1 1 2 D1 - 600
NCS oper ID Code Matrix ベクター 1 given(1), (1), (1)2 given(0.47269, 0.00365, -0.881222), (0.00359, -0.999991, -0.002216), (-0.881222, -0.002116, -0.472698)28.41823, -8.42104, 47.5023993 given(-0.688317, 0.505702, 0.520082), (0.497168, -0.193219, 0.845867), (0.528247, 0.840792, -0.118423)16.64119, -32.232811, 20.8872394 given(-0.782447, -0.508546, 0.359385), (-0.510341, 0.192973, -0.838041), (0.356831, -0.839132, -0.410523)17.48921, 23.76466, 23.15801
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード OXIDOREDUCTASE (酸化還元酵素) / L-amino acid oxidase/monooxygenase / flavin-containing monoamine oxidase family / flavin monooxygenases /
OXIDOREDUCTASE (酸化還元酵素) / L-amino acid oxidase/monooxygenase / flavin-containing monoamine oxidase family / flavin monooxygenases /  L-ornithine (オルニチン)
L-ornithine (オルニチン) 機能・相同性情報
機能・相同性情報
 Pseudomonas sp. AIU 813 (シュードモナス属)
Pseudomonas sp. AIU 813 (シュードモナス属) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: FEBS Open Bio / 年: 2018
ジャーナル: FEBS Open Bio / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5yb7.cif.gz
5yb7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5yb7.ent.gz
pdb5yb7.ent.gz PDB形式
PDB形式 5yb7.json.gz
5yb7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/yb/5yb7
https://data.pdbj.org/pub/pdb/validation_reports/yb/5yb7 ftp://data.pdbj.org/pub/pdb/validation_reports/yb/5yb7
ftp://data.pdbj.org/pub/pdb/validation_reports/yb/5yb7 リンク
リンク 集合体
集合体


 要素
要素
 Pseudomonas sp. AIU 813 (シュードモナス属)
Pseudomonas sp. AIU 813 (シュードモナス属)
 Escherichia coli (大腸菌) / 参照: UniProt: W6JQJ6
Escherichia coli (大腸菌) / 参照: UniProt: W6JQJ6 フラビンアデニンジヌクレオチド
フラビンアデニンジヌクレオチド オルニチン
オルニチン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Photon Factory
Photon Factory  / ビームライン: BL-5A / 波長: 1 Å
/ ビームライン: BL-5A / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj