+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5lnk | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Entire ovine respiratory complex I | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / NADH:ubiquinone / OXIDOREDUCTASE (酸化還元酵素) / NADH:ubiquinone /  complex I (NADH:ユビキノン還元酵素 (水素イオン輸送型)) / complex I (NADH:ユビキノン還元酵素 (水素イオン輸送型)) /  mammalian (哺乳類) / mammalian (哺乳類) /  mitochondrial (ミトコンドリア) mitochondrial (ミトコンドリア) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Complex I biogenesis / Respiratory electron transport / mitochondrial respirasome /  NADH dehydrogenase activity / respiratory chain complex I / oxidoreductase activity, acting on NAD(P)H / electron transport coupled proton transport / mitochondrial ATP synthesis coupled electron transport / NADH dehydrogenase activity / respiratory chain complex I / oxidoreductase activity, acting on NAD(P)H / electron transport coupled proton transport / mitochondrial ATP synthesis coupled electron transport /  NADH:ユビキノン還元酵素 (水素イオン輸送型) / mitochondrial respiratory chain complex I ...Complex I biogenesis / Respiratory electron transport / mitochondrial respirasome / NADH:ユビキノン還元酵素 (水素イオン輸送型) / mitochondrial respiratory chain complex I ...Complex I biogenesis / Respiratory electron transport / mitochondrial respirasome /  NADH dehydrogenase activity / respiratory chain complex I / oxidoreductase activity, acting on NAD(P)H / electron transport coupled proton transport / mitochondrial ATP synthesis coupled electron transport / NADH dehydrogenase activity / respiratory chain complex I / oxidoreductase activity, acting on NAD(P)H / electron transport coupled proton transport / mitochondrial ATP synthesis coupled electron transport /  NADH:ユビキノン還元酵素 (水素イオン輸送型) / mitochondrial respiratory chain complex I / apoptotic mitochondrial changes / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone / NADH:ユビキノン還元酵素 (水素イオン輸送型) / mitochondrial respiratory chain complex I / apoptotic mitochondrial changes / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone /  電子伝達系 / 電子伝達系 /  NADH dehydrogenase (ubiquinone) activity / NADH dehydrogenase (ubiquinone) activity /  quinone binding / ATP synthesis coupled electron transport / quinone binding / ATP synthesis coupled electron transport /  respirasome / respirasome /  細胞呼吸 / ATP metabolic process / reactive oxygen species metabolic process / respiratory electron transport chain / regulation of mitochondrial membrane potential / apoptotic signaling pathway / 細胞呼吸 / ATP metabolic process / reactive oxygen species metabolic process / respiratory electron transport chain / regulation of mitochondrial membrane potential / apoptotic signaling pathway /  ミトコンドリア / 2 iron, 2 sulfur cluster binding / negative regulation of cell growth / ミトコンドリア / 2 iron, 2 sulfur cluster binding / negative regulation of cell growth /  概日リズム / fatty acid biosynthetic process / NAD binding / FMN binding / 4 iron, 4 sulfur cluster binding / response to oxidative stress / 概日リズム / fatty acid biosynthetic process / NAD binding / FMN binding / 4 iron, 4 sulfur cluster binding / response to oxidative stress /  ミトコンドリア内膜 / membrane => GO:0016020 / ミトコンドリア内膜 / membrane => GO:0016020 /  oxidoreductase activity / oxidoreductase activity /  ミトコンドリアマトリックス / negative regulation of DNA-templated transcription / protein-containing complex binding / ミトコンドリアマトリックス / negative regulation of DNA-templated transcription / protein-containing complex binding /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Ovis aries (ヒツジ) Ovis aries (ヒツジ) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||
 データ登録者 データ登録者 | Fiedorczuk, K. / Letts, J.A. / Kaszuba, K. / Sazanov, L.A. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Atomic structure of the entire mammalian mitochondrial complex I. 著者: Karol Fiedorczuk / James A Letts / Gianluca Degliesposti / Karol Kaszuba / Mark Skehel / Leonid A Sazanov /   要旨: Mitochondrial complex I (also known as NADH:ubiquinone oxidoreductase) contributes to cellular energy production by transferring electrons from NADH to ubiquinone coupled to proton translocation ...Mitochondrial complex I (also known as NADH:ubiquinone oxidoreductase) contributes to cellular energy production by transferring electrons from NADH to ubiquinone coupled to proton translocation across the membrane. It is the largest protein assembly of the respiratory chain with a total mass of 970 kilodaltons. Here we present a nearly complete atomic structure of ovine (Ovis aries) mitochondrial complex I at 3.9 Å resolution, solved by cryo-electron microscopy with cross-linking and mass-spectrometry mapping experiments. All 14 conserved core subunits and 31 mitochondria-specific supernumerary subunits are resolved within the L-shaped molecule. The hydrophilic matrix arm comprises flavin mononucleotide and 8 iron-sulfur clusters involved in electron transfer, and the membrane arm contains 78 transmembrane helices, mostly contributed by antiporter-like subunits involved in proton translocation. Supernumerary subunits form an interlinked, stabilizing shell around the conserved core. Tightly bound lipids (including cardiolipins) further stabilize interactions between the hydrophobic subunits. Subunits with possible regulatory roles contain additional cofactors, NADPH and two phosphopantetheine molecules, which are shown to be involved in inter-subunit interactions. We observe two different conformations of the complex, which may be related to the conformationally driven coupling mechanism and to the active-deactive transition of the enzyme. Our structure provides insight into the mechanism, assembly, maturation and dysfunction of mitochondrial complex I, and allows detailed molecular analysis of disease-causing mutations. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5lnk.cif.gz 5lnk.cif.gz | 1.4 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5lnk.ent.gz pdb5lnk.ent.gz | 1.2 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5lnk.json.gz 5lnk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ln/5lnk https://data.pdbj.org/pub/pdb/validation_reports/ln/5lnk ftp://data.pdbj.org/pub/pdb/validation_reports/ln/5lnk ftp://data.pdbj.org/pub/pdb/validation_reports/ln/5lnk | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
+Mitochondrial complex I, ... , 42種, 42分子 1234569HNAMKLJabcdefghiklmnopq...
-タンパク質 , 2種, 3分子 jXv
| #24: タンパク質 |  分子量: 10119.541 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Ovis aries (ヒツジ) / 参照: UniProt: W5NQT7 Ovis aries (ヒツジ) / 参照: UniProt: W5NQT7#36: タンパク質 | | 分子量: 18818.070 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Ovis aries (ヒツジ) / 参照: UniProt: W5Q1B0 Ovis aries (ヒツジ) / 参照: UniProt: W5Q1B0 |
|---|
-非ポリマー , 10種, 25分子 








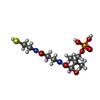









| #45: 化合物 | ChemComp-SF4 /  鉄・硫黄クラスター 鉄・硫黄クラスター#46: 化合物 | ChemComp-FMN / |  フラビンモノヌクレオチド フラビンモノヌクレオチド#47: 化合物 |  鉄・硫黄クラスター 鉄・硫黄クラスター#48: 化合物 | ChemComp-3PE /  ホスファチジルエタノールアミン ホスファチジルエタノールアミン#49: 化合物 | ChemComp-PC1 /  ホスファチジルコリン ホスファチジルコリン#50: 化合物 | ChemComp-CDL /  Cardiolipin Cardiolipin#51: 化合物 | ChemComp-ZN / | #52: 化合物 | ChemComp-NDP / |  ニコチンアミドアデニンジヌクレオチドリン酸 ニコチンアミドアデニンジヌクレオチドリン酸#53: 化合物 | ChemComp-ZMP / | #54: 化合物 | ChemComp-PNS / |  Phosphopantetheine Phosphopantetheine |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Mitochondrial complex I NADH:ユビキノン還元酵素 (水素イオン輸送型) NADH:ユビキノン還元酵素 (水素イオン輸送型)タイプ: COMPLEX / Entity ID: #1-#44 / 由来: NATURAL | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.97 MDa / 実験値: YES | ||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:   Ovis aries (ヒツジ) Ovis aries (ヒツジ) | ||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.4 | ||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||
| 試料 | 濃度: 5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R0.6/1 | ||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 90 % / 凍結前の試料温度: 277 K / 詳細: 34s blotting |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 100720 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 500 nm / Cs Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 100720 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 500 nm / Cs : 2.7 mm / アライメント法: COMA FREE : 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 1 sec. / 電子線照射量: 26 e/Å2 / 検出モード: INTEGRATING フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 撮影したグリッド数: 2 / 実像数: 2600 |
| 画像スキャン | サンプリングサイズ: 14 µm / 横: 4096 / 縦: 4096 / 動画フレーム数/画像: 7 / 利用したフレーム数/画像: 1-7 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.10_2148: / 分類: 精密化 | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 241000 | ||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 82000 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: REAL | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 4HEA 詳細: For the fourteen core subunits we used as a starting point our previous structure from T. thermophilus (PDB 4HEA) with sequence adjusted to ovine and side-chains rebuilt in SQWRL4. These are ...詳細: For the fourteen core subunits we used as a starting point our previous structure from T. thermophilus (PDB 4HEA) with sequence adjusted to ovine and side-chains rebuilt in SQWRL4. These are chains 1, 2, 3, 4, 5, 6, 9, H, A, J, K, N, M and L (with the same chain IDs as in 4HEA). For the remaining supernumerary subunits it was mostly de novo building with some help from homology modeling, which in most cases was not very useful due to low sequence homology. In few cases models produced by Phyre2 server were useful and were used as a starting point, due to the presence of templates with high coverage and confidence: 42kDa (chain k) - PDB ID 1P6X (xray) 39kDa (chain d) - PDB ID 3WJ7 (xray) SDAPs (chains j and X) - PDB ID 2DNW (NMR) B8 (chain e) - PDB ID 1S3A (NMR) | ||||||||||||||||||||||||||||||||
| 精密化 | 最高解像度: 3.9 Å | ||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj




























